# wir machen etwas, und speichern es in ein Objekt
habe.getan <- mache.etwas(Datensatz)
# dann machen wir etwas weiteres,
# und speichern wieder in ein Objekt
habe.weiteres.getan <- mache.weiteres(habe.getan)
# und kommen schließlich zum Endergebnis
endergebnis <- mache.noch.letzte.Sache(habe.weiteres.getan)24 Tidyverse
Das Tidyverse ist eine Weiterentwicklung von R, die maßgeblich von Hadley Wickham vorangetrieben wurde (weshalb auch die Bezeichung Hadleyverse noch gebräuchlich ist. Wickham ist mittlerweile Chefentwickler von RStudio). Das Standardwerk zum Tiddyverse “R for Data Science” (kurz R4DS) stammt ebenfalls von Wickham (2017) und ist online frei verfügbar unter https://r4ds.had.co.nz/ (eine physische Kopie des Buches kann ebenfalls gekauft werden).
Das Tidyverse folgt streng dem Konzept Tidy Data (Wickham, 2014), welches besagt, dass ein Datensatz so aufgebaut sein muss, dass jeweils ein Fall pro Zeile abgebildet wird. Das bedeutet, dass jede Beobachtung (auch Wiederholungen) in einer eigenen Zeile steht, und die jeweiligen Variablen durch die Spalten repräsentiert werden.
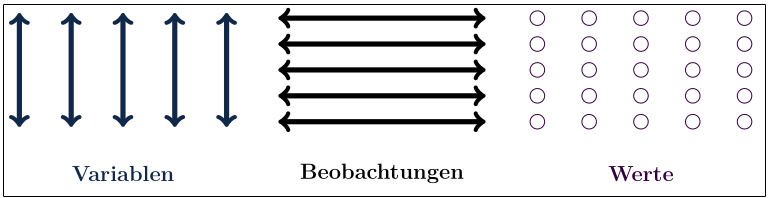
Abbildung 24.1 zeigt die Sicht auf ein Datenobjekt nach dem Tidy Data Konzept. Jede Variable wird als eigene Spalte dargestellt. Jede Reihe entspricht einer Beobachtung. Somit entsprechen die einzelnen Werte in der Tabelle genau einer Variable zu einer Beobachtung.
Man spricht in diesem Zusammenhang von “long table” und “wide table”.
Die Matrix der Pflegeberufe, die wir aus Abbildung 8.1 übernommen haben, stellt dabei die wide table, die breite Tabelle dar.
1999 2001 2003 2005 2007 2009 2011 2013 2015
Krankenpflegeassistenz 16624 19061 19478 21537 27731 36481 46517 54371 64127
Altenpflegehilfe 55770 52710 49727 45776 48326 47903 47978 48363 49507
Kinderkrankenpflege 47779 48203 48822 48519 49080 49307 48291 48937 48913
Krankenpflege 430983 436767 444783 449355 457322 465446 468192 472580 476416
Altenpflege 109161 124879 141965 158817 178902 194195 208304 227154 246412“Breit” bedeutet, dass die Tabelle, wenn wir ihr nun 10 weitere Jahrgänge hinzufügen würden, immer breiter und breiter werden würde. Die wide table ist als Darstellung der Daten sicherlich gut geeignet, und die Leser erhalten eine gute Übersicht. Für die Datenanalyse und -verarbeitung ist dieses Format eher ungeeignet, da mit jedem Wert in der Tabelle eigentlich zwei Variablen andressiert werden (konkrete Berufsgruppe in einem konkreten Jahr). Welche beiden Variablen das nun genau sind, lässt sich am Wert alleine nicht ablesen, und wir hätten große Probleme, wenn Zeilen und Spalten nicht benannt wären!
Als Faustregel kann man sich merken, dass eine wide table dann vorliegt, wenn auch (bedeutsame) Zeilennamen im Datensatz vergeben wurden (was bei einer matrix häufig der Fall ist).
Ein Datensatz nach dem Tidy Data-Konzept ist vom Typ long table.
Für die Matrix der Pflegeberufe sähe die long table-Version das Datensatzes so aus:
Jahr Berufsgruppe Anzahl
1 1999 Krankenpflegeassistenz 16624
2 2001 Krankenpflegeassistenz 19061
3 2003 Krankenpflegeassistenz 19478
4 2005 Krankenpflegeassistenz 21537
5 2007 Krankenpflegeassistenz 27731
6 2009 Krankenpflegeassistenz 36481
7 2011 Krankenpflegeassistenz 46517
8 2013 Krankenpflegeassistenz 54371
9 2015 Krankenpflegeassistenz 64127
10 1999 Altenpflegehilfe 55770
11 2001 Altenpflegehilfe 52710
12 2003 Altenpflegehilfe 49727
( . . . )Jede Spalte repräsentiert eine Variable, und jede Zeile repräsentiert eine Beobachtung. Auch ohne die Spaltennamen (Zeilennamen gibt es gar keine) könnten wir erkennen, worum es in dem Datensatz geht.
Der Name long table leitet sich davon ab, dass die Tabelle, wenn wir ihr mehr Daten hinzufügen würden, immer länger und länger werden würde.
Die Tatsache, dass die Verarbeitung solcher long tables wesentlich einfacher ist, bildet den Grundgedanken des Tidyverse, und es wurden spezielle R-Erweitrungen implementiert, die speziell an Tidy Data angepasst sind. Die Erweiterungen wurden nach dieser gemeinsame Philosophie entworfen, sie verwenden die selbe “Grammatik” und greifen auf die selbe Datenstruktur (tibbles, dazu später mehr) zurück.
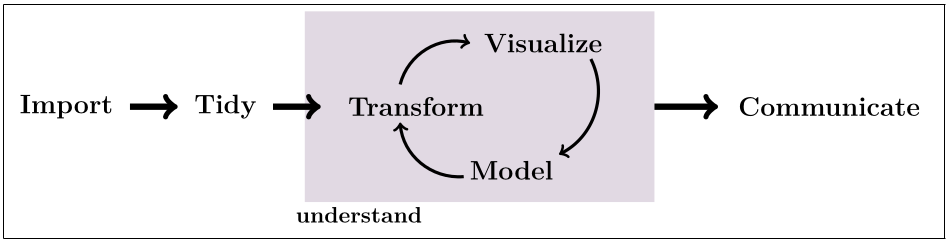
Abbildung 24.2 zeigt den typischen Ablauf einer Datenauswertung in 4 Schritten.
Die Daten müssen importiert und
gegebenenfalls angepasst (ins
tidy databzw.long tableFormat überführt werden).Anschließend werden die Daten “statistisch ausgewertet”, d.h. wir versuchen zu verstehen, was wir in den Daten sehen und welche spezifischen Informationen extrahiert werden können. Das heisst die Daten werden transformiert (z.B. indem man Untergruppen bildet) und visualisiert (z.B. in Form von Diagrammen und Tabellen) oder wir rechnen Modelle wie Korrelationen, Regressionen und Signifikanztests.
Unsere Erkenntnisse nutzen wir dann zur Kommunikation (Präsentation) unserer Forschungsergebnisse.
Für all diese Arbeitsschritte stellt das Tidyverse passende Pakete und Funktionen in R zur Verfügung, und wir werden die Schritte nun einzeln durchlaufen.
24.1 Pakete
In R besteht das Tidyverse im Wesentlichen aus dem Zusatzpaket tidyverse, welches alle notwendingen Unterpakete als Abhängigkeit mit-installiert:
Tibble, die Datenklasse im Tidyverse, im Paket
tibble, mit den Funktionen
tibble()as_tibble()
Daten importieren:
readr, mit den Funktionen
read_csv(),read_tsv(),read_delim(),read_table()
Daten tidy machen:
tidyr, mit den Funktionen
pivot_longer(),pivot_wider(),separate(),unite(),drop_na(),replace_na(),fill(),complete(),expand()forcats, mit den Funktionen
as_factor()
Umgang mit Datensätzen:
dplyr, mit den Funktionen
arrange(),filter(),select(),pull(),mutate(),transmute(),rename(),summarise(),bind_cols(),bind_rows()purrr, mit den Funktionen
modify(),map(),reduce(),nest()stringr, mit den Funktionen
str_sub(),bind_locate(),bind_extract(),bind_count(),bind_match()
Daten visualisieren:
ggplot, mit den Funktionen
ggplot(),qplot()
Dazu später mehr, denn um die Funktionen vorzustellen, müssen wir uns zunächst der Pipe zuwenden.
24.2 Pipe
Ein wichtiger Operator für die Arbeit im Tidyverse ist die Pipe aus dem magrittr-Zusatzpaket (die Pipe wird mit tidyverse aktiviert, Sie müssen magrittr nicht extra installieren). Sie erlaubt es, Datenströme weiterzuleiten. Ihre Funktionalität hat so sehr überzeugt, dass sie im Standard-R ab Version 4.1 enthalten ist, siehe Kapitel 11.
Hierfür wurde ein eigener Operator implementiert, die Zeichenkette %>% (im Standard-R lautet die Zeichenkette |>).
Sie bedeutet so viel wie “und dann”.
Zur Eklärung sei nochmals der “klassische Weg” der Arbeitsschritte in R aufgezeigt:
Mit Einsatz der Pipe wird dieser Prozess quasi umgekehrt in
# Speichere Endergebnis
endergebnis <- Datensatz %>%
gruppiere.nach.geschlecht %>%
sortiere Alter aufsteigend %>%
nimm die letzten 4 WerteDie liest sich in etwa so:
“Speichere etwas ins Objekt
endergebnis, und das geht so…nimm den
Datensatzund danngruppiere die Daten nach
geschlechtund dannsortiere nach
Alteraufsteigend und dannnimm die letzten 4 Werte. ”
Die Pipe reicht das jeweilige Ergebnis (den Datenstrom) an die nächste Code-Zeile weiter. Erst wenn die letzte Zeile durchgelaufen ist, wird das Resultat in endergebnis gespeichert.
Dies liest sich zu Beginn evtl. etwas ungewohnt, aber Sie können erkennen, dass die Befehle so wesentlich übersichtlicher und die einzelnen Manipulationsschritte nachvollziehbarer geworden sind. Auch kann man sich diese Art der “Grammatik” relativ leicht merken.
Benötigt eine Funktion die Angabe eines Datensets, so kann mit einem Punkt . auf den Pipe-Datenstrom verwiesen werden.
# Der Punkt übergibt den Pipe-Strom an die Funktion sum()
endergebnis <- Datensatz %>%
wähle Spalte "Alter" %>%
# berechne Summe
sum(.)Die Pipe funktioniert bei jedem R-Objekt, egal in welchem Format (long table vs. wide table, Faktor, Vektor, Matrix, usw.) es vorliegt.
24.2.1 Unterschiede zwischen %>% und |>
Während sich |> und %>% in einfachen Fällen identisch verhalten, gibt es ein paar wichtige Unterschiede. Diese betreffen Sie aber nur, wenn Sie %>% schon lange nutzen und einige der fortgeschrittenen Funktionen verwendet haben.
Standardmäßig übergibt die Pipe das Objekt auf ihrer linken Seite an das erste Argument der Funktion auf der rechten Seite. Mit
%>%können Sie die Platzierung des Datenstroms mit dem Platzhalter “.” ändern. Zum Beispiel istx %>% f(1)äquivalent zuf(x, 1), aberx %>% f(1, .)ist äquivalent zuf(1, x). Seit R 4.2.0 ist ein ähnlicher Platzhalte über_zur Basis-Pipe hinzugefügt worden, jedoch muss hier immer ein Argument benannt werden. Zum Beispiel istx |> f(1, y = _)äquivalent zuf(1, y = x).Mit
%>%kann man die Klammern weglassen, wenn man eine Funktion ohne weitere Argumente aufruft;|>erfordert immer die Klammern.Der Platzhalter
|>ist absichtlich einfach gehalten und kann viele Eigenschaften des Platzhalters%>%nicht wiedergeben. Sie können ihn nicht an mehrere Argumente übergeben, und er hat kein besonderes Verhalten, wenn er innerhalb einer anderen Funktion verwendet wird. Zum Beispiel istdf %>% split(.$var)äquivalent zusplit(df, df$var)unddf %>% {plot(.$x, .$y)}ist äquivalent zuplot(df$x, df$y).Mit
%>%können Sie den Punkt.auf der linken Seite von Operatoren verwenden, so dass Sie eine einzelne Spalte aus einem Datenframe mit (z. B.)mtcars %>% .$cylextrahieren können. Dies geht mit|>nicht.
Glücklicherweise gibt es keinen Grund, sich ganz auf die eine oder andere Pipe festzulegen - Sie können die Basis-Pipe für die meisten Fälle verwenden, und die Magrittr-Pipe benutzen, wenn Sie deren speziellen Eigenschaften wirklich benötigen.
24.3 tibbles
Da dieses Konzept der long table so essenziell ist, wurde im Tidyverse eine eigene Datenklasse eingeführt, die tibble heisst (das kommt daher, dass tidy data immer Tabellen sind, was häufig per tbl abgekürzt wird. Liest man im Englischn tbl laut und schnell, klingt das wie - genau - tibble).
Sie entspricht in etwa dem Datenframe, hat aber intern noch weitere wichtige Änderungen eingebaut.
Tibbles werden so wie Datenframes erzeugt, indem man gleichlange Vektoren übergibt. Der Funktionsname ist tibble().
# aktiviere Tidyverse
library(tidyverse)
# erzeuge ein tibble
tibble(a=c(1,2,3), b=c("a", "b", "c"), c=c(T, F, T))## # A tibble: 3 × 3
## a b c
## <dbl> <chr> <lgl>
## 1 1 a TRUE
## 2 2 b FALSE
## 3 3 c TRUEDie Schwesterfunktion tribble() (mit r) erlaubt diese Schreibweise
# erzeuge ein tibble mit tRibble
tribble(
~Text, ~Zahl, ~Logic,
"a", 1, T,
"b", 2, F,
"c", 3, T
)## # A tibble: 3 × 3
## Text Zahl Logic
## <chr> <dbl> <lgl>
## 1 a 1 TRUE
## 2 b 2 FALSE
## 3 c 3 TRUESind die übergebenen Reihen nicht gleich lang, werden sie als list gespeichert.
# Datenreihen NICHT gleich lang
tribble(
~Text, ~Zahl, ~Logic,
"a", 1:3, T,
"b", 4:6, F,
"c", 7:9, T
)## # A tibble: 3 × 3
## Text Zahl Logic
## <chr> <list> <lgl>
## 1 a <int [3]> TRUE
## 2 b <int [3]> FALSE
## 3 c <int [3]> TRUEDie meisten Funktionen des Tiddyverse funktioneren auch mit Datenframes, eben weil sie sich so ähnlich sind. Dennoch ergibt es (schon ideologisch) Sinn, die Datenframes in tibbles umzuwandeln.
Schauen wir uns hierfür die Daten der Pflegeberufe als long table an:
# Pflegeframe
head(Pflegeframe)## Jahr Berufsgruppe Anzahl
## 1 1999 Krankenpflegeassistenz 16624
## 2 2001 Krankenpflegeassistenz 19061
## 3 2003 Krankenpflegeassistenz 19478
## 4 2005 Krankenpflegeassistenz 21537
## 5 2007 Krankenpflegeassistenz 27731
## 6 2009 Krankenpflegeassistenz 36481# Übersicht von "Pflegeframe"
str(Pflegeframe)## 'data.frame': 45 obs. of 3 variables:
## $ Jahr : Factor w/ 9 levels "1999","2001",..: 1 2 3 4 5 6 7 8 9 1 ...
## $ Berufsgruppe: Factor w/ 5 levels "Krankenpflegeassistenz",..: 1 1 1 1 1 1 1 1 1 2 ...
## $ Anzahl : num 16624 19061 19478 21537 27731 ...Wir erhalten eine Zusammenstellung von wichtigen Informationen. Unser Pflegetibble hat 45 Beobachtungen (Reihen) von 3 Variablen (Spalten). Die Variablen Jahr und Berufsgruppe liegen als Faktoren vor, Anzahl ist numerisch.
# Datenklasse von "Pflegeframe"
class(Pflegeframe)## [1] "data.frame"Jetzt wandeln wir das Pflegeframe in ein Pflegetibble um.
# erzeuge Pflegetibble aus Pflegeframe
Pflegetibble <- tibble(Pflegeframe)
# Datenklasse von "Pflegeframe"
class(Pflegetibble)## [1] "tbl_df" "tbl" "data.frame"Wie Sie sehen, ist unser Tibble immernoch ein Datenframe. Es ist aber auch ein “Tibble Datenframe” (tbl_df) und einfach ein Tibble (tbl).
Die Funktion zum Einsehen von Tibbles heisst glimpse() (englisch für Blick).
# werfe einen Blick auf "Pflegetibble"
glimpse(Pflegetibble)## Rows: 45
## Columns: 3
## $ Jahr <fct> 1999, 2001, 2003, 2005, 2007, 2009, 2011, 2013, 2015, 199…
## $ Berufsgruppe <fct> Krankenpflegeassistenz, Krankenpflegeassistenz, Krankenpf…
## $ Anzahl <dbl> 16624, 19061, 19478, 21537, 27731, 36481, 46517, 54371, 6…Die Ausgabe sieht fast genau so aus wie der str()-Aufruf. Neben den Variablennamen steht der Datentyp, wobei <fct> für Faktor, <dbl> für Dezimalzahlen und <lgl> für logical steht.
Im klassischen R hat man ein data.frame mit str() angeschaut, im Tidyverse schaut man mit glimpse() auf ein tibble.
24.4 Funktionsaufrufe
In R gibt es jede Menge Zusatzpakete, und obschon die Programmierer sich Mühe geben, einen Funktionsnamen nicht doppelt zu vergeben (vielleicht ist Ihnen schon aufgefallen, dass im Tidyverse viele Funktionen mit einem Unterstrich _ geschrieben werden, z.B. read_sav() oder as_factor()), kann dies durchaus vorkommen.
Wenn wir das Tidyverse per library() aktivieren, erhalten wir folgende Informationen:
library(tidyverse)## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## ✖ dplyr::filter() masks stats::filter()
## ✖ dplyr::lag() masks stats::lag()
## ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errors── Attaching packages ─────────────────── tidyverse 1.3.0 ──
✓ ggplot2 3.3.3 ✓ purrr 0.3.4
✓ tibble 3.1.0 ✓ dplyr 1.0.5
✓ tidyr 1.1.3 ✓ stringr 1.4.0
✓ readr 1.4.0 ✓ forcats 0.5.1
── Conflicts ──────────────────────────── tidyverse_conflicts() ──
x dplyr::filter() masks stats::filter()
x dplyr::lag() masks stats::lag()Wie Sie sehen, gibt es nun Konflikte, da die Funktionen filter() und lag() jeweils in den Paketen tidyverse und stats (R-Basispaket) existieren. Das heisst, möchte man die Funktion filter() verwenden, muss man R mitteilen, aus welchem Paket sie stammt. Dies erfolgt in R indem man den Paketnamen getrenn durch zwei Doppelpunkte vor die Funktion schreibt.
# nutze Funktion filter() aus dem Paket "dplyr"
dplyr::filter()
# nutze Funktion filter() aus dem "stats" Paket
stats::filter()Im Tidyverse ist es daher durchaus üblich, den Paketnamen mit zwei Doppelpunkten getrennt vor den Funktionsnamen zu setzen. So ist immer klar, welche Funktion aus welchem Paket denn nun aufgerufen werden soll.
# Paketname::Funktionsname
haven::read_sav("spss.sav")Durch die Refernzierung des Paketnamens sparen Sie sich das Einbinden des gesamten Paketes per library().
Dies funktioniert auch bei den zahlreichen Datensätzen, die R mitliefert. Möchten Sie den Datensatz flights aus dem Paket nycflights13 nutzen, können Sie (nachdem das Paket nycflights13 installiert wurde) wie folgt referenzieren:
# Installiere Paket "nycflights13"
install.packages("nycflights13")
# Paketname::Datensatz
head(nycflights13::flights)