# installiere data.table
install.packages("data.table", dependencies=TRUE)29 data.table
Neben dem zuvor besprochenen tidyverse steht mit data.table ein weiterer R-Dialekt zur Verfügung, der sich immer größerer Beliebtheit erfreut. Im Kern sind data.tables verbesserte Versionen von data.frames, die schneller und speichereffizienter arbeiten und mit einer prägnanteren Syntax manipuliert werden können. Das Paket stellt außerdem eine Reihe zusätzlicher Funktionen zum Lesen und Schreiben von tabellarischen Dateien, zum Umformen von Daten zwischen langen und breiten Formaten und zum Verbinden von Datensätzen zur Verfügung.
29.1 Installation
Alle Funktionen sind über das Paket data.table implementiert, welches wie gewohnt installiert und aktiviert werden kann.
# data.table aktivieren
library(data.table)29.2 Modify-in-Place
Der größte Unterschied besteht darin, dass data.table die Modify-in-Place-Methode verwendet. Das klassische R und auch das Tidyverse verwenden die Copy-on-Modify-Methode, welche besagt, dass bei der Manipulation eines Objektes das Ergebnis in einem neuen Objekt gespeichert wird.
# klassisches "Copy-on-Modify"
meine.daten %>%
mutate(Neu = Alt*10)Bei oben stehendem Code wird das Objekt meine.daten nicht verändert. Das Ergebnis der mutate()-Funktion wird als neues Objekt ausgegeben. Dieses neue Objekt ist eine Kopie der Ursprungsdaten meine.daten, an welcher die Veränderungen vorgenommen werden.
Mit data.table wird der Ansatz Modify-in-Place verfolgt.
# Modify-in-Place
meine.daten[, Neu := Alt*10]Der oben stehende Code erzeugt keine Kopie von meine.daten. Vielmehr wird das Objekt meine.daten direkt verändert. Im klassischen R entspricht diese Vorgehensweise dem Code
meine.daten$Neu <- meine.daten$Alt*10Durch Modify-in-Place wird data.table sehr effizient, wenn größere Datenmengen verarbeitet werden sollen. Es kann jedoch auch dazu führen, dass der Code schwieriger zu verstehen ist und überraschende Ergebnisse liefert (insbesondere, wenn ein data.table innerhalb einer Funktion modifiziert wird).
29.3 Grundlegende Syntax
Die generelle Syntax von data.table lautet
dt[i, j, by]
wobei
dteindata.table-Objekt ist.izum Filtern und für join-Funktionen genutzt wird.jzum Manipulieren, Transformieren und Zusammenfassen der Daten verwendet wird.byzum Gruppieren genutzt wird.
Man kann die Syntax lesen als:
„In diesen Zeilen, mache dies, gruppiert nach jenem“.
29.4 Daten einlesen
Der erste Schritt der meisten Datenanalysen besteht darin, Daten in den Speicher zu laden. Wir können die Funktion data.table::fread() verwenden (das f steht für fast (schnell)), um reguläre, durch Trennzeichen getrennte Dateien wie txt- oder csv-Dateien zu lesen. Diese Funktion ist nicht nur schnell, sondern erkennt automatisch das Trennzeichen und errät die Klasse jeder Spalte sowie die Anzahl der Zeilen in der Datei.
# Daten einlesen mit fread()
dt <- fread("data/Befragung22.csv")
# anschauen
str(dt)Classes 'data.table' and 'data.frame': 37 obs. of 6 variables:
$ alter : int 20 28 41 34 26 38 28 21 27 26 ...
$ geschlecht: chr "weiblich" "weiblich" "männlich" "weiblich" ...
$ stifte : int 12 7 1 13 18 25 29 1 2 5 ...
$ geburtsort: chr "Düren" "Neuss" "Bonn" "Düsseldorf" ...
$ fahrzeit : int 1 45 60 25 15 50 40 60 60 40 ...
$ podcast : chr "selten" "selten" "selten" "oft" ...
- attr(*, ".internal.selfref")=<externalptr> Das Objekt dt gehört sowohl zur Klasse data.frame als auch zu der neuen Klasse data.table.
Die Daten können auch direkt über eine URL eingelesen werden.
# lade per URL
dt <- fread("https://www.produnis.de/R/data/Befragung22.csv")Liegen die Daten bereits als data.frame vor, können sie per as.data.table() umgewandelt werden.
# lade klassisches Datenframe
df <- read.table("https://www.produnis.de/R/data/Datentabelle.txt",
header=TRUE)
# wandle in data.table um
dt2 <- as.data.table(df)Classes 'data.table' and 'data.frame': 10 obs. of 4 variables:
$ Geschlecht: chr "m" "w" "w" "m" ...
$ Alter : int 28 18 25 29 21 19 27 26 31 22
$ Gewicht : int 80 55 74 101 84 74 65 56 88 78
$ Groesse : int 170 174 183 190 185 178 169 163 189 184
- attr(*, ".internal.selfref")=<externalptr> Sollen die Daten von Hand eingegeben werden, wird die Funktion data.table() verwendet.
# erzeuge von Hand
dt3 <- data.table(x = 1:10,
y = 11:20,
z = factor(rep(c("foo", "bar"), 5))
)
# anschauen
str(dt3)Classes 'data.table' and 'data.frame': 10 obs. of 3 variables:
$ x: int 1 2 3 4 5 6 7 8 9 10
$ y: int 11 12 13 14 15 16 17 18 19 20
$ z: Factor w/ 2 levels "bar","foo": 2 1 2 1 2 1 2 1 2 1
- attr(*, ".internal.selfref")=<externalptr> 29.5 Daten speichern
Mit der Funktion fwrite() können data.tables (aber auch data.frames) in eine Datei gespeichert werden. Sie funktioniert ähnlich wie write.csv, ist aber wesentlich schneller. Wird kein Dateiname angegeben, erfolgt die Ausgabe in der Konsole. So kann überprüft werden, was in die Datei geschrieben würde.
# schreibe Objekt "dt2" in die Konsole
fwrite(dt2)Geschlecht,Alter,Gewicht,Groesse
m,28,80,170
w,18,55,174
w,25,74,183
m,29,101,190
m,21,84,185
w,19,74,178
w,27,65,169
w,26,56,163
m,31,88,189
m,22,78,184# schreibe Objekt "dt" in datei "dt.csv"
fwrite(dt2, "dt2.csv")
# schreibe Objekt "dt" in datei "dt.txt"
fwrite(dt2, "dt2.txt")29.6 Fälle filtern mit i
Wir erinnern uns, dass die allgemeine Syntax dt[i, j, by] lautet. Über den Parameter i können die Daten gefilter werden, so dass nur bestimmte Fälle berücksichtigt werden. Beispielsweise könnten wir im Objekt dt nur solche Fälle auswählen, bei denen das Alter größer als 30 ist.
dt[alter > 30] alter geschlecht stifte geburtsort fahrzeit podcast
<int> <char> <int> <char> <int> <char>
1: 41 männlich 1 Bonn 60 selten
2: 34 weiblich 13 Düsseldorf 25 oft
3: 38 weiblich 25 Dinslaken 50 oft
4: 38 männlich 5 Donezk 57 manchmal
5: 31 weiblich 16 Charkov Ukraine 135 oft
6: 36 weiblich 1 Rybnik 90 manchmal
7: 45 männlich 1 Gelsenkirchen 85 oftDies ist Vergleichbar mit dem klassischen R-Aufruf
# klassischer R-Befehl
dt[dt$alter > 30] alter geschlecht stifte geburtsort fahrzeit podcast
<int> <char> <int> <char> <int> <char>
1: 41 männlich 1 Bonn 60 selten
2: 34 weiblich 13 Düsseldorf 25 oft
3: 38 weiblich 25 Dinslaken 50 oft
4: 38 männlich 5 Donezk 57 manchmal
5: 31 weiblich 16 Charkov Ukraine 135 oft
6: 36 weiblich 1 Rybnik 90 manchmal
7: 45 männlich 1 Gelsenkirchen 85 oftDa alle Ausdrücke in i im Kontext der data.table ausgewertet werden, müssen wir den (eventuell sehr langen) Namen des Objektes nicht erneut eingeben. Dies ist vor allem bei längeren Ausdrücken sehr bequem.
# erzeuge langen Objektnamen
langer.Objekt.name <- dtDer klassische R-Aufruf
# klassischer R-Befehl
langer.Objekt.name[langer.Objekt.name$alter > 25 &
langer.Objekt.name$geschlecht=="männlich" |
langer.Objekt.name$stifte > 30] alter geschlecht stifte geburtsort fahrzeit podcast
<int> <char> <int> <char> <int> <char>
1: 41 männlich 1 Bonn 60 selten
2: 26 männlich 5 Düsseldorf 40 nie
3: 38 männlich 5 Donezk 57 manchmal
4: 20 weiblich 32 Wesel 89 nie
5: 45 männlich 1 Gelsenkirchen 85 oftverkürzt sich auf
langer.Objekt.name[alter > 25 & geschlecht=="männlich" | stifte > 30] alter geschlecht stifte geburtsort fahrzeit podcast
<int> <char> <int> <char> <int> <char>
1: 41 männlich 1 Bonn 60 selten
2: 26 männlich 5 Düsseldorf 40 nie
3: 38 männlich 5 Donezk 57 manchmal
4: 20 weiblich 32 Wesel 89 nie
5: 45 männlich 1 Gelsenkirchen 85 oft29.7 Fälle sortieren mit i
Dem Parameter i können auch Funktionen übergeben werden. So lassen sich die Daten beispielsweise über die order()-Funktion sortieren.
# nehme anderen (kürzeren) Datensatz zur Demonstration
dt2[order(Alter)] Geschlecht Alter Gewicht Groesse
<char> <int> <int> <int>
1: w 18 55 174
2: w 19 74 178
3: m 21 84 185
4: m 22 78 184
5: w 25 74 183
6: w 26 56 163
7: w 27 65 169
8: m 28 80 170
9: m 29 101 190
10: m 31 88 189# absteigend
dt2[order(Gewicht, decreasing = TRUE)] Geschlecht Alter Gewicht Groesse
<char> <int> <int> <int>
1: m 29 101 190
2: m 31 88 189
3: m 21 84 185
4: m 28 80 170
5: m 22 78 184
6: w 25 74 183
7: w 19 74 178
8: w 27 65 169
9: w 26 56 163
10: w 18 55 17429.8 Daten verarbeiten mit j
Nachdem der Datensatz mittels i eventuell vorsortiert und -gefiltert wurde, erfolgen die eigentlichen Operationen über den Parameter j. So können wir den Mittelwert des Alters der Probanden wie folgt bestimmen:
# Mittelwert des Alters
dt[, mean(alter)][1] 25.2973# Mittelwert des Alters der Männer
dt[geschlecht == "männlich", mean(alter)][1] 29Innerhalb von j kann jede Funktion verwendet werden. So könnten wir überprüfen, ob die Variablen fahrzeit und alter miteinander korrelieren (ja, das ist quatsch).
# korrelieren alter und fahrzeit?
dt[, cor(alter, fahrzeit)][1] 0.1504465Es können auch mehrere Funktionen angewendet werden. Hierfür müssen diese per list() an den Parameter j übergeben werden. Auf diese Weise könnten wir Median, Mittelwert und Standardabweichung des Alters der Probanden bestimmen.
# mehrere Funktionen per list()
dt[, list(Median = median(alter),
Mittelw = mean(alter),
Stdabw = sd(alter))] Median Mittelw Stdabw
<int> <num> <num>
1: 22 25.2973 6.765373Da der Parameter j immer eine Liste erwartet, kann die Funktion list() mit einem Punkt abgekürzt werden.
# geht auch mit "."
dt[, .(Median = median(alter),
Mittelw = mean(alter),
Stdabw = sd(alter),
InterquA = IQR(alter))] Median Mittelw Stdabw InterquA
<int> <num> <num> <num>
1: 22 25.2973 6.765373 629.9 Daten bearbeiten mit j
Über den Parameter j können die Daten auch manipuliert werden, ähnlich wie bei der mutate()-Funktion des Tidyverse. Eine neue Variable kann über die Zeichenkette := definiert werden (dem so genannten Walrus Operator (Walross-Operator), der so heisst, weil die Zeichenfolge := an die Stoßzähne eines Walrosses erinnert. Das Logo des data.table-Pakets zeigt eine Robbe, was zur humorvollen Verbindung beigetragen hat).
Mit folgendem Aufruf erzeugen wir eine neue Variable FahrzeitH, welche die fahrzeit in Stunden beinhalten soll.
# FahrzeitH in Stunden
dt[, FahrzeitH := fahrzeit/60]
# anzeigen
str(dt)Classes 'data.table' and 'data.frame': 37 obs. of 7 variables:
$ alter : int 20 28 41 34 26 38 28 21 27 26 ...
$ geschlecht: chr "weiblich" "weiblich" "männlich" "weiblich" ...
$ stifte : int 12 7 1 13 18 25 29 1 2 5 ...
$ geburtsort: chr "Düren" "Neuss" "Bonn" "Düsseldorf" ...
$ fahrzeit : int 1 45 60 25 15 50 40 60 60 40 ...
$ podcast : chr "selten" "selten" "selten" "oft" ...
$ FahrzeitH : num 0.0167 0.75 1 0.4167 0.25 ...
- attr(*, ".internal.selfref")=<externalptr>
- attr(*, "index")= int(0)
..- attr(*, "__geschlecht")= int [1:37] 3 8 10 11 13 31 33 34 37 1 ...So können wir auch mittels der cut()-Funktion die Daten klassieren, zum Beispiel das Alter:
dt[, alterK := cut(alter, breaks=c(0,20,25,30,40,50),
ordered=TRUE)]
# anzeigen
str(dt)Classes 'data.table' and 'data.frame': 37 obs. of 8 variables:
$ alter : int 20 28 41 34 26 38 28 21 27 26 ...
$ geschlecht: chr "weiblich" "weiblich" "männlich" "weiblich" ...
$ stifte : int 12 7 1 13 18 25 29 1 2 5 ...
$ geburtsort: chr "Düren" "Neuss" "Bonn" "Düsseldorf" ...
$ fahrzeit : int 1 45 60 25 15 50 40 60 60 40 ...
$ podcast : chr "selten" "selten" "selten" "oft" ...
$ FahrzeitH : num 0.0167 0.75 1 0.4167 0.25 ...
$ alterK : Ord.factor w/ 5 levels "(0,20]"<"(20,25]"<..: 1 3 5 4 3 4 3 2 3 3 ...
- attr(*, ".internal.selfref")=<externalptr>
- attr(*, "index")= int(0)
..- attr(*, "__geschlecht")= int [1:37] 3 8 10 11 13 31 33 34 37 1 ...Pro Aufruf kann der Walross-Operator nur einmal verwendet werden. Sollen mehrere Variablen verändert oder hinzugefügt werden, steht die let()-Funktion bereit. Innerhalb von let() werden wie gewohnt einfache Gleichheitszeichen verwendet.
# mehrere Manipulationen per let()
dt[, let(geschlecht = factor(geschlecht),
geburtsort = factor(geburtsort),
podcast = factor(podcast, ordered=TRUE,
levels=c("nie", "selten", "manchmal",
"oft", "immer")))]
# anzeigen
str(dt)Classes 'data.table' and 'data.frame': 37 obs. of 8 variables:
$ alter : int 20 28 41 34 26 38 28 21 27 26 ...
$ geschlecht: Factor w/ 2 levels "männlich","weiblich": 2 2 1 2 2 2 2 1 2 1 ...
$ stifte : int 12 7 1 13 18 25 29 1 2 5 ...
$ geburtsort: Factor w/ 26 levels "Bagdad","Bonn",..: 9 21 2 10 8 5 21 7 18 10 ...
$ fahrzeit : int 1 45 60 25 15 50 40 60 60 40 ...
$ podcast : Ord.factor w/ 5 levels "nie"<"selten"<..: 2 2 2 4 NA 4 4 3 1 1 ...
$ FahrzeitH : num 0.0167 0.75 1 0.4167 0.25 ...
$ alterK : Ord.factor w/ 5 levels "(0,20]"<"(20,25]"<..: 1 3 5 4 3 4 3 2 3 3 ...
- attr(*, ".internal.selfref")=<externalptr>
- attr(*, "index")= int(0) Die Änderungen wurden direkt im Objekt dt gespeichert.
29.10 data.table kopieren
Eine weitere wesentliche Eigenschaft von data.table-Objekten besteht darin, dass man sie gesondert kopieren muss. Wir eine data.table auf klassischem Wege in ein neues Objekt “kopiert”, so erfolgt keine echte Kopie, sondern lediglich ein symbolischer Link auf das ursprüngliche Objekt.
# weise dt einem neuen Objekt zu
neu <- dt
str(neu)Classes 'data.table' and 'data.frame': 37 obs. of 8 variables:
$ alter : int 20 28 41 34 26 38 28 21 27 26 ...
$ geschlecht: Factor w/ 2 levels "männlich","weiblich": 2 2 1 2 2 2 2 1 2 1 ...
$ stifte : int 12 7 1 13 18 25 29 1 2 5 ...
$ geburtsort: Factor w/ 26 levels "Bagdad","Bonn",..: 9 21 2 10 8 5 21 7 18 10 ...
$ fahrzeit : int 1 45 60 25 15 50 40 60 60 40 ...
$ podcast : Ord.factor w/ 5 levels "nie"<"selten"<..: 2 2 2 4 NA 4 4 3 1 1 ...
$ FahrzeitH : num 0.0167 0.75 1 0.4167 0.25 ...
$ alterK : Ord.factor w/ 5 levels "(0,20]"<"(20,25]"<..: 1 3 5 4 3 4 3 2 3 3 ...
- attr(*, ".internal.selfref")=<externalptr>
- attr(*, "index")= int(0) Wir haben das Objekt dt nur scheinbar in das neue Objekt neu kopiert. Wenn wir Änderungen am Objekt neu vornehmen, so sind diese auch im Objekt dt präsent, weil eben nicht kopiert, sondern nur ein Verweis erstellt wurde.
# erstelle neue Variable in "neu"
neu[, kuckuck := fahrzeit * stifte]
# die neue Variable ist auch in "dt" enthalten
str(dt)Classes 'data.table' and 'data.frame': 37 obs. of 9 variables:
$ alter : int 20 28 41 34 26 38 28 21 27 26 ...
$ geschlecht: Factor w/ 2 levels "männlich","weiblich": 2 2 1 2 2 2 2 1 2 1 ...
$ stifte : int 12 7 1 13 18 25 29 1 2 5 ...
$ geburtsort: Factor w/ 26 levels "Bagdad","Bonn",..: 9 21 2 10 8 5 21 7 18 10 ...
$ fahrzeit : int 1 45 60 25 15 50 40 60 60 40 ...
$ podcast : Ord.factor w/ 5 levels "nie"<"selten"<..: 2 2 2 4 NA 4 4 3 1 1 ...
$ FahrzeitH : num 0.0167 0.75 1 0.4167 0.25 ...
$ alterK : Ord.factor w/ 5 levels "(0,20]"<"(20,25]"<..: 1 3 5 4 3 4 3 2 3 3 ...
$ kuckuck : int 12 315 60 325 270 1250 1160 60 120 200 ...
- attr(*, ".internal.selfref")=<externalptr>
- attr(*, "index")= int(0) Dies ist ein häufiger fataler Anfängerfehler, der zum Datenverlust führen kann!
Um das Objekt tatsächlich zu kopieren, muss die Funktion copy() verwendet werden.
# kopieren dt2 nach neu2
neu2 <- copy(dt2)
# anzeigen
str(neu2)Classes 'data.table' and 'data.frame': 10 obs. of 4 variables:
$ Geschlecht: chr "m" "w" "w" "m" ...
$ Alter : int 28 18 25 29 21 19 27 26 31 22
$ Gewicht : int 80 55 74 101 84 74 65 56 88 78
$ Groesse : int 170 174 183 190 185 178 169 163 189 184
- attr(*, ".internal.selfref")=<externalptr> # manipulieren
neu2[, Kuckuck := Groesse/Gewicht]
# dt2 ist unverändert
str(dt2)Classes 'data.table' and 'data.frame': 10 obs. of 4 variables:
$ Geschlecht: chr "m" "w" "w" "m" ...
$ Alter : int 28 18 25 29 21 19 27 26 31 22
$ Gewicht : int 80 55 74 101 84 74 65 56 88 78
$ Groesse : int 170 174 183 190 185 178 169 163 189 184
- attr(*, ".internal.selfref")=<externalptr> 29.11 pipen
Innerhalb von data.table kann auch die Pipe verwendet werden. Wird die R-Base-Pipe |> verwendet, kann mittels Unterstrich _ auf den weitergeleiteten Datenstrom zugegriffen werden. Bei der Tidyverse-Pipe (eigentlich von magrittr) mit der Zeichenfolge %>% muss ein Punkt . verwendet werden.
Folgende Aufrufe filtern das geschlecht und pipen den Datenstrom weiter. Anschließend wird nach alter sortiert.
# Daten pipen mit R_Base
dt2[Geschlecht=="m"] |>
_[order(Alter)] Geschlecht Alter Gewicht Groesse
<char> <int> <int> <int>
1: m 21 84 185
2: m 22 78 184
3: m 28 80 170
4: m 29 101 190
5: m 31 88 189# Daten pipen mit magrittr
dt2[Geschlecht=="m"] %>%
.[order(Alter)] Geschlecht Alter Gewicht Groesse
<char> <int> <int> <int>
1: m 21 84 185
2: m 22 78 184
3: m 28 80 170
4: m 29 101 190
5: m 31 88 189Oder wir erstellen ein linerares Modell und pipen es an die summary()-Funktion weiter.
dt2[, lm(Gewicht ~ Groesse)] |>
summary()
Call:
lm(formula = Gewicht ~ Groesse)
Residuals:
Min 1Q Median 3Q Max
-14.9024 -3.4756 -0.3902 1.0915 15.0732
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -146.5366 58.3503 -2.511 0.03630 *
Groesse 1.2439 0.3265 3.810 0.00516 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 8.992 on 8 degrees of freedom
Multiple R-squared: 0.6447, Adjusted R-squared: 0.6003
F-statistic: 14.51 on 1 and 8 DF, p-value: 0.005164Wir können den Ausdruck aber auch direkt in die summary()-Funktion schreiben.
summary(dt2[, lm(Gewicht ~ Groesse)])
Call:
lm(formula = Gewicht ~ Groesse)
Residuals:
Min 1Q Median 3Q Max
-14.9024 -3.4756 -0.3902 1.0915 15.0732
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) -146.5366 58.3503 -2.511 0.03630 *
Groesse 1.2439 0.3265 3.810 0.00516 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 8.992 on 8 degrees of freedom
Multiple R-squared: 0.6447, Adjusted R-squared: 0.6003
F-statistic: 14.51 on 1 and 8 DF, p-value: 0.00516429.12 Ergebnisse gruppieren mit by
Über den Paramter by können die Ergebnisse gruppiert werden.
# gruppiert nach Geschlecht
dt[, .(Median = median(alter),
Mittelw = mean(alter),
Stdabw = sd(alter)),
by = geschlecht] geschlecht Median Mittelw Stdabw
<fctr> <num> <num> <num>
1: weiblich 21.5 24.10714 5.251732
2: männlich 25.0 29.00000 9.617692Die Ausgabe kann gepipet und weiterverarbeitet werden. In folgendem Beispiel berechnen wir den Variationskoeffizienten (\(sd / \bar x\)) aus den gruppierten Ergebnissen.
dt[, .(Median = median(alter),
Mittelw = mean(alter),
Stdabw = sd(alter)),
by = geschlecht] |>
# berechnen
_[, VK := Stdabw / Mittelw] |>
# anzeigen
_[] geschlecht Median Mittelw Stdabw VK
<fctr> <num> <num> <num> <num>
1: weiblich 21.5 24.10714 5.251732 0.2178496
2: männlich 25.0 29.00000 9.617692 0.3316446Bitte beachten Sie, dass wir in diesem Beispiel die Anzeige der Endergebnisse mittels |> _[] erzwingen mussten. Dies ist notwendig, wenn per by gruppierte Ergebnisse weiter manipuliert werden sollen. Data.table speichert Änderungen durch := immer direkt im Objekt, wobei keine Ausgabe der Daten erfolgt. Im vorliegenden Fall von VK := Stdabw / Mittelw ist diese Speicherung jedoch nicht möglich (ausgegeben wird ja eh nichts), da sich das Endergebnis nicht mehr auf das ursprüngliche Objekt dt bezieht. In diesem Fall ist es (sogar) möglich und üblich, das Ergebnis wie gewohnt in einem neuen Objekt zu speichern, ohne dass dabei ein symbolischer Link angelegt wird.
neu3 <- dt[, .(Median = median(alter),
Mittelw = mean(alter),
Stdabw = sd(alter)),
by = geschlecht] |>
_[, VK := Stdabw / Mittelw] |>
_[]
# anzeigen
neu3 geschlecht Median Mittelw Stdabw VK
<fctr> <num> <num> <num> <num>
1: weiblich 21.5 24.10714 5.251732 0.2178496
2: männlich 25.0 29.00000 9.617692 0.3316446Wir können den letzten Pipevorgang abkürzen, indem wir einfach eckige Klammern [] an unseren Aufruf anhängen.
neu4 <- dt[, .(Median = median(alter),
Mittelw = mean(alter),
Stdabw = sd(alter)),
by = geschlecht] |>
_[, VK := Stdabw / Mittelw][]
# anzeigen
neu4 geschlecht Median Mittelw Stdabw VK
<fctr> <num> <num> <num> <num>
1: weiblich 21.5 24.10714 5.251732 0.2178496
2: männlich 25.0 29.00000 9.617692 0.331644629.13 Weitere Funktionen aus dem data.table Paket
Das Paket data.table bringt zahlreiche eigene Funktionen mit, um typische Aufgabenstellungen effizienter bearbeiten zu können.
29.13.1 Einzigartige bestimmen mit uniqueN()
Um zum Beispiel die Anzahl verschiedener Städte innerhalb der Variable geburtsort zu bestimmen, können wir auf die paketeigene Funktion uniqueN() zurückgreifen:
# wieviele unterschiedliche Städte sind in "geburtsort"?
dt[, uniqueN(geburtsort)][1] 2629.13.2 Anzahl der Fälle mit .N
Mit der Funktion .N kann die Anzahl der Fälle ermittelt werden.
dt[, .(Anzahl = .N),
by = geschlecht] geschlecht Anzahl
<fctr> <int>
1: weiblich 28
2: männlich 9Mit Hilfe von nrow() können so prozentuale Anteile berechnet werden.
dt[, .(Anzahl = .N,
Prozent = .N/nrow(dt)*100),
by = alterK] alterK Anzahl Prozent
<ord> <int> <num>
1: (0,20] 9 24.324324
2: (25,30] 6 16.216216
3: (40,50] 2 5.405405
4: (30,40] 5 13.513514
5: (20,25] 15 40.540541Die Ergebnisse können an ggplot() weitergereicht werden.
# ggplot
library(ggplot2)
dt[, .(Anzahl = .N,
Prozent = .N/nrow(dt)*100),
by = alterK] |>
ggplot(aes(x=alterK, y=Prozent)) +
geom_col(color="black", fill="orchid")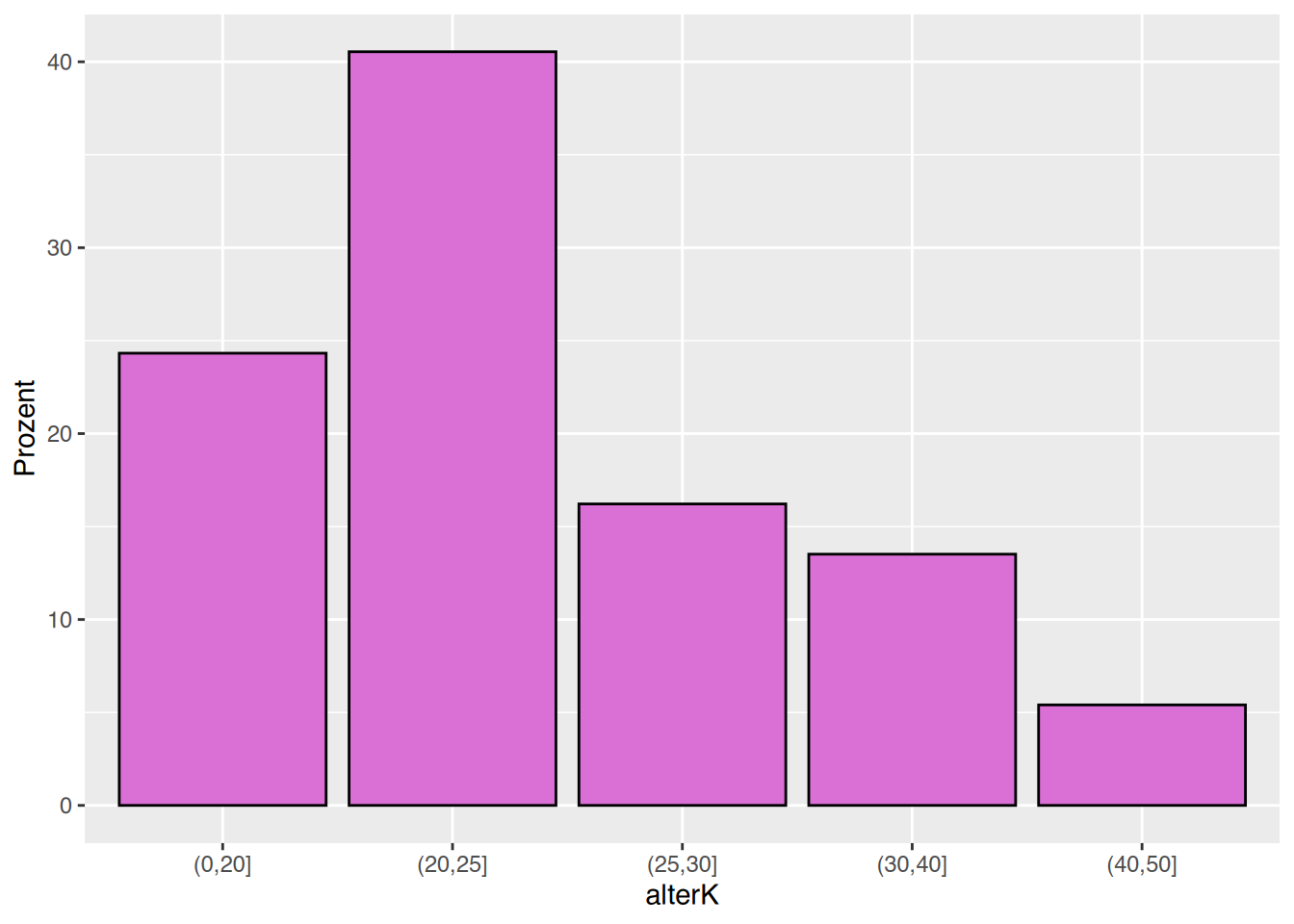
29.13.3 Lange Tabelle erzeugen mit melt()
Mit der Funktion melt() können breite Tabellen in lange (tidy) umgewandelt werden, ähnlich wie mit dplyr::pivot_longer(). Zur Demonstration verwenden wir die Pflegetabelle von Isfort (2018)
# lade Testdaten
load("https://www.produnis.de/R/data/Pflegeberufe.RData") 1999 2001 2003 2005 2007 2009 2011 2013
Krankenpflegeassistenz 16624 19061 19478 21537 27731 36481 46517 54371
Altenpflegehilfe 55770 52710 49727 45776 48326 47903 47978 48363
Kinderkrankenpflege 47779 48203 48822 48519 49080 49307 48291 48937
Krankenpflege 430983 436767 444783 449355 457322 465446 468192 472580
Altenpflege 109161 124879 141965 158817 178902 194195 208304 227154
2015
Krankenpflegeassistenz 64127
Altenpflegehilfe 49507
Kinderkrankenpflege 48913
Krankenpflege 476416
Altenpflege 246412Die Tabelle ist nicht tidy und liegt im breiten Format vor. Ausserdem ist sie von der Klasse matrix.
# wandle um in data.table
pf <- as.data.table(Pflegeberufe, keep.rownames = "Berufsgruppe")
# anzeigen
pf Berufsgruppe 1999 2001 2003 2005 2007 2009 2011
<char> <num> <num> <num> <num> <num> <num> <num>
1: Krankenpflegeassistenz 16624 19061 19478 21537 27731 36481 46517
2: Altenpflegehilfe 55770 52710 49727 45776 48326 47903 47978
3: Kinderkrankenpflege 47779 48203 48822 48519 49080 49307 48291
4: Krankenpflege 430983 436767 444783 449355 457322 465446 468192
5: Altenpflege 109161 124879 141965 158817 178902 194195 208304
2013 2015
<num> <num>
1: 54371 64127
2: 48363 49507
3: 48937 48913
4: 472580 476416
5: 227154 246412Mittels melt() transformieren wir pf in eine lange (tidy) Tabelle. Dabei übergeben wir dem Parameter
id.varsalle Variabelen, welche “Identifikatoren” beinhalten. Damit sind alle Spalten gemeint, die keine konkrekten Messwerte enhalten, sondern weitere bezeichnende Kennwerte. Klassischer Weise sind dies vor allem die Zeilennamen, in unserem Falle alsoBerufsgruppe. Es können mehrereid.varsmittelsc()aneinandergereiht werden.measure.varsalle Spalten, welche die eigentlichen Messwerte enthalten, in unserem Falle 1999:2015 (alles außerBerufsgruppe). Wird dieser Parameter leer gelassen, nimmtdata.tableautomatisch alle Spalten, die keineid.varssind.variable.nameden Name der neuen Spalte, in welche die Bezeichnungen dermeasure.varsüberführt werden sollen, in unserem FallJahr.value.nameden Name der neuen Spalte, in welche die Werte dermeasure.varsüberführt werden sollen, in unserem FallAnzahl.
Da wir alle Spalten außer Berufsgruppe melten wollen, kann der Parameter measure.vars weggelassen werden.
# pf mit melt() tidy machen
pf_tidy <- melt(pf, id.vars = "Berufsgruppe",
variable.name = "Jahr",
value.name = "Anzahl")
# anschauen
head(pf_tidy) Berufsgruppe Jahr Anzahl
<char> <fctr> <num>
1: Krankenpflegeassistenz 1999 16624
2: Altenpflegehilfe 1999 55770
3: Kinderkrankenpflege 1999 47779
4: Krankenpflege 1999 430983
5: Altenpflege 1999 109161
6: Krankenpflegeassistenz 2001 1906129.13.4 Breite Tabelle erzeugen mit dcast()
Mittels dcast() können lange Tabellen wieder in breite Tabellen transformiert werden, so wie bei dplyr::pivot_wider().
Der Aufruf folgt der Semantik:
dcast(Bezeichner ~ Spaltenname, value.var = "Wertename")
wobei
Bezeichnerdie Spalten derid.varsmeint.Spaltennamedie Spalte mit dervariable.namemeint.value.varden Namen der Spalte meint, welche die konkreten Messwerte enthält. Diese muss in Anführungszeichen angegeben werden. Wird dieser Parameter weggelassen, versuchtdata.tabledie korrekte Spalte zu erraten (was einfach ist, wenn nur noch eine Spalte übrig bleibt).
# wandle pf_tdiy mit dcast() in breite Tabelle
pf_wide <- dcast(pf_tidy, Berufsgruppe ~ Jahr,
value.var = "Anzahl")
# anschauen
head(pf_wide)Key: <Berufsgruppe>
Berufsgruppe 1999 2001 2003 2005 2007 2009 2011
<char> <num> <num> <num> <num> <num> <num> <num>
1: Altenpflege 109161 124879 141965 158817 178902 194195 208304
2: Altenpflegehilfe 55770 52710 49727 45776 48326 47903 47978
3: Kinderkrankenpflege 47779 48203 48822 48519 49080 49307 48291
4: Krankenpflege 430983 436767 444783 449355 457322 465446 468192
5: Krankenpflegeassistenz 16624 19061 19478 21537 27731 36481 46517
2013 2015
<num> <num>
1: 227154 246412
2: 48363 49507
3: 48937 48913
4: 472580 476416
5: 54371 6412729.13.5 Subset of Data (.SD)
Subset of Data wird in der data.table-Syntax verwendet, um eine Teilmenge der Daten in einer speziellen Umgebung zu referenzieren. Die Funktion hierfür heisst .SD und enthält eine Auswahl an Spalten der data.table, die weiterverarbeitet werden können, z. B. durch Berechnungen oder Transformationen.
.SD ist besonders nützlich, wenn Sie Berechnungen oder Transformationen nur auf bestimmte Spalten anwenden möchten, während die anderen Spalten beibehalten werden sollen.
Schauen wir uns nochmal unser Objekt pf an.
str(pf)Classes 'data.table' and 'data.frame': 5 obs. of 10 variables:
$ Berufsgruppe: chr "Krankenpflegeassistenz" "Altenpflegehilfe" "Kinderkrankenpflege" "Krankenpflege" ...
$ 1999 : num 16624 55770 47779 430983 109161
$ 2001 : num 19061 52710 48203 436767 124879
$ 2003 : num 19478 49727 48822 444783 141965
$ 2005 : num 21537 45776 48519 449355 158817
$ 2007 : num 27731 48326 49080 457322 178902
$ 2009 : num 36481 47903 49307 465446 194195
$ 2011 : num 46517 47978 48291 468192 208304
$ 2013 : num 54371 48363 48937 472580 227154
$ 2015 : num 64127 49507 48913 476416 246412
- attr(*, ".internal.selfref")=<externalptr> Nun verwenden wir .SD, um für jede Spalte den Mittelwert zu berechnen.
pf[, lapply(.SD, mean)]Warning in mean.default(X[[i]], ...): Argument ist weder numerisch noch
boolesch: gebe NA zurück Berufsgruppe 1999 2001 2003 2005 2007 2009 2011
<num> <num> <num> <num> <num> <num> <num> <num>
1: NA 132063.4 136324 140955 144800.8 152272.2 158666.4 163856.4
2013 2015
<num> <num>
1: 170281 177075Der Aufruf lapply(.SD, mean) wendet die Funktion mean auf jede Spalte in .SD an.
29.13.5.1 Verwendung von .SDcols
.SDcols ist ein optionaler Parameter, mit dem die Spalten, die in .SD enthalten sind, gezielt ausgewählt werden können.
Schauen wir uns das Objekt dt an.
str(dt)Classes 'data.table' and 'data.frame': 37 obs. of 9 variables:
$ alter : int 20 28 41 34 26 38 28 21 27 26 ...
$ geschlecht: Factor w/ 2 levels "männlich","weiblich": 2 2 1 2 2 2 2 1 2 1 ...
$ stifte : int 12 7 1 13 18 25 29 1 2 5 ...
$ geburtsort: Factor w/ 26 levels "Bagdad","Bonn",..: 9 21 2 10 8 5 21 7 18 10 ...
$ fahrzeit : int 1 45 60 25 15 50 40 60 60 40 ...
$ podcast : Ord.factor w/ 5 levels "nie"<"selten"<..: 2 2 2 4 NA 4 4 3 1 1 ...
$ FahrzeitH : num 0.0167 0.75 1 0.4167 0.25 ...
$ alterK : Ord.factor w/ 5 levels "(0,20]"<"(20,25]"<..: 1 3 5 4 3 4 3 2 3 3 ...
$ kuckuck : int 12 315 60 325 270 1250 1160 60 120 200 ...
- attr(*, ".internal.selfref")=<externalptr>
- attr(*, "index")= int(0) Wir können nun den Median für alle mindestens ordinalskalierten Variablen (alter, stifte, fahrzeit) berechnen, indem wir diese Variablen per .SDcols angeben.
# Wähle die Spalten aus, die verwendet werden sollen
dt[, lapply(.SD, median), .SDcols=c("alter", "stifte", "fahrzeit")] alter stifte fahrzeit
<int> <int> <int>
1: 22 8 60Dies klappt auch mit Gruppierungen. Wir berechnen die Werte erneut, diesmal aber getrennt nach geschlecht.
dt[, lapply(.SD, median),
by=geschlecht, .SDcols=c("alter", "stifte", "fahrzeit")] geschlecht alter stifte fahrzeit
<fctr> <num> <num> <num>
1: weiblich 21.5 12 60
2: männlich 25.0 5 57Auf diese Weise können Spalten auch transformiert werden. Angenommen, wir möchten die Werte für alter und stifte verdoppeln, dann lautet der Aufruf:
# Verdopple alter und stifte
dt[, (c("alter", "stifte")) := lapply(.SD, function(x) x*2),
.SDcols = c("alter", "stifte")]
head(dt) alter geschlecht stifte geburtsort fahrzeit podcast FahrzeitH alterK
<num> <fctr> <num> <fctr> <int> <ord> <num> <ord>
1: 40 weiblich 24 Düren 1 selten 0.01666667 (0,20]
2: 56 weiblich 14 Neuss 45 selten 0.75000000 (25,30]
3: 82 männlich 2 Bonn 60 selten 1.00000000 (40,50]
4: 68 weiblich 26 Düsseldorf 25 oft 0.41666667 (30,40]
5: 52 weiblich 36 Duisburg 15 <NA> 0.25000000 (25,30]
6: 76 weiblich 50 Dinslaken 50 oft 0.83333333 (30,40]
kuckuck
<int>
1: 12
2: 315
3: 60
4: 325
5: 270
6: 125029.14 Cheat Sheet und Übungsaufgaben
Auf GitHub ist ein schöner Cheat-Sheet für data.table vorhanden. Das PDF können Sie unter https://raw.githubusercontent.com/rstudio/cheatsheets/master/datatable.pdf herunterladen.
Des weiteren stehen mit dem table traineR (große Schlarmann (2025b)) eine Reihe an Übungsaufgaben bereit, an denen Sie Ihre data.table-Fähigkeiten ausprobieren können, siehe https://www.produnis.de/tabletrainer/.