34 Diagramme plotten
In diesem Kapitel werden die Plotfunktionen des klassischen R vorgestellt. Sie erzeugen immernoch brauchbare Ergebnisse, und man kann viele Einstellungen vornehmen. Wenn Sie allerdings ein etwas komplexeres Diagramm aufbauen möchten, lohnt es sich nicht, die Spezialeinstellungen der klassischen Plotfunktionen zu durchstöbern. Schwenken Sie in diesem Falle lieber gleich zum moderneren ggplot() um, welches in Kapitel Kapitel 35 vorgestellt wird. So kommen Sie viel “einfacher” an Ihre Ziel.
Die universelle Funktion zum erstellen von Diagrammen heisst plot(). Mit ihr werden ein Großteil der Diagramme erstellt, und viele Funktionen rufen letztendlich auch nur plot() auf.
Da plot() so universal ist, nimmt sie viele Parameter entgegen. Die nachfolgende Tabelle zeigt diese Parameter in einer Übersicht. Viele der Parameter sind auch in weiteren Plotfunktionen implementiert. Es kann dennoch nie schaden, sich mit ?barplot in der R-Konsole die explizieten Parameter zu vergegenwärtigen.
| Parameter | Argumente / Beschreibung | Beispiel |
|---|---|---|
type |
legt den Diagrammtyp fest | type="p" |
| (mittlerweile veraltet, | l = Liniendiagramm |
|
| funktioniert aber noch) | p = Punktdiagramm |
|
b = (both) Kombination aus l und p |
||
| s = Stufendiagramm | ||
| h = Histogramm-artig(!) | ||
| n = keine Darstellung | ||
col |
Füllfarbe | col="blue" |
(colors() zeigt alle Farben) |
col="#112233" (RGB) |
|
gray = schwarz bis gray99 = weiß |
col="gray77" |
|
main |
Überschrift | main="Mein Plot" |
sub |
Untertitel | main="Abbildung 12" |
xlab |
Beschriftung der X-Achse | xlab="Jahrgänge" |
ylab |
Beschriftung der Y-Achse | ylab="Häufigkeit" |
xlim |
Start- und Endwert der X-Achse | xlim=c(2,10) |
ylim |
Start- und Endwert der Y-Achse | ylim=c(0,100) |
xaxt |
X-Achse komplett unbeschriftet | xaxt="n" |
yaxt |
Y-Achse komplett unbeschriftet | yaxt="n" |
pch |
Darstellungssymbol | pch=4 |
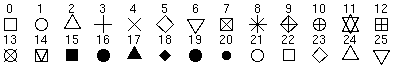 |
||
cex |
Symbolgröße | cex=2 |
lty |
Linientyp | lty=1 |
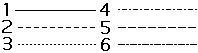 |
||
lwd |
Strichstärke der Linien | lwd=3 |
bty |
Boxtyp, setzt Rahmen ums Plot | bty="o" |
n = kein Rahmen |
||
o = komplett |
||
C = oben, unten und links |
||
L = unten und links |
||
u = unten, rechts und links |
||
7 = oben und rechts |
||
| (die Buchstaben sehen so aus wie der Rahmen) | ||
font |
Fontformat | font=2 |
1 = normal |
||
2 = fett |
||
3 = kursiv |
||
5 = griechische Symbole |
34.1 Punktwolke
Die Punktwolke wird mit besagter Funktion plot() erstellt. Ihr werden zwei Vektoren mit den entsprechenden x und y Werten übergeben.
# erzeuge Testwerte
x <- sort(rnorm(100, mean=12, sd=2))
y <- sort(rnorm(100, mean=140, sd=15))
# Plotten
plot(x,y) 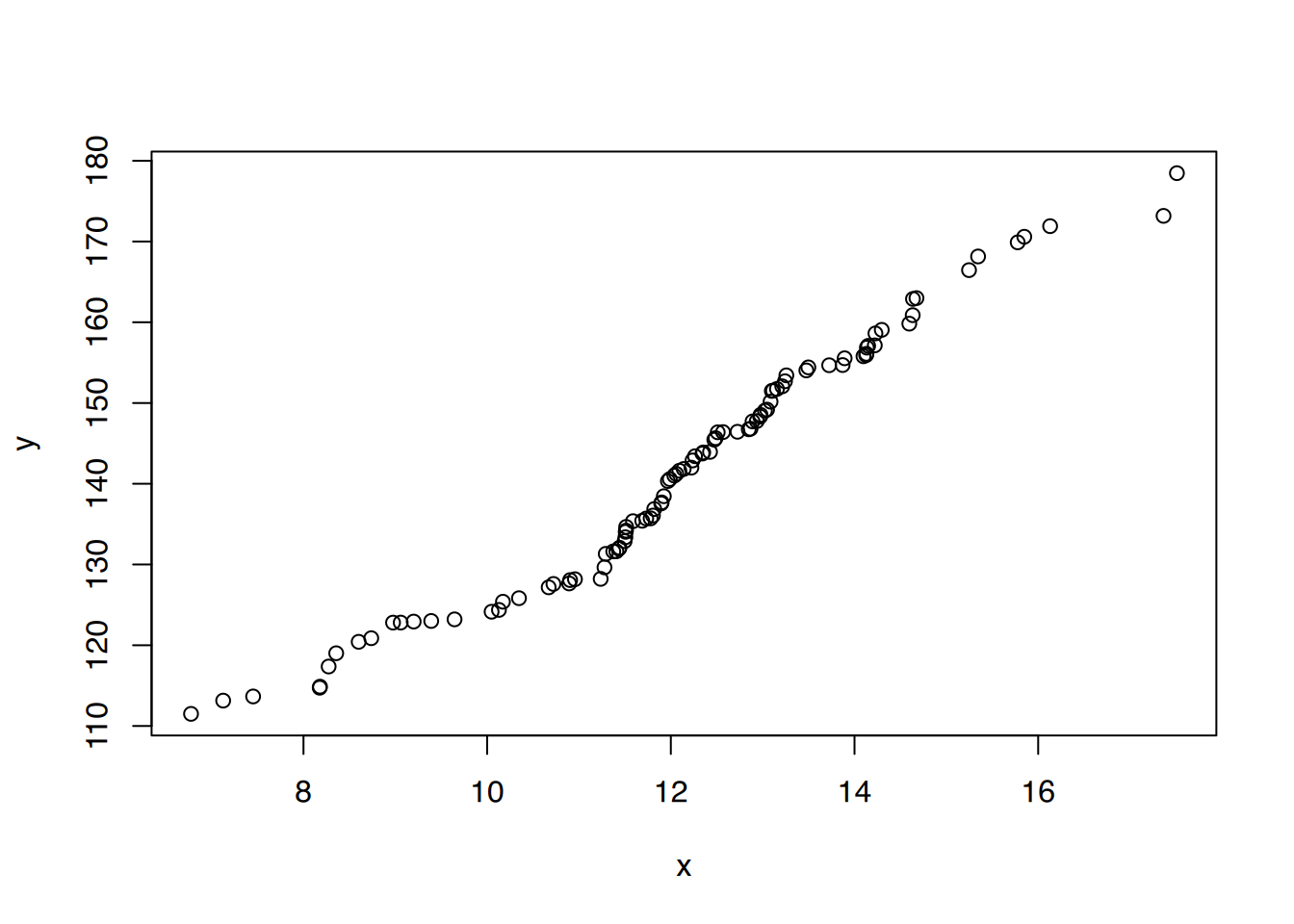
Über die Parameter aus der Tabelle kann das Plot aufgehübscht werden.
plot(x,y, col="blue",
ylab="Körpergröße", xlab="Alter",
main="Ein Plot mit R base",
sub="Dieses Plot wurde aus Zufallswerten erstellt",
pch=13) 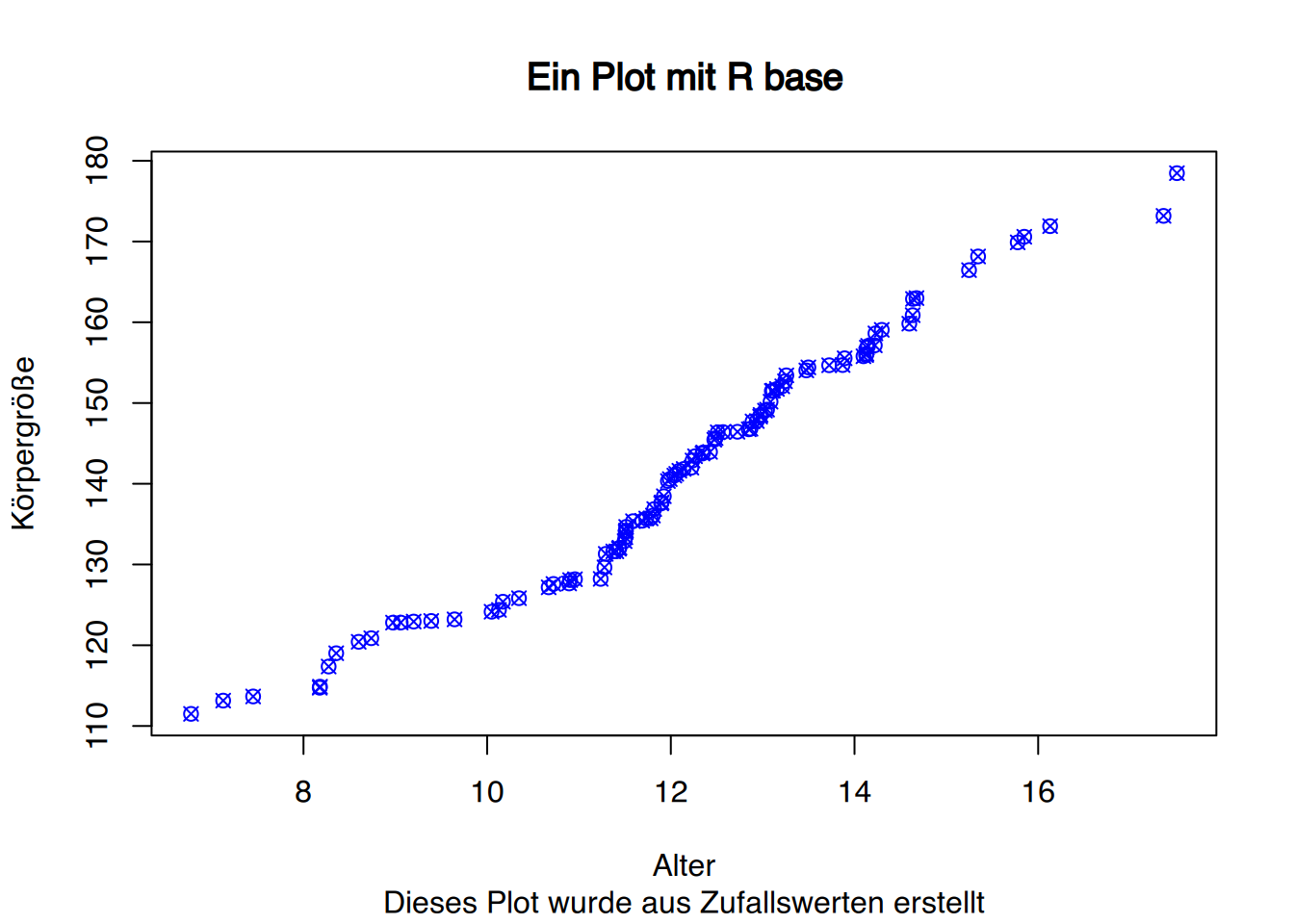
Mit der Funktion abline() kann dem Plot eine Gerade hinzugefügt werden, z.B. eine Regressionsgerade. Die Funktion benötigt als Angaben den Schnitt durch die Y-Achse sowie den Steigungswert. Die Funktion lm() liefert für Regressionsgeraden genau diese Werte.
# Regressionswerte von Körpergröße ~ Alter
lm(y~x)##
## Call:
## lm(formula = y ~ x)
##
## Coefficients:
## (Intercept) x
## 55.563 7.096Das heisst, wir rufen die Funktion innerhalb von abline() auf:
plot(x,y, col="blue",
ylab="Körpergröße", xlab="Alter",
main="Ein Plot mit R base",
sub="Dieses Plot wurde aus Zufallswerten erstellt",
pch=13)
abline(lm(y~x))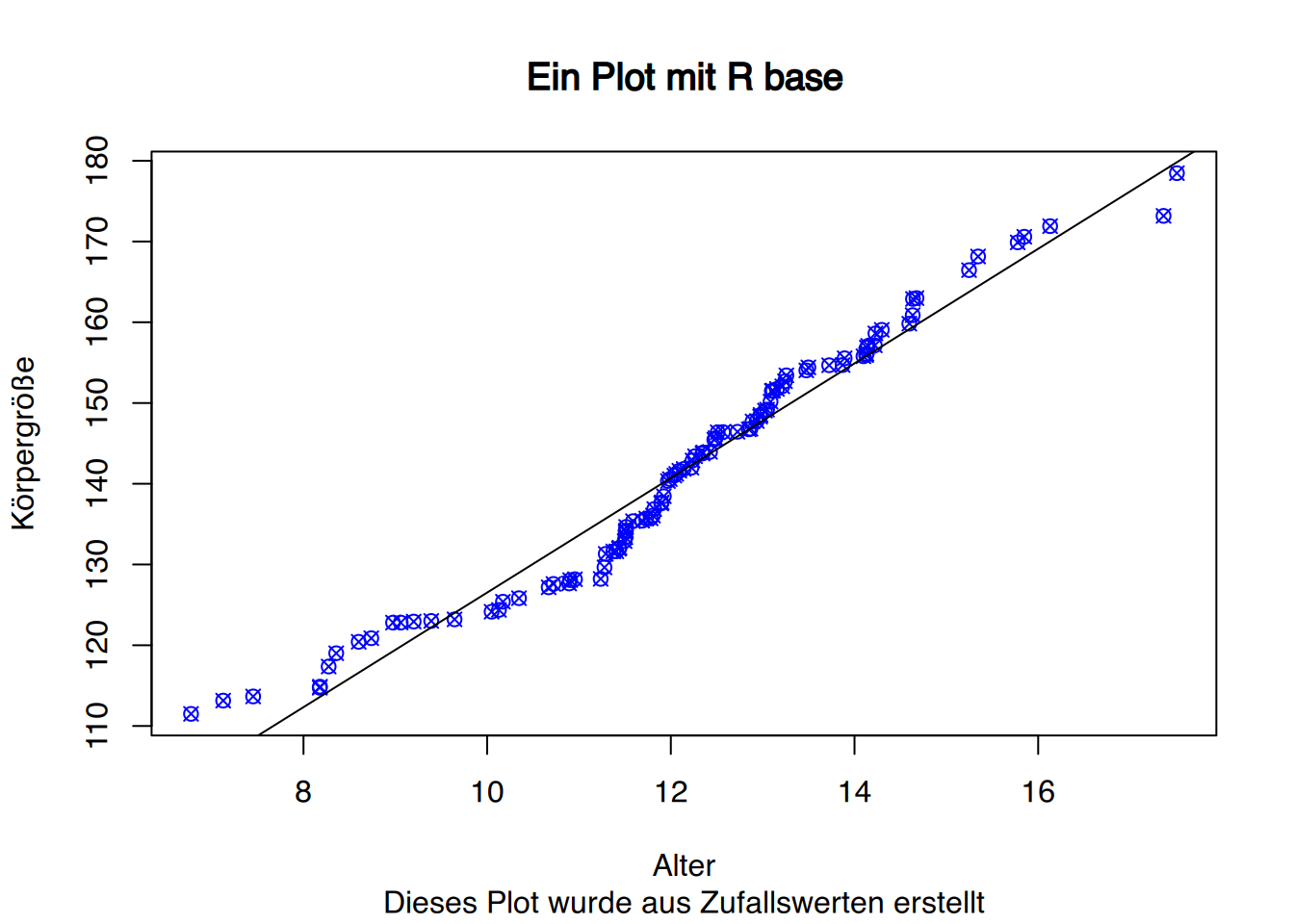
In rot sieht sie evtl. besser aus:
plot(x,y, col="blue",
ylab="Körpergröße", xlab="Alter",
main="Ein Plot mit R base",
sub="Dieses Plot wurde aus Zufallswerten erstellt",
pch=13)
abline(lm(y~x), col="red")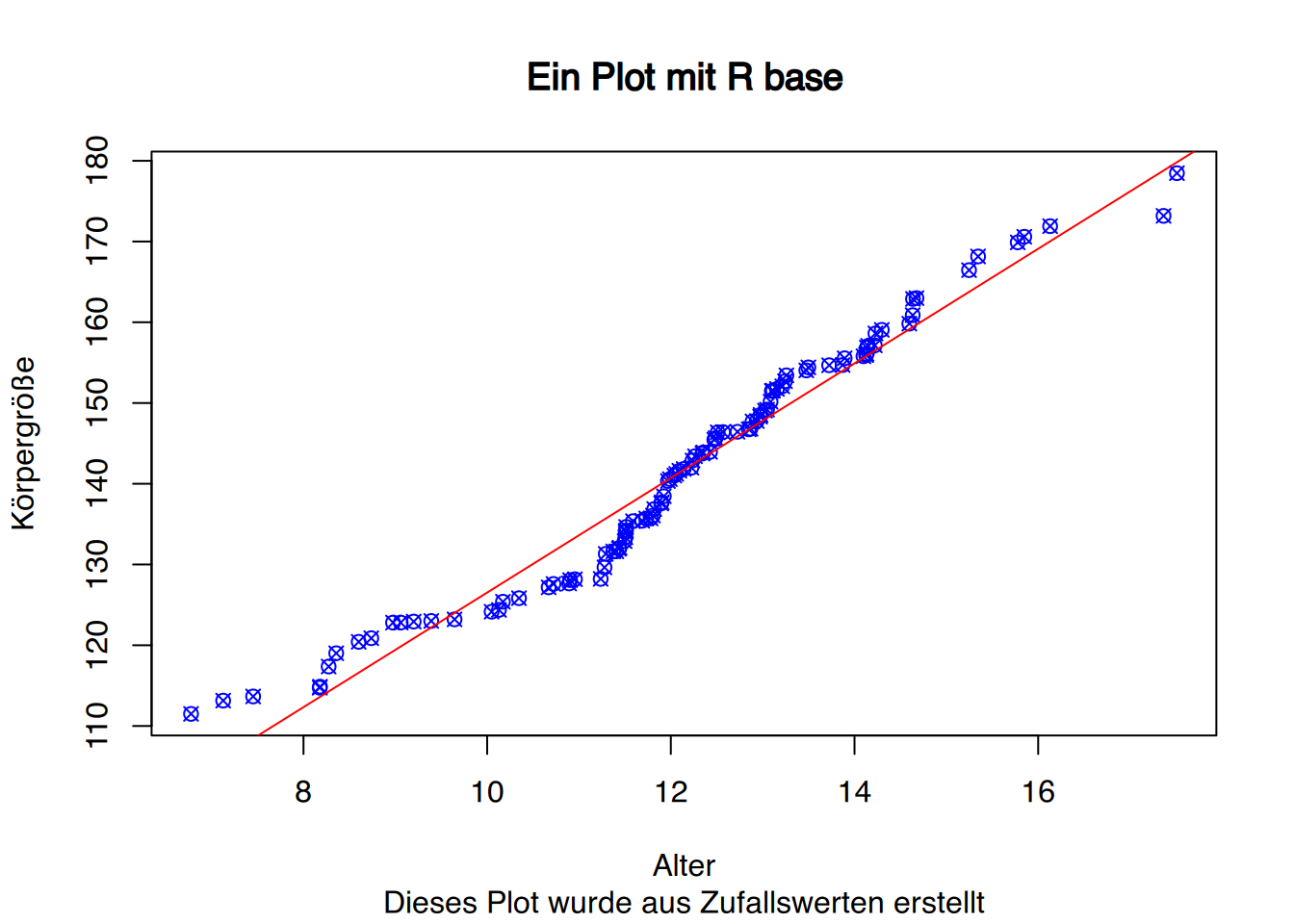
Mit der Funktion lines() können Linien hinzugefügt werden. Dafür übergeben wir die entsprechenden Koordinaten des Start- und Endpunkts. So können wir z.B. den Mittelwert für Alter markieren.
# Mittelwer als x-Koordinate
mx <- mean(x)
# Berechne Schnittpunkt mit Regressionsgeraden
ymx <- coef(lm(y~x))[1] + mx*coef(lm(y~x))[2]
plot(x,y, col="blue",
ylab="Körpergröße", xlab="Alter",
main="Ein Plot mit R base",
sub="Dieses Plot wurde aus Zufallswerten erstellt",
pch=13)
abline(lm(y~x), col="red")
# Füge Mittelwertlinie hinzu
lines(x=c(mx, mx), y=c(0, ymx), lty=3)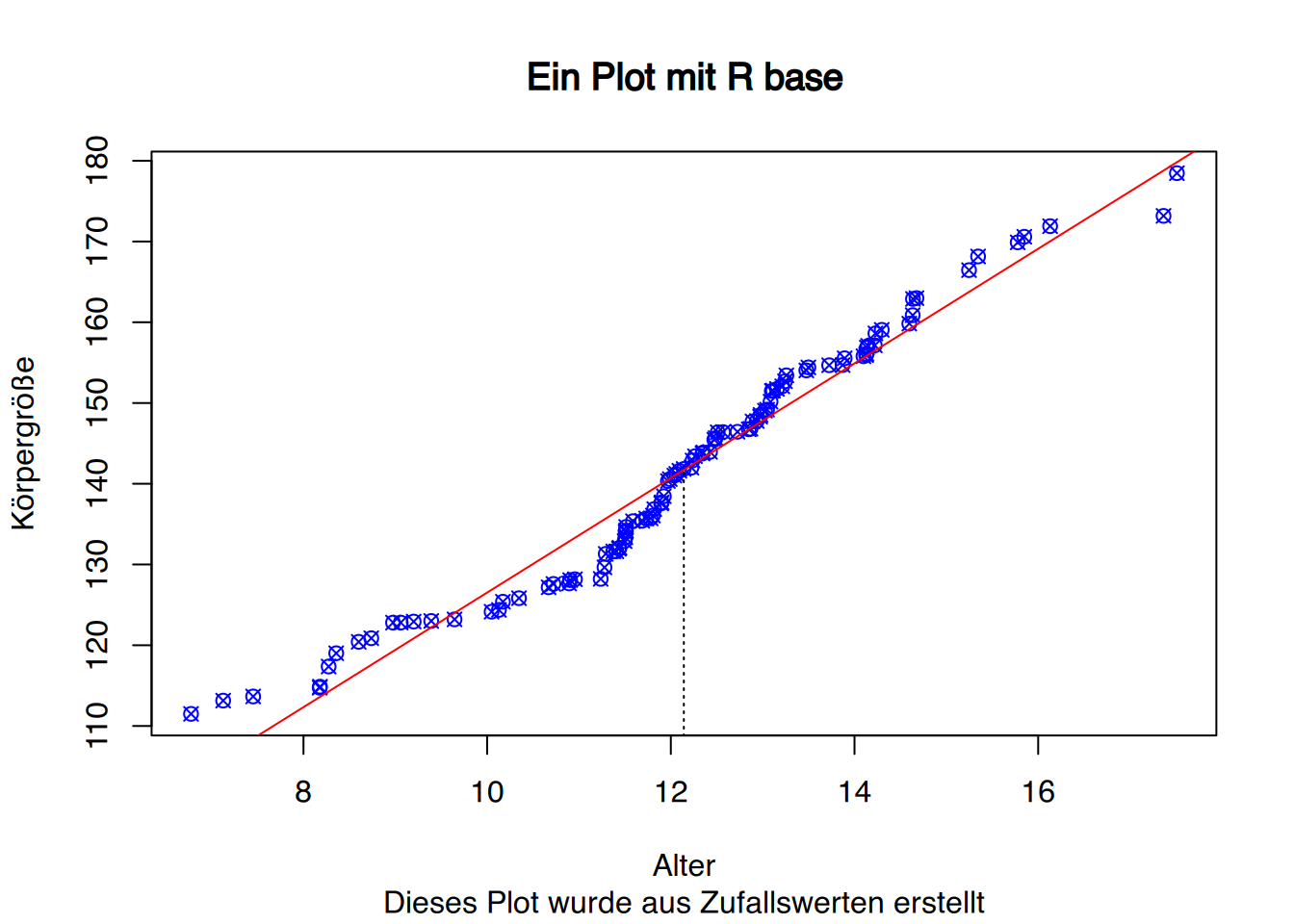
Liegen mehrere Variablen in einem Datenframe vor, stellt plot() nach Kombinationen getrennt dar.
x <- data.frame(Lesetest = sort(rnorm(100, mean=20, sd=6)),
Rechentest = rnorm(100, mean=15, sd=12),
Rechtschreibung = sort(rnorm(100, mean=21, sd=4))
)
plot(x)
Je mehr Variablen vorliegen, desto komplexer wird der Plot.
x <- data.frame(Lesetest = sort(rnorm(100, mean=20, sd=6)),
Rechentest = rnorm(100, mean=15, sd=12),
Rechtschreibung = sort(rnorm(100, mean=21, sd=4)),
Grammatikfehler = sort(rnorm(100, mean=21, sd=4), decreasing=T)
)
plot(x)
Mit Farben kann man es etwas aufhübschen.
x <- data.frame(Lesetest = sort(rnorm(100, mean=20, sd=6)),
Rechentest = rnorm(100, mean=15, sd=12),
Rechtschreibung = sort(rnorm(100, mean=21, sd=4)),
Grammatikfehler = sort(rnorm(100, mean=21, sd=4), decreasing=T)
)
plot(x, col=rainbow(3))
34.2 verfügbare Farben
Die Funktion colors() zeigt die verfügbaren Farben an.
# zeige verfügbare Farben an
colors()## [1] "white" "aliceblue" "antiquewhite"
## [4] "antiquewhite1" "antiquewhite2" "antiquewhite3"
## [7] "antiquewhite4" "aquamarine" "aquamarine1"
## [10] "aquamarine2" "aquamarine3" "aquamarine4"
## [13] "azure" "azure1" "azure2"
## [16] "azure3" "azure4" "beige"
## [19] "bisque" "bisque1" "bisque2"
## [22] "bisque3" "bisque4" "black"
## [25] "blanchedalmond" "blue" "blue1"
## [28] "blue2" "blue3" "blue4"
## [31] "blueviolet" "brown" "brown1"
## [34] "brown2" "brown3" "brown4"
## [37] "burlywood" "burlywood1" "burlywood2"
## [40] "burlywood3" "burlywood4" "cadetblue"
## [43] "cadetblue1" "cadetblue2" "cadetblue3"
## [46] "cadetblue4" "chartreuse" "chartreuse1"
## [49] "chartreuse2" "chartreuse3" "chartreuse4"
## [52] "chocolate" "chocolate1" "chocolate2"
## [55] "chocolate3" "chocolate4" "coral"
## [58] "coral1" "coral2" "coral3"
## [61] "coral4" "cornflowerblue" "cornsilk"
## [64] "cornsilk1" "cornsilk2" "cornsilk3"
## [67] "cornsilk4" "cyan" "cyan1"
## [70] "cyan2" "cyan3" "cyan4"
## [73] "darkblue" "darkcyan" "darkgoldenrod"
## [76] "darkgoldenrod1" "darkgoldenrod2" "darkgoldenrod3"
## [79] "darkgoldenrod4" "darkgray" "darkgreen"
## [82] "darkgrey" "darkkhaki" "darkmagenta"
## [85] "darkolivegreen" "darkolivegreen1" "darkolivegreen2"
## [88] "darkolivegreen3" "darkolivegreen4" "darkorange"
## [91] "darkorange1" "darkorange2" "darkorange3"
## [94] "darkorange4" "darkorchid" "darkorchid1"
## [97] "darkorchid2" "darkorchid3" "darkorchid4"
## [100] "darkred" "darksalmon" "darkseagreen"
## [103] "darkseagreen1" "darkseagreen2" "darkseagreen3"
## [106] "darkseagreen4" "darkslateblue" "darkslategray"
## [109] "darkslategray1" "darkslategray2" "darkslategray3"
## [112] "darkslategray4" "darkslategrey" "darkturquoise"
## [115] "darkviolet" "deeppink" "deeppink1"
## [118] "deeppink2" "deeppink3" "deeppink4"
## [121] "deepskyblue" "deepskyblue1" "deepskyblue2"
## [124] "deepskyblue3" "deepskyblue4" "dimgray"
## [127] "dimgrey" "dodgerblue" "dodgerblue1"
## [130] "dodgerblue2" "dodgerblue3" "dodgerblue4"
## [133] "firebrick" "firebrick1" "firebrick2"
## [136] "firebrick3" "firebrick4" "floralwhite"
## [139] "forestgreen" "gainsboro" "ghostwhite"
## [142] "gold" "gold1" "gold2"
## [145] "gold3" "gold4" "goldenrod"
## [148] "goldenrod1" "goldenrod2" "goldenrod3"
## [151] "goldenrod4" "gray" "gray0"
## [154] "gray1" "gray2" "gray3"
## [157] "gray4" "gray5" "gray6"
## [160] "gray7" "gray8" "gray9"
## [163] "gray10" "gray11" "gray12"
## [166] "gray13" "gray14" "gray15"
## [169] "gray16" "gray17" "gray18"
## [172] "gray19" "gray20" "gray21"
## [175] "gray22" "gray23" "gray24"
## [178] "gray25" "gray26" "gray27"
## [181] "gray28" "gray29" "gray30"
## [184] "gray31" "gray32" "gray33"
## [187] "gray34" "gray35" "gray36"
## [190] "gray37" "gray38" "gray39"
## [193] "gray40" "gray41" "gray42"
## [196] "gray43" "gray44" "gray45"
## [199] "gray46" "gray47" "gray48"
## [202] "gray49" "gray50" "gray51"
## [205] "gray52" "gray53" "gray54"
## [208] "gray55" "gray56" "gray57"
## [211] "gray58" "gray59" "gray60"
## [214] "gray61" "gray62" "gray63"
## [217] "gray64" "gray65" "gray66"
## [220] "gray67" "gray68" "gray69"
## [223] "gray70" "gray71" "gray72"
## [226] "gray73" "gray74" "gray75"
## [229] "gray76" "gray77" "gray78"
## [232] "gray79" "gray80" "gray81"
## [235] "gray82" "gray83" "gray84"
## [238] "gray85" "gray86" "gray87"
## [241] "gray88" "gray89" "gray90"
## [244] "gray91" "gray92" "gray93"
## [247] "gray94" "gray95" "gray96"
## [250] "gray97" "gray98" "gray99"
## [253] "gray100" "green" "green1"
## [256] "green2" "green3" "green4"
## [259] "greenyellow" "grey" "grey0"
## [262] "grey1" "grey2" "grey3"
## [265] "grey4" "grey5" "grey6"
## [268] "grey7" "grey8" "grey9"
## [271] "grey10" "grey11" "grey12"
## [274] "grey13" "grey14" "grey15"
## [277] "grey16" "grey17" "grey18"
## [280] "grey19" "grey20" "grey21"
## [283] "grey22" "grey23" "grey24"
## [286] "grey25" "grey26" "grey27"
## [289] "grey28" "grey29" "grey30"
## [292] "grey31" "grey32" "grey33"
## [295] "grey34" "grey35" "grey36"
## [298] "grey37" "grey38" "grey39"
## [301] "grey40" "grey41" "grey42"
## [304] "grey43" "grey44" "grey45"
## [307] "grey46" "grey47" "grey48"
## [310] "grey49" "grey50" "grey51"
## [313] "grey52" "grey53" "grey54"
## [316] "grey55" "grey56" "grey57"
## [319] "grey58" "grey59" "grey60"
## [322] "grey61" "grey62" "grey63"
## [325] "grey64" "grey65" "grey66"
## [328] "grey67" "grey68" "grey69"
## [331] "grey70" "grey71" "grey72"
## [334] "grey73" "grey74" "grey75"
## [337] "grey76" "grey77" "grey78"
## [340] "grey79" "grey80" "grey81"
## [343] "grey82" "grey83" "grey84"
## [346] "grey85" "grey86" "grey87"
## [349] "grey88" "grey89" "grey90"
## [352] "grey91" "grey92" "grey93"
## [355] "grey94" "grey95" "grey96"
## [358] "grey97" "grey98" "grey99"
## [361] "grey100" "honeydew" "honeydew1"
## [364] "honeydew2" "honeydew3" "honeydew4"
## [367] "hotpink" "hotpink1" "hotpink2"
## [370] "hotpink3" "hotpink4" "indianred"
## [373] "indianred1" "indianred2" "indianred3"
## [376] "indianred4" "ivory" "ivory1"
## [379] "ivory2" "ivory3" "ivory4"
## [382] "khaki" "khaki1" "khaki2"
## [385] "khaki3" "khaki4" "lavender"
## [388] "lavenderblush" "lavenderblush1" "lavenderblush2"
## [391] "lavenderblush3" "lavenderblush4" "lawngreen"
## [394] "lemonchiffon" "lemonchiffon1" "lemonchiffon2"
## [397] "lemonchiffon3" "lemonchiffon4" "lightblue"
## [400] "lightblue1" "lightblue2" "lightblue3"
## [403] "lightblue4" "lightcoral" "lightcyan"
## [406] "lightcyan1" "lightcyan2" "lightcyan3"
## [409] "lightcyan4" "lightgoldenrod" "lightgoldenrod1"
## [412] "lightgoldenrod2" "lightgoldenrod3" "lightgoldenrod4"
## [415] "lightgoldenrodyellow" "lightgray" "lightgreen"
## [418] "lightgrey" "lightpink" "lightpink1"
## [421] "lightpink2" "lightpink3" "lightpink4"
## [424] "lightsalmon" "lightsalmon1" "lightsalmon2"
## [427] "lightsalmon3" "lightsalmon4" "lightseagreen"
## [430] "lightskyblue" "lightskyblue1" "lightskyblue2"
## [433] "lightskyblue3" "lightskyblue4" "lightslateblue"
## [436] "lightslategray" "lightslategrey" "lightsteelblue"
## [439] "lightsteelblue1" "lightsteelblue2" "lightsteelblue3"
## [442] "lightsteelblue4" "lightyellow" "lightyellow1"
## [445] "lightyellow2" "lightyellow3" "lightyellow4"
## [448] "limegreen" "linen" "magenta"
## [451] "magenta1" "magenta2" "magenta3"
## [454] "magenta4" "maroon" "maroon1"
## [457] "maroon2" "maroon3" "maroon4"
## [460] "mediumaquamarine" "mediumblue" "mediumorchid"
## [463] "mediumorchid1" "mediumorchid2" "mediumorchid3"
## [466] "mediumorchid4" "mediumpurple" "mediumpurple1"
## [469] "mediumpurple2" "mediumpurple3" "mediumpurple4"
## [472] "mediumseagreen" "mediumslateblue" "mediumspringgreen"
## [475] "mediumturquoise" "mediumvioletred" "midnightblue"
## [478] "mintcream" "mistyrose" "mistyrose1"
## [481] "mistyrose2" "mistyrose3" "mistyrose4"
## [484] "moccasin" "navajowhite" "navajowhite1"
## [487] "navajowhite2" "navajowhite3" "navajowhite4"
## [490] "navy" "navyblue" "oldlace"
## [493] "olivedrab" "olivedrab1" "olivedrab2"
## [496] "olivedrab3" "olivedrab4" "orange"
## [499] "orange1" "orange2" "orange3"
## [502] "orange4" "orangered" "orangered1"
## [505] "orangered2" "orangered3" "orangered4"
## [508] "orchid" "orchid1" "orchid2"
## [511] "orchid3" "orchid4" "palegoldenrod"
## [514] "palegreen" "palegreen1" "palegreen2"
## [517] "palegreen3" "palegreen4" "paleturquoise"
## [520] "paleturquoise1" "paleturquoise2" "paleturquoise3"
## [523] "paleturquoise4" "palevioletred" "palevioletred1"
## [526] "palevioletred2" "palevioletred3" "palevioletred4"
## [529] "papayawhip" "peachpuff" "peachpuff1"
## [532] "peachpuff2" "peachpuff3" "peachpuff4"
## [535] "peru" "pink" "pink1"
## [538] "pink2" "pink3" "pink4"
## [541] "plum" "plum1" "plum2"
## [544] "plum3" "plum4" "powderblue"
## [547] "purple" "purple1" "purple2"
## [550] "purple3" "purple4" "red"
## [553] "red1" "red2" "red3"
## [556] "red4" "rosybrown" "rosybrown1"
## [559] "rosybrown2" "rosybrown3" "rosybrown4"
## [562] "royalblue" "royalblue1" "royalblue2"
## [565] "royalblue3" "royalblue4" "saddlebrown"
## [568] "salmon" "salmon1" "salmon2"
## [571] "salmon3" "salmon4" "sandybrown"
## [574] "seagreen" "seagreen1" "seagreen2"
## [577] "seagreen3" "seagreen4" "seashell"
## [580] "seashell1" "seashell2" "seashell3"
## [583] "seashell4" "sienna" "sienna1"
## [586] "sienna2" "sienna3" "sienna4"
## [589] "skyblue" "skyblue1" "skyblue2"
## [592] "skyblue3" "skyblue4" "slateblue"
## [595] "slateblue1" "slateblue2" "slateblue3"
## [598] "slateblue4" "slategray" "slategray1"
## [601] "slategray2" "slategray3" "slategray4"
## [604] "slategrey" "snow" "snow1"
## [607] "snow2" "snow3" "snow4"
## [610] "springgreen" "springgreen1" "springgreen2"
## [613] "springgreen3" "springgreen4" "steelblue"
## [616] "steelblue1" "steelblue2" "steelblue3"
## [619] "steelblue4" "tan" "tan1"
## [622] "tan2" "tan3" "tan4"
## [625] "thistle" "thistle1" "thistle2"
## [628] "thistle3" "thistle4" "tomato"
## [631] "tomato1" "tomato2" "tomato3"
## [634] "tomato4" "turquoise" "turquoise1"
## [637] "turquoise2" "turquoise3" "turquoise4"
## [640] "violet" "violetred" "violetred1"
## [643] "violetred2" "violetred3" "violetred4"
## [646] "wheat" "wheat1" "wheat2"
## [649] "wheat3" "wheat4" "whitesmoke"
## [652] "yellow" "yellow1" "yellow2"
## [655] "yellow3" "yellow4" "yellowgreen"Der Output ist 657 Farben lang.
# wieviele Farben gibt es?
length(colors())## [1] 657
Der R-Code, mit dem Abbildung 34.1 erstellt wurde, ist in Abschnitt 37.1 hinterlegt.
Eine weitere Übersicht der Farben in “echten Farbtönen” findet sich bei Björn Walther: https://www.bjoernwalther.com/farben-in-r-der-col-befehl/
Zu erwähnen ist noch die Farbe gray, die in den Abstufungen gray0 (=schwarz) bis gray99 (= weiß) knapp 99 Grauschattierungen ermöglicht. Dies ist bei schwarz-weiß Publikationen sehr hilfreich.
Soll es möglichst bunt sein, erzeugt die Funktion rainbow() die gewünschte Anzahl an Farben.
34.3 Liniendiagramm
Ein Liniendiagramm wird mit der Parametereinstellung type="l" erzeugt.
# Erzeuge ein paar Daten
x <-1:20
y1 <- x*x+1
# Erzeuge Liniendiagramm
plot(x, y1, type="l", pch=19, col="red", xlab="x", ylab="y")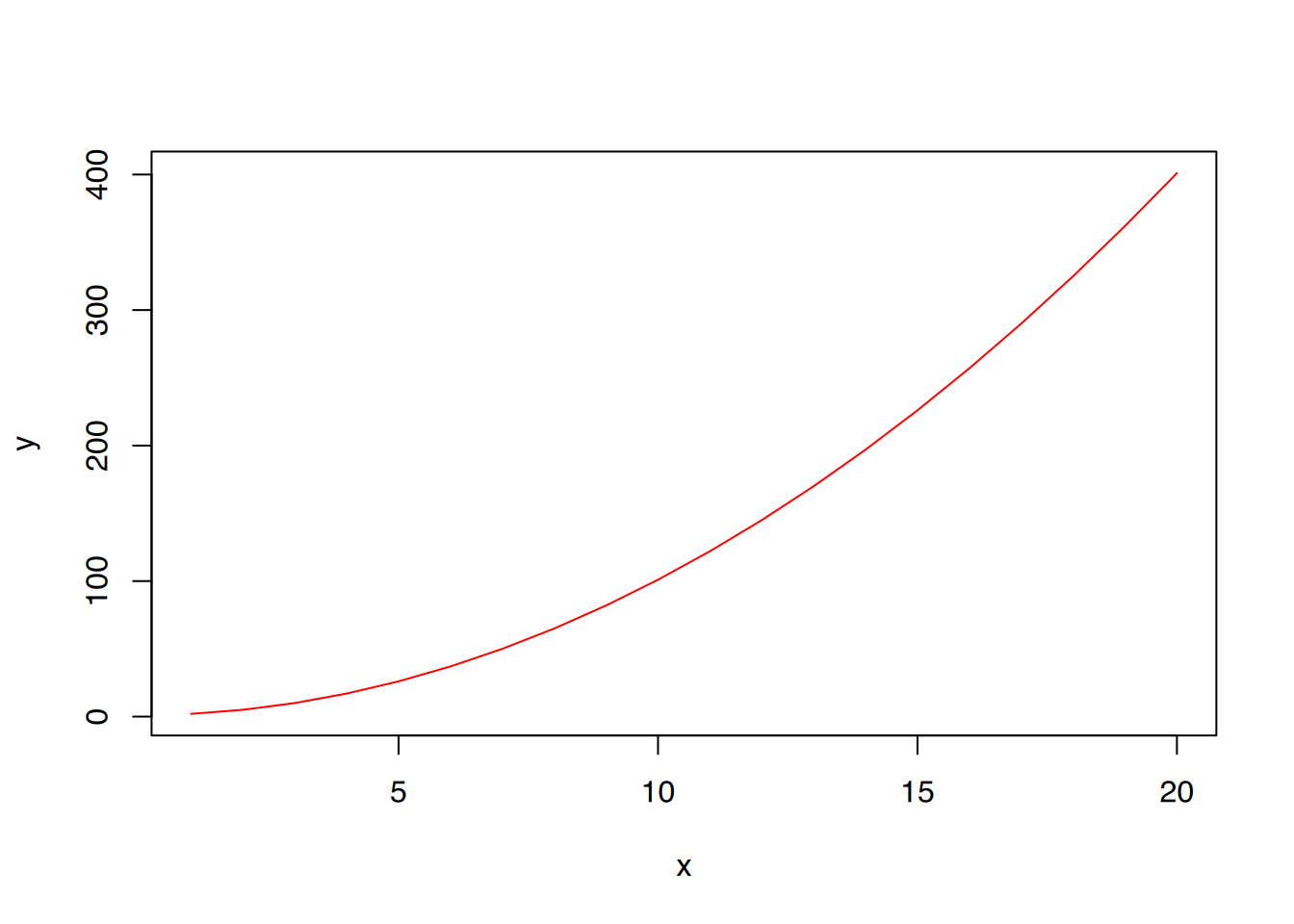
Mit type="b" (für both) wird sowohl Punkt- als auch Liniendiagramm erzeugt
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
Mit der Funktion lines() können weitere Linien hinzugefügt werden.
# erzeuge zweite Y-Reihe
y2 <- 2*y1
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
# Füge Linie hinzu
lines(x, y2, pch=18, col="blue", type="b", lty=2)
34.4 Histogram
Ein Histogram wird mit der Funktion hist() erstellt. Plotten wir ein Histogramm der Variable Alter im Beispieldatensatz pf8.
# lade Datensatz "pf8"
load(url("https://www.produnis.de/R/data/pf8.RData"))
# erstelle Histogramm von Variable "Alter" aus Datensatz "pf8"
hist(pf8$Alter)
Zum Vergleich:
# zum Vergleich plot()
plot(pf8$Alter, type="h")
Wie Sie sehen ist das plot()-Resultat nur entfernt dem Histogram ähnlich.
Die Klassengröße des Histograms kann mit dem Parameter breaks übergeben werden. Entweder schreiben wir die Anzahl der “Balken”, dann versucht R das “möglichst gut” auszuführen, oder wir übergeben einen Vektor mit den konkreten Breakpunkten.
# Histogramm mit "möglichst" 4 Balken
hist(pf8$Alter, breaks=4)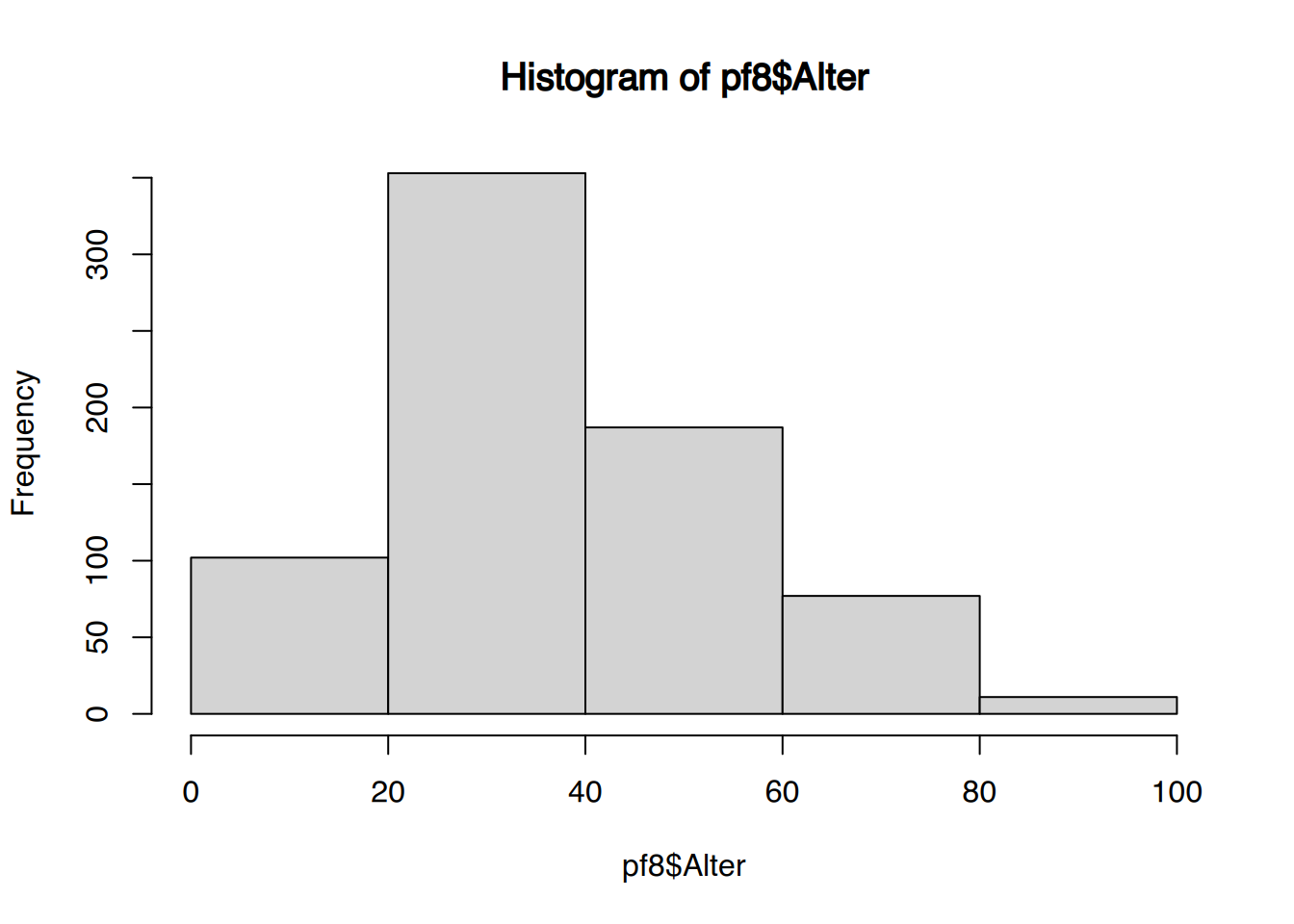
Wie Sie sehen stellt R dennoch 5 Balken dar.
Über den Parameter breaks können die Klassengrenzen manuell gesetzt werden.
hist(pf8$Alter, breaks=c(10, 30, 60, 90, 100))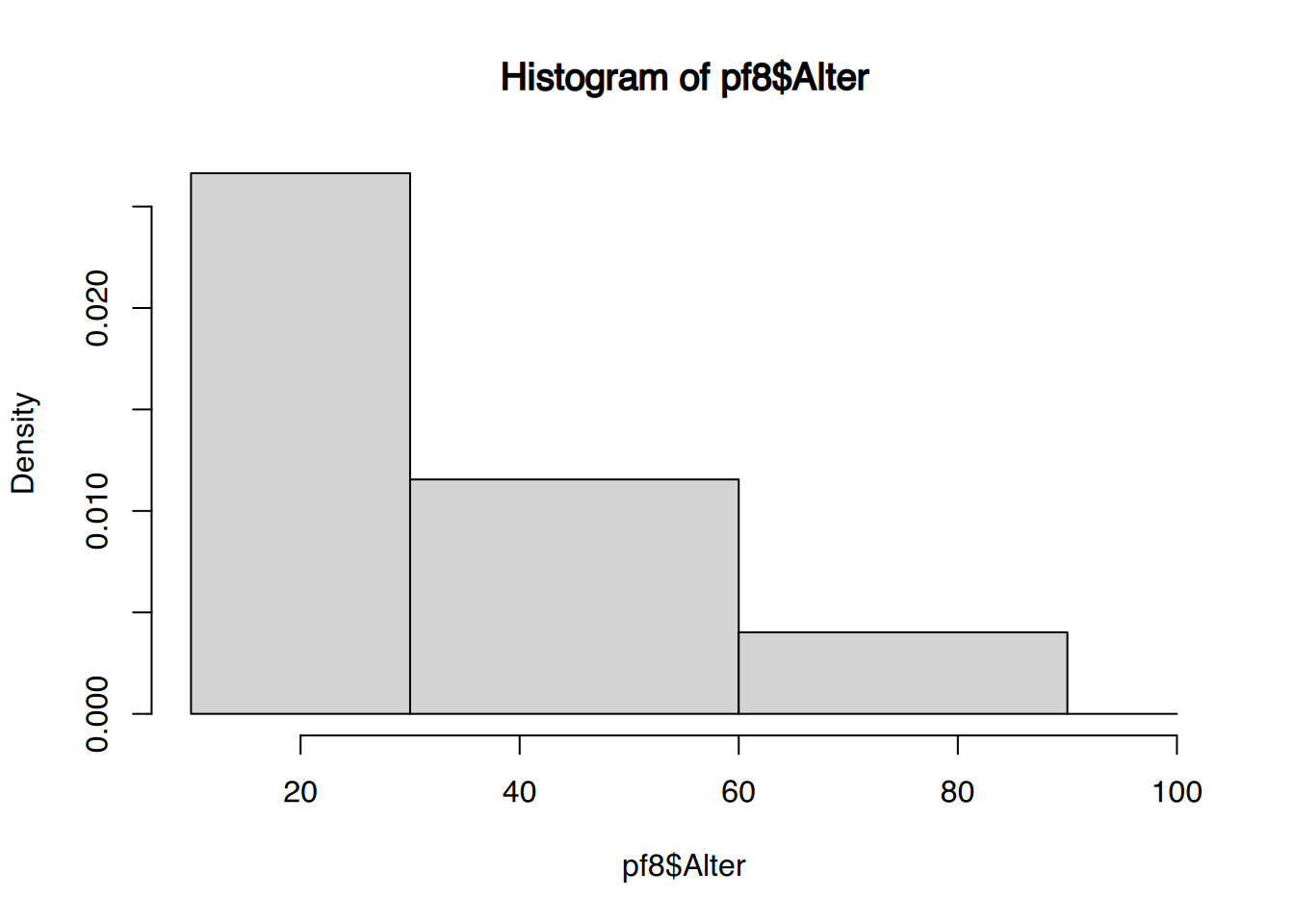
Über die generellen Parameter kann der Plot nun aufgehübscht werden.
# in schön
hist(pf8$Alter, breaks=4,
# Überschrift
main="Mein Histogramm",
# Unterschrift
sub="Abbildung x.y: Ein Histogramm",
# X-achse
xlab="Alter in Jahren",
# Y-Achse
ylab="Häufigkeit",
# blaue Balken
col="blue")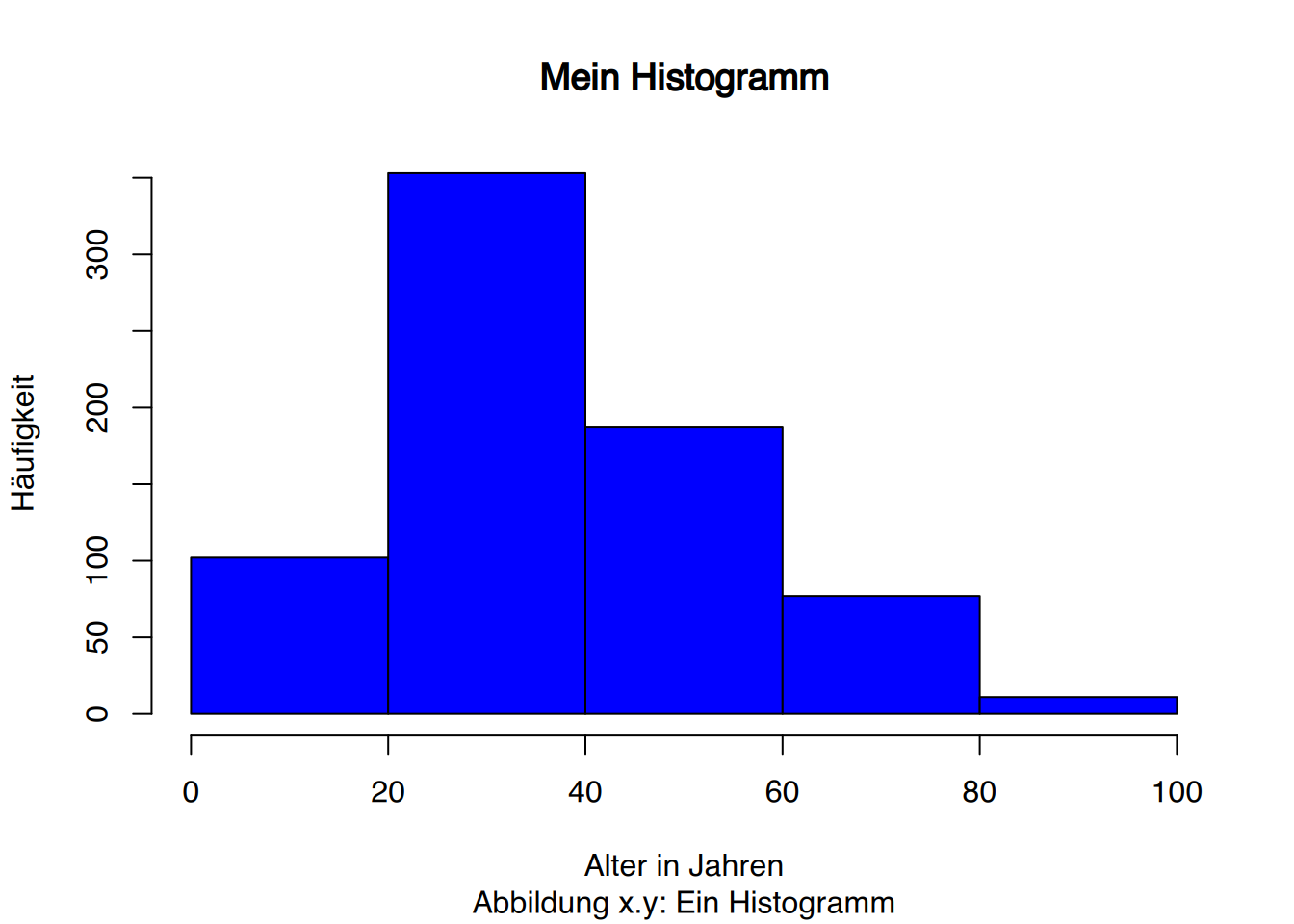
Mittels des Parameters freq=FALSE kann auf relative Häufigkeiten umgeschaltet werden.
# relative Häufigkeiten
hist(pf8$Alter, breaks=c(0, 20, 40, 60, 90), freq=FALSE)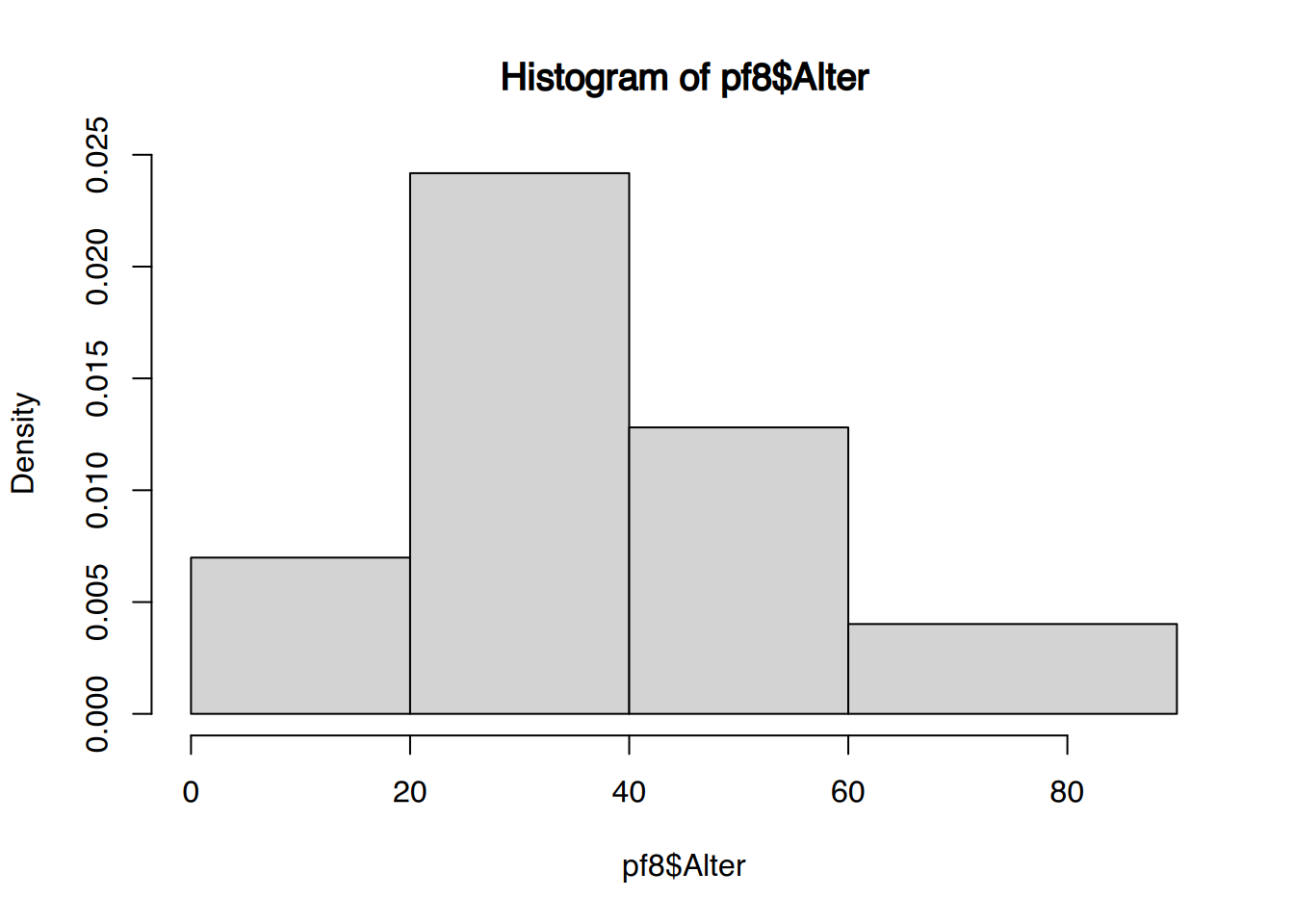
Für kumulierte Häufigkeiten muss das Histogramm zunächst in ein Objekt geschrieben werden.
# speichere in Objekt h
h <- hist(pf8$Alter, breaks=5, plot=FALSE)Nun können wir auf alle Werte des Histograms zugreifen. Wir ersetzen die gezählten Häufigkeiten (h$counts) mittels cumsum() durch kumulierte Werte.
# ersetze die Zellen durch kumulierte Häufigkeiten
h$counts <- cumsum(h$counts)
# plotte das kumulative Histogram
plot(h, col="hotpink", main = "kumulierte Häufigkeiten")
# füge Polygonzug hinzu
lines(c(h$breaks),c(0,h$counts), col="blue") # type="s" für `Steps`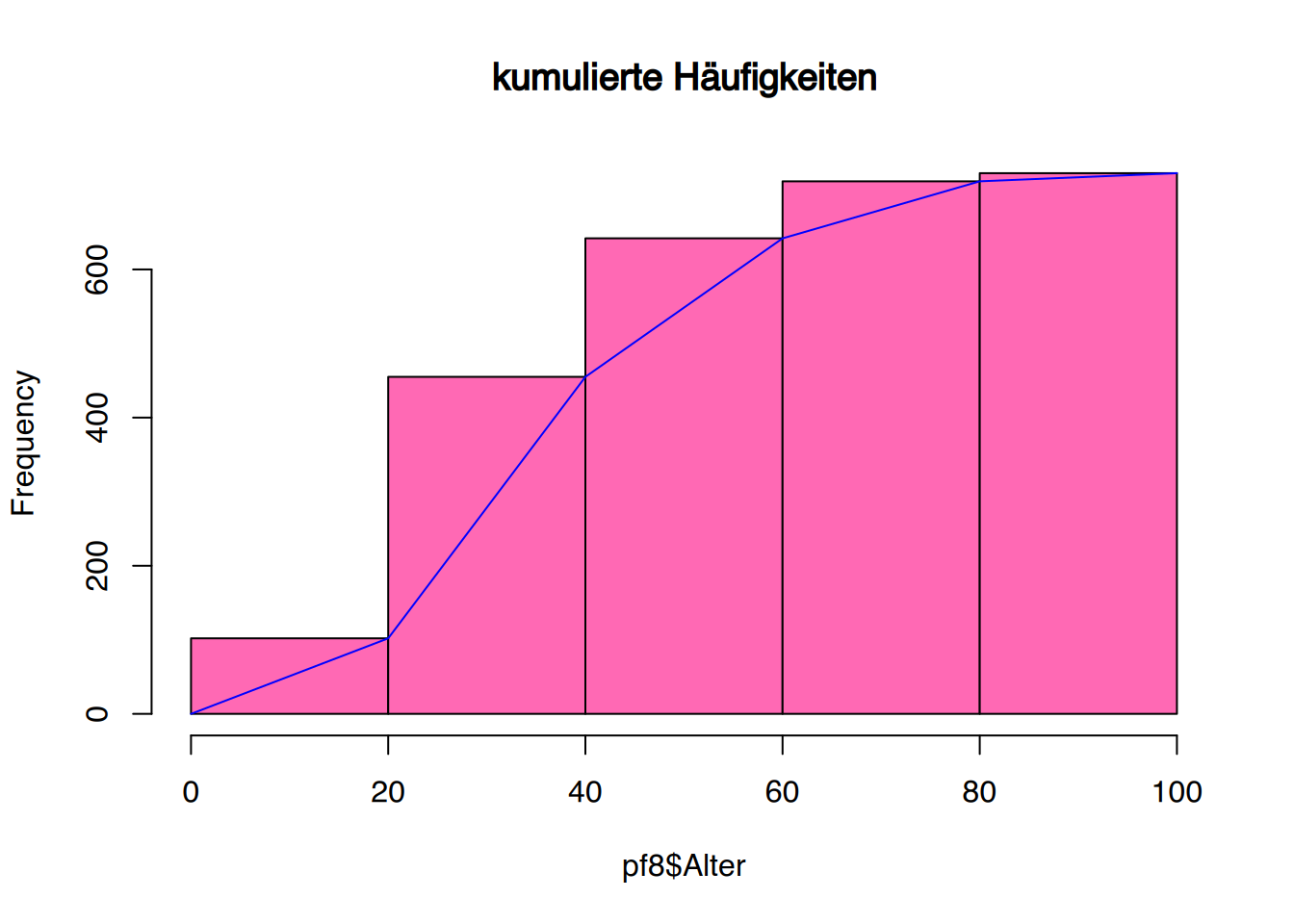
Bei den Polygonzügen können wir die X-Werte noch mittig setzen (h$mids), und den Linienstil auf “steps” ändern.
# plotte das kumulative Histogram
plot(h, col="hotpink", main = "kumulierte Häufigkeiten")
# füge Polygonzug hinzu
lines(c(0, h$mids),c(0,h$counts), col="blue", type="s")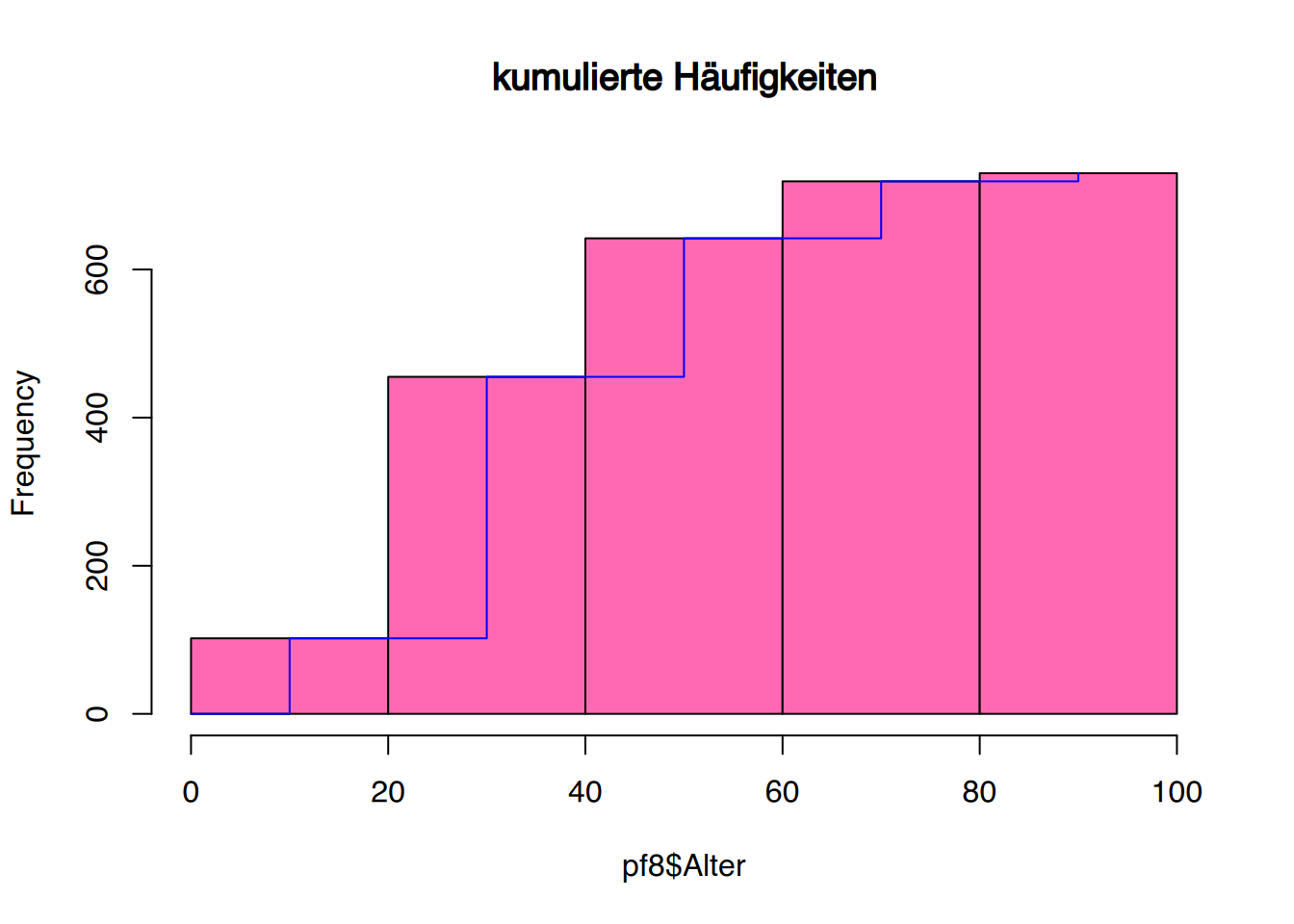
34.5 Kreisdiagramm
Ein Kreisdiagramm wird mit der Funktion pie() erzeugt. Die Funktion benötigt einen Vektor mit den Anteilsgrößen.
# Kreisdiagramm 60 / 40
pie(c(60,40))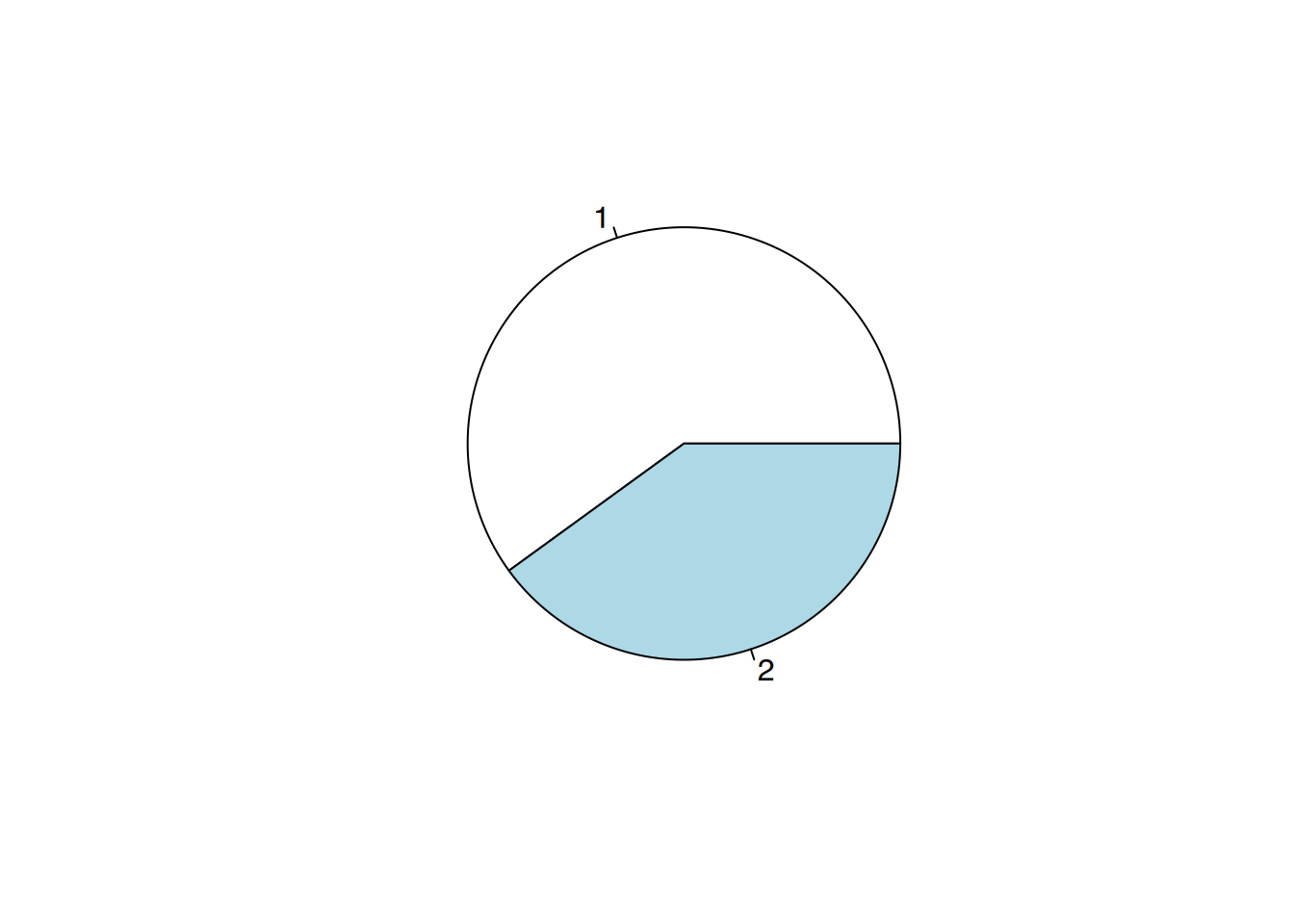
Soll z.B. ein Kreisdiagramm aus einem Faktor erzeugt werden, gelingt dies über die Integration der Funktion table().
# Kreisdiagramm von "Geschlecht" in "pf8"
pie(table(pf8$Geschlecht))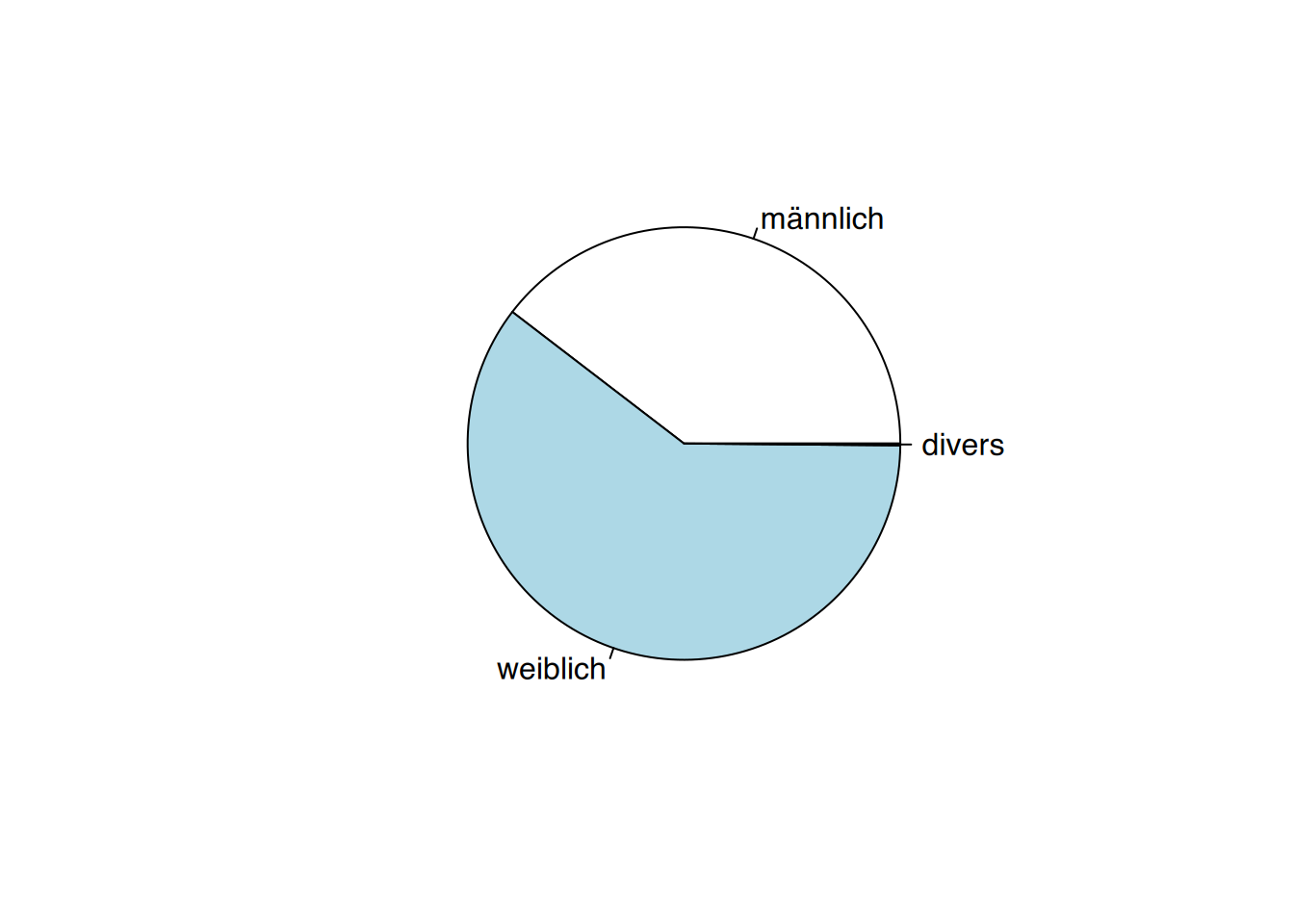
Mit den Parametern col, clockwise und labels können Sie weitere Anpassungen vornehmen.
pie(c(60,40), labels=c("ja", "nein"), col=c("azure", "coral"))
pie(table(pf8$Geschlecht), clockwise=TRUE, col=c("burlywood", "chartreuse","black"))
Und auch lustige Dinge kann man plotten1:
par(mar = c(0, 1, 0, 1))
pie(
c(280, 60, 20),
c('Sky', 'Sunny side of pyramid', 'Shady side of pyramid'),
col = c('#0292D8', '#F7EA39', '#C4B632'),
init.angle = -50, border = NA
)
34.6 Säulendiagramm
Säulendiagramme werden mit der Funktion barplot() erstellt. Sie benötigt einen Vektor mit den jeweiligen Ausprägungen der Säulen sowie optional die Namen der Säulen über den Parameter names.arg:
# Säulendiagramm
barplot(c(10,40,20), names.arg=c("Dritter", "Erster", "Zweiter"))
Liegen die Daten als Faktor vor, können sie mit Hilfe der Funktion table() übergeben werden.
# Säulendiagramm von "Standort" in "pf8"
barplot(table(pf8$Standort))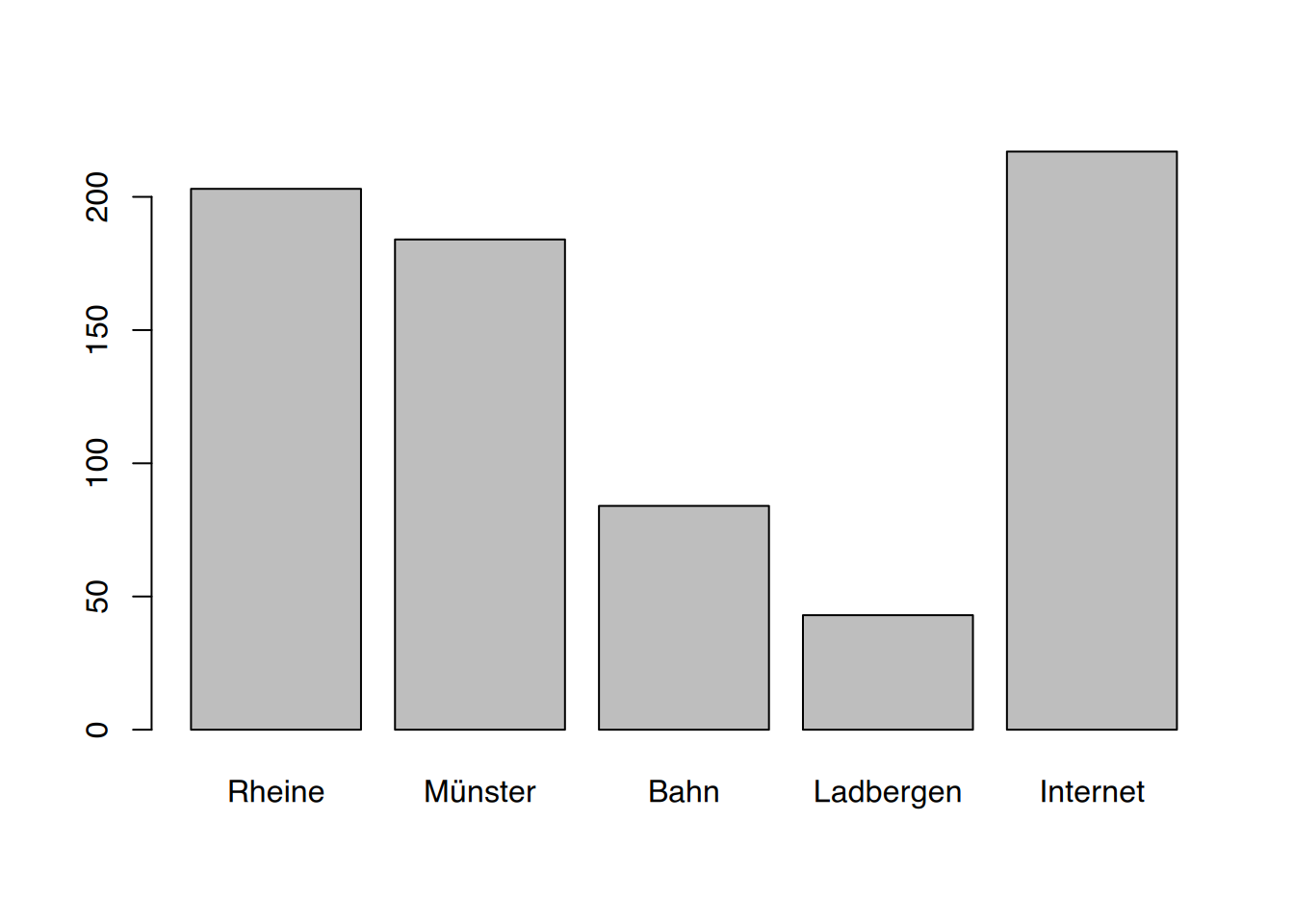
Liegen die Daten als wide table vor (z.B als Matrix mit bedeutsamen rownames), so wie unsere Matrix der Pflegeberufe, erstellt barplot() gruppierte Säulen. Über das Argument legend.text können die Reihennamen übergeben werden.
# Säulendiagramm der Matrix Pflegeberufe
barplot(Pflegeberufe, beside=T, col=rainbow(5), legend.text = rownames(Pflegeberufe))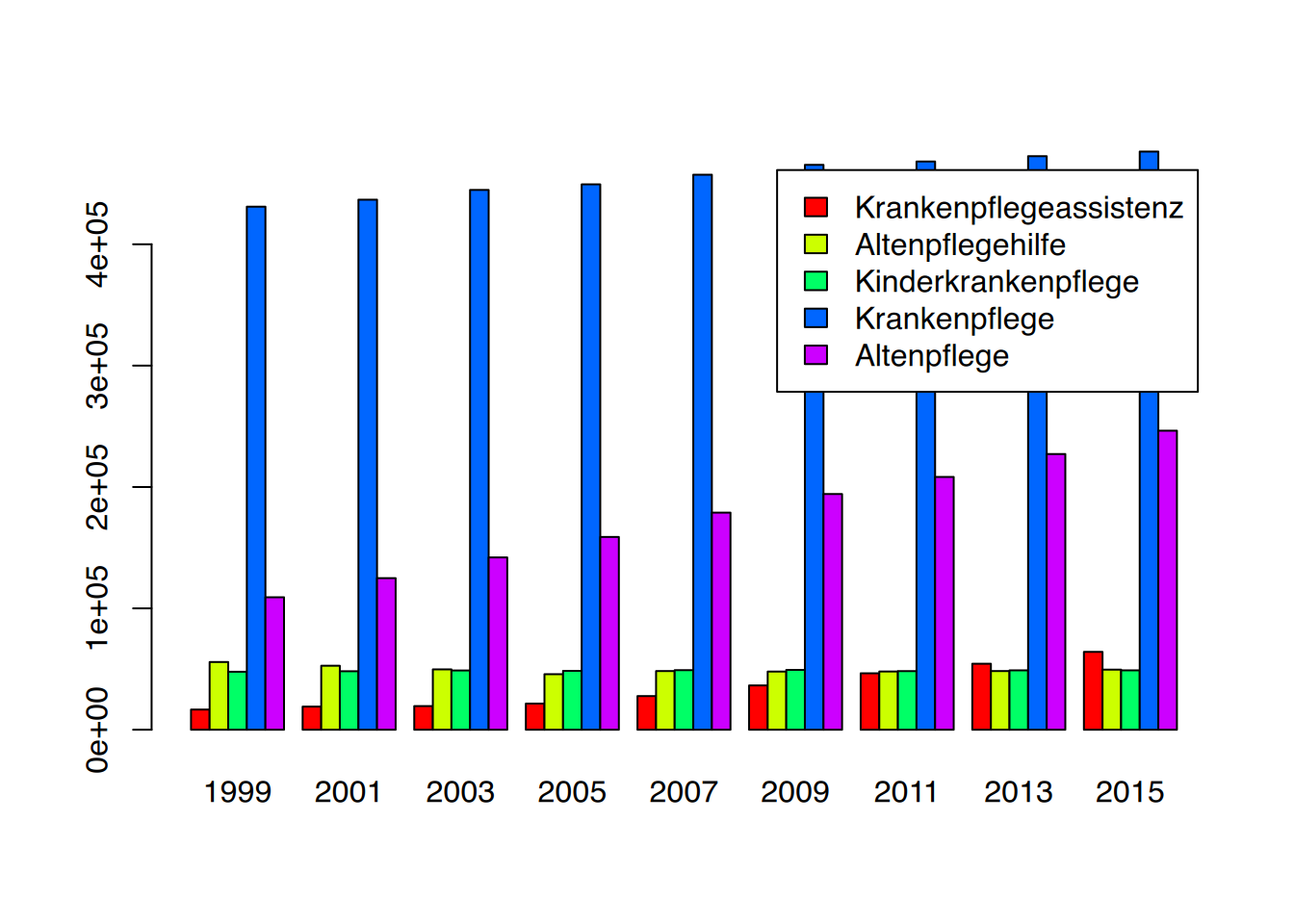
Wird der Parameter beside weggelassen (entspricht FALSE), werden die Reihen in einer gemeinsamen Säule zusammengefasst.
# Säulendiagramm der Matrix Pflegeberufe
barplot(Pflegeberufe, col=rainbow(5), legend.text = rownames(Pflegeberufe))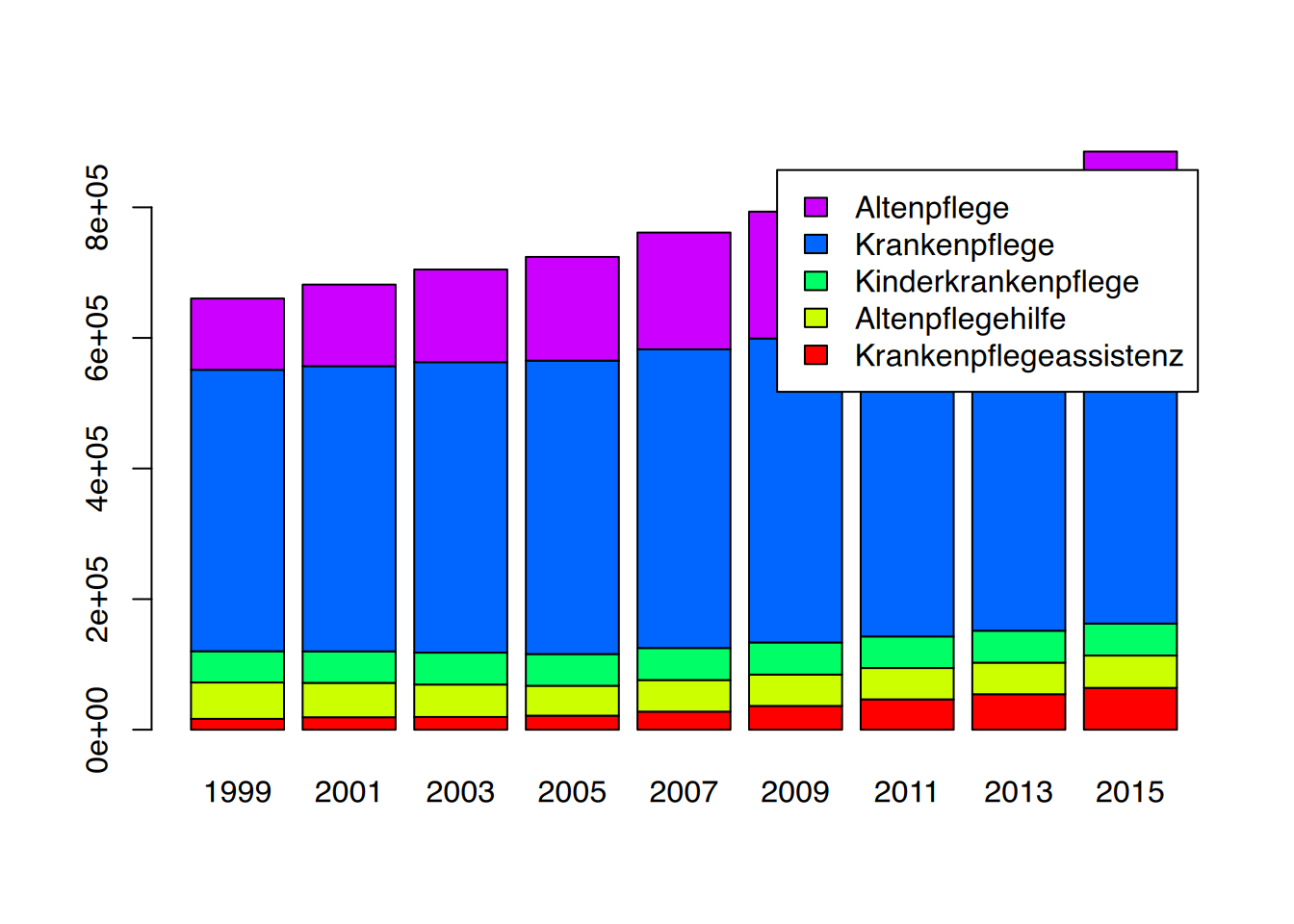
Das entspricht vom Prinzip her der Abbildung Abbildung 8.1.
34.7 Balkendiagramm
Balkendiagramme werden genau so erstellt wie Säulendiagramme, nämlich über die Funktion barplot() mit dem Parameter horiz=TRUE.
# Balkendiagramm der Matrix Pflegeberufe
# diesmal Schwarz-weiss
barplot(Pflegeberufe,
# schalte um auf "Balkendiagramm"
horiz=TRUE,
# Graustufen führende 0 kann weggelassen werden
col=gray(c(.1, .2, .3, .4, .5)),
legend.text = rownames(Pflegeberufe))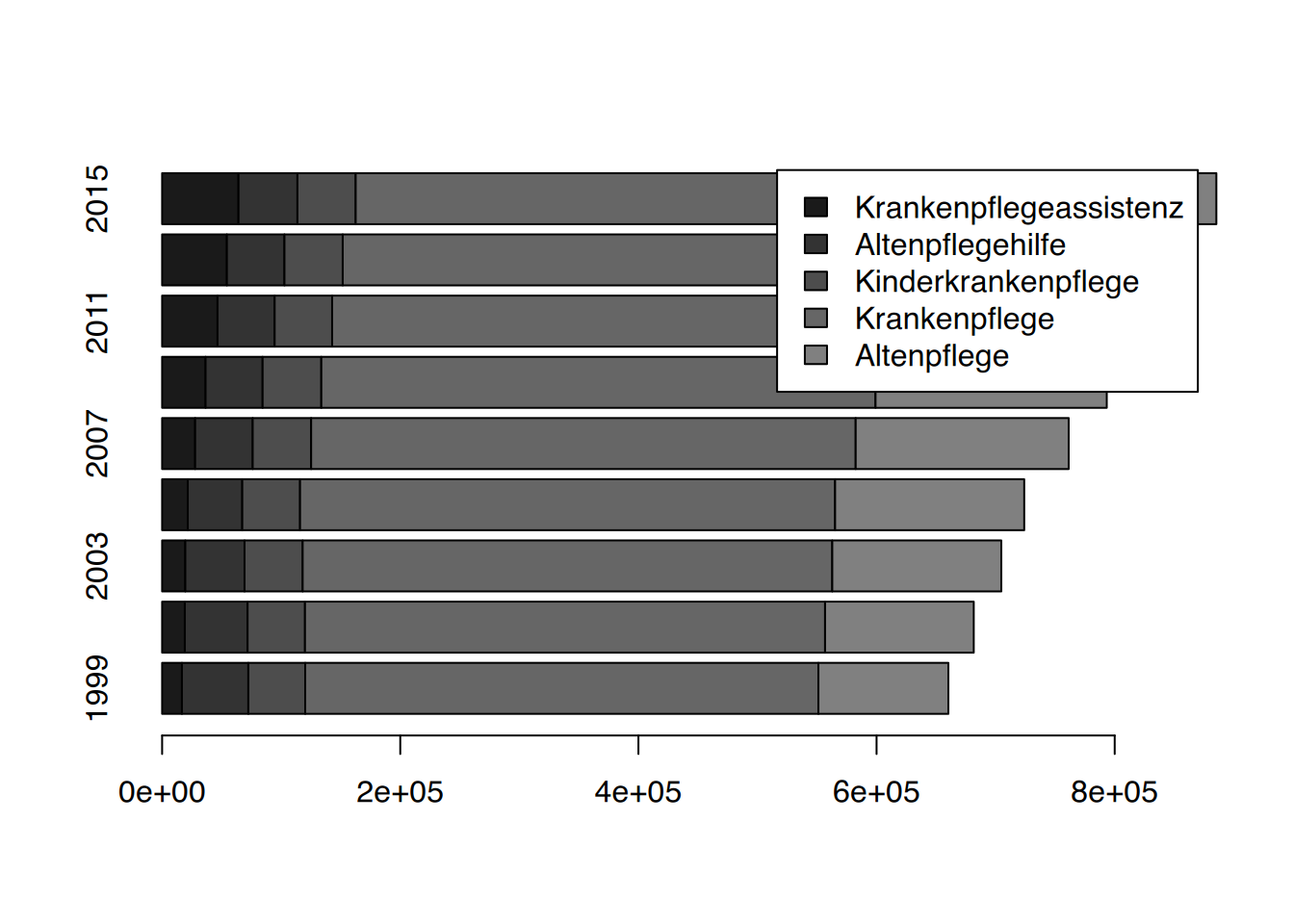
# Balkendiagramm "category" aus "mma"
# diesmal Schwarz-weiss
barplot(table(mma$Kategorie),
horiz=TRUE,
col=rainbow(7),
# Legende aus Faktorenlevels
legend.text = levels(mma$Kategorie))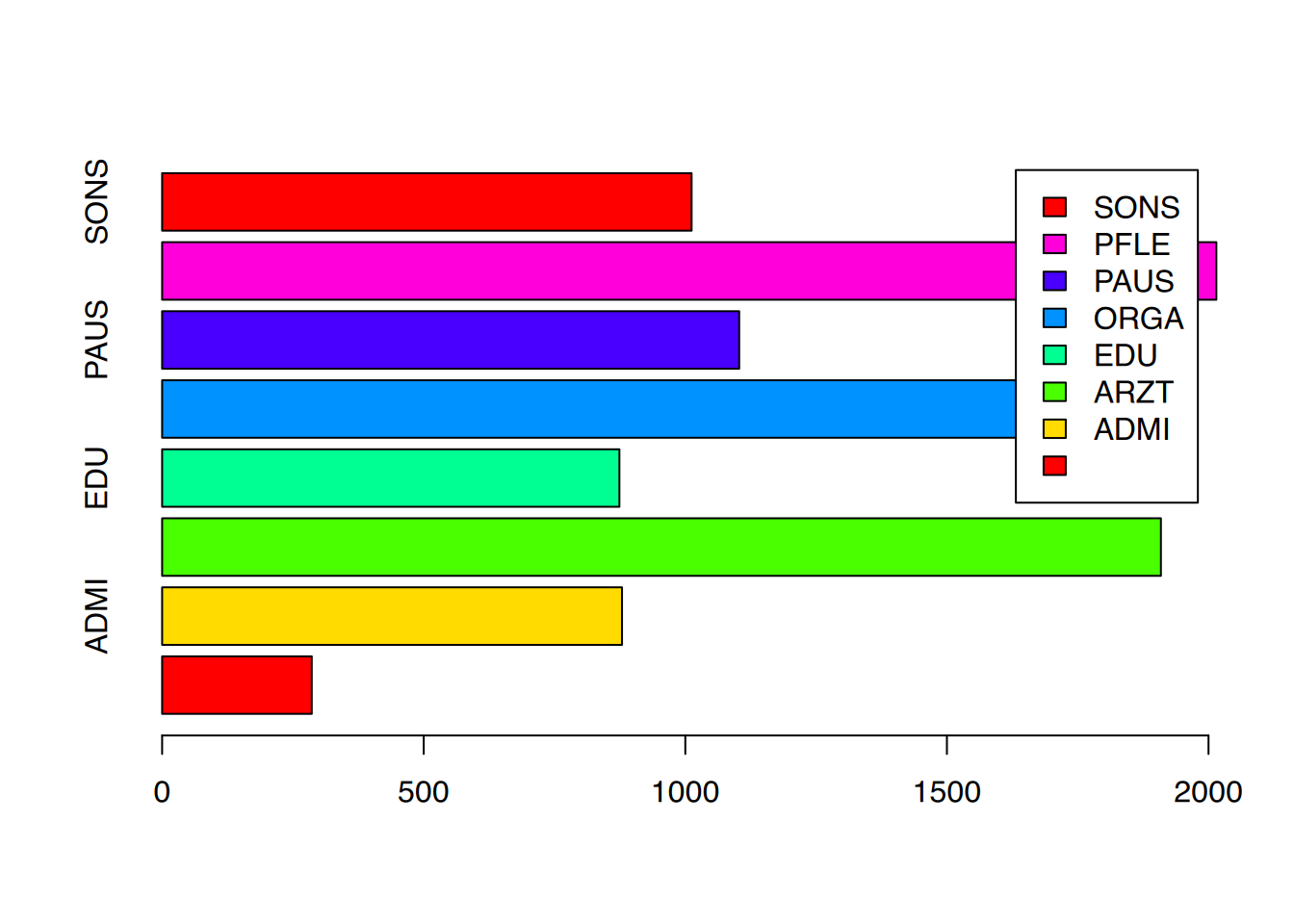
34.8 Boxplot
Boxplots können mit der Funktion boxplot() erstellt werden.
# lade Datensatz
load(url("https://www.produnis.de/R/data/nw.RData"))
# Boxplot des Alters in Datensatz "nw"
boxplot(nw$age)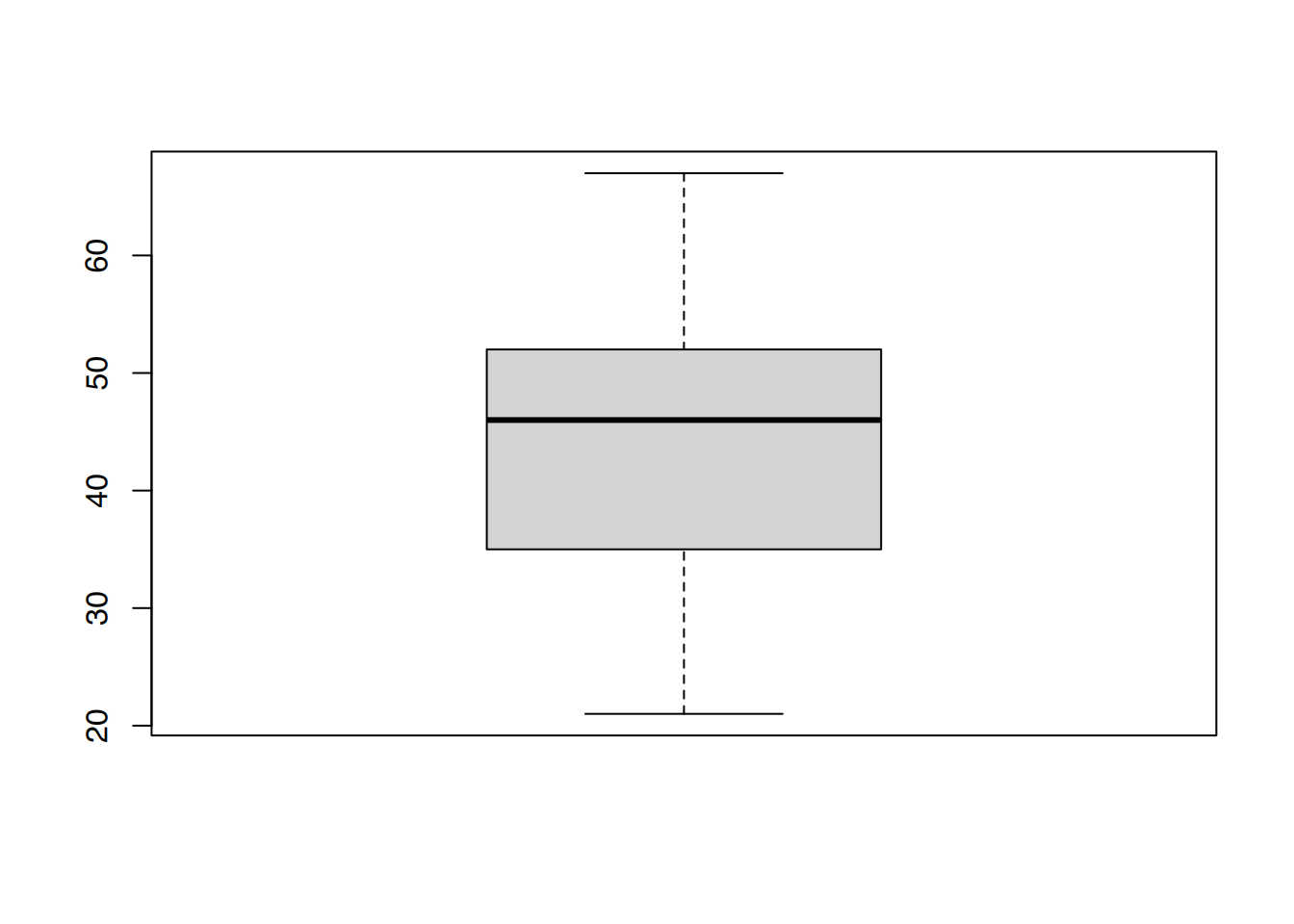
Die Funktion erkennt auch die Syntax “erklärt durch”.
# Boxplot des Alters in Datensatz "nw" erklärt durch "sex"
boxplot(age ~ sex, data=nw)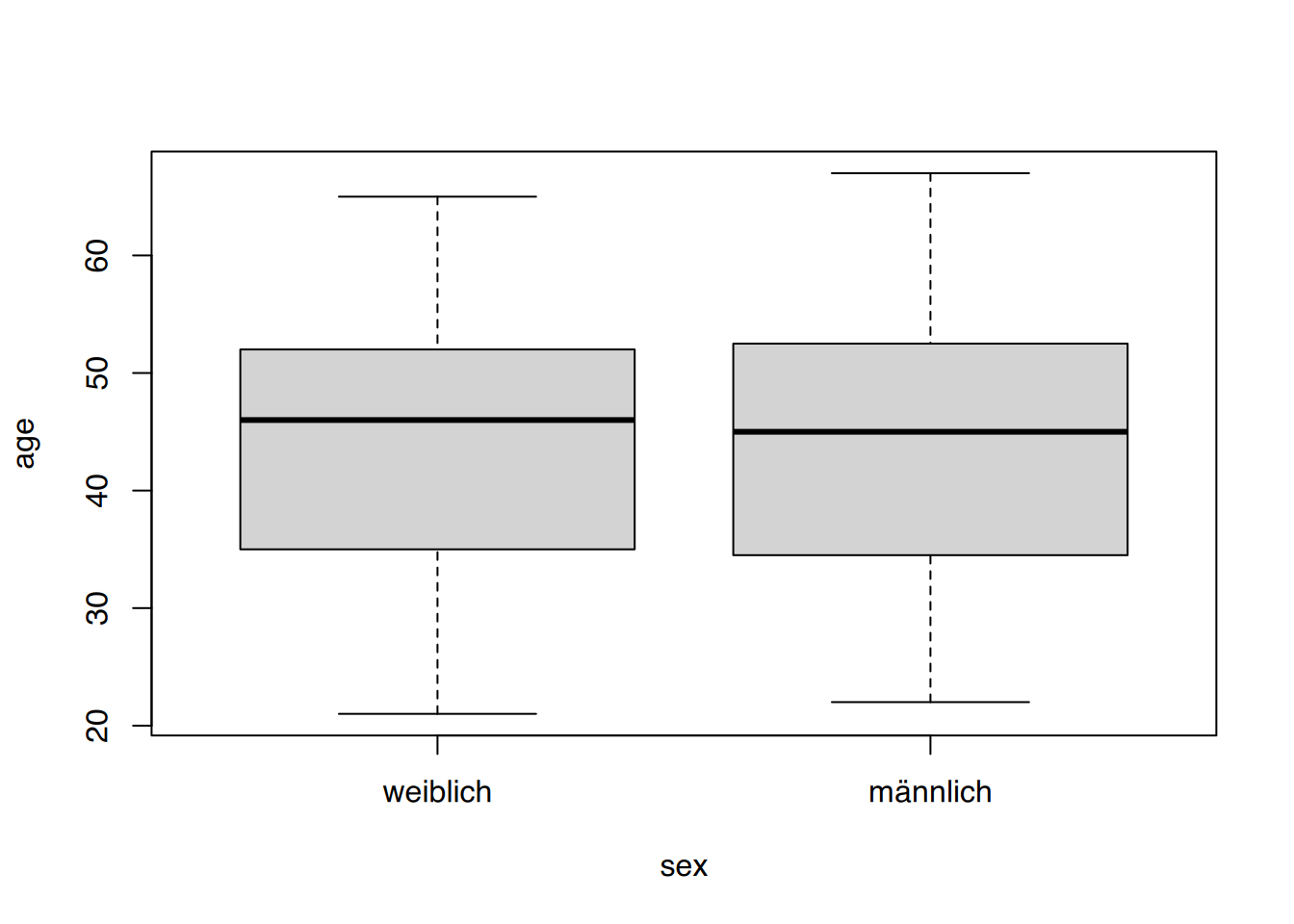
Über die Parameter frame, border, col und notch können weitere Anpassungen erfolgen.
boxplot(age ~ sex, data=nw, frame = FALSE, border="blue", horizontal = TRUE)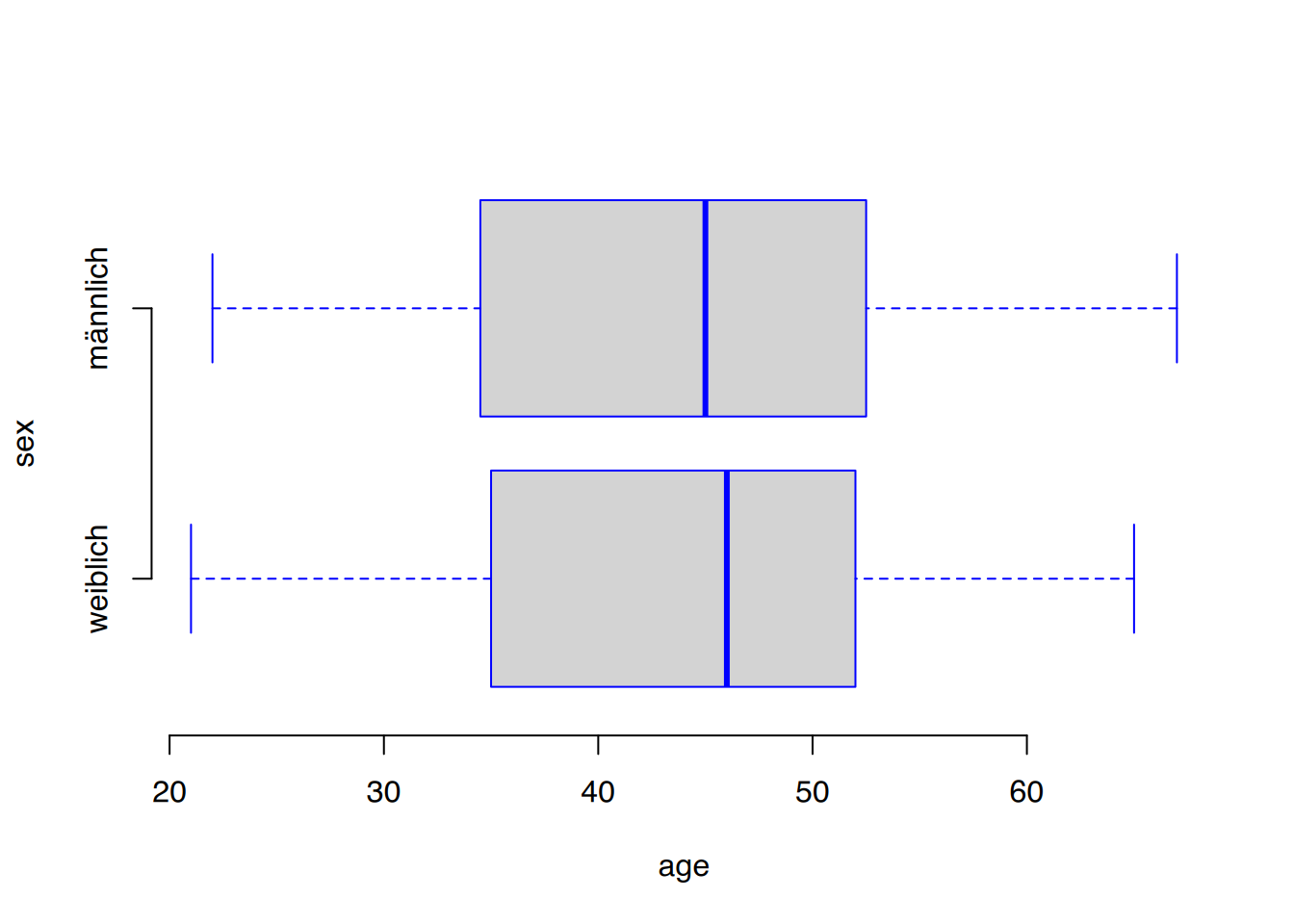
boxplot(age ~ sex, data=nw, frame = FALSE, col=c("green", "red"), notch = TRUE)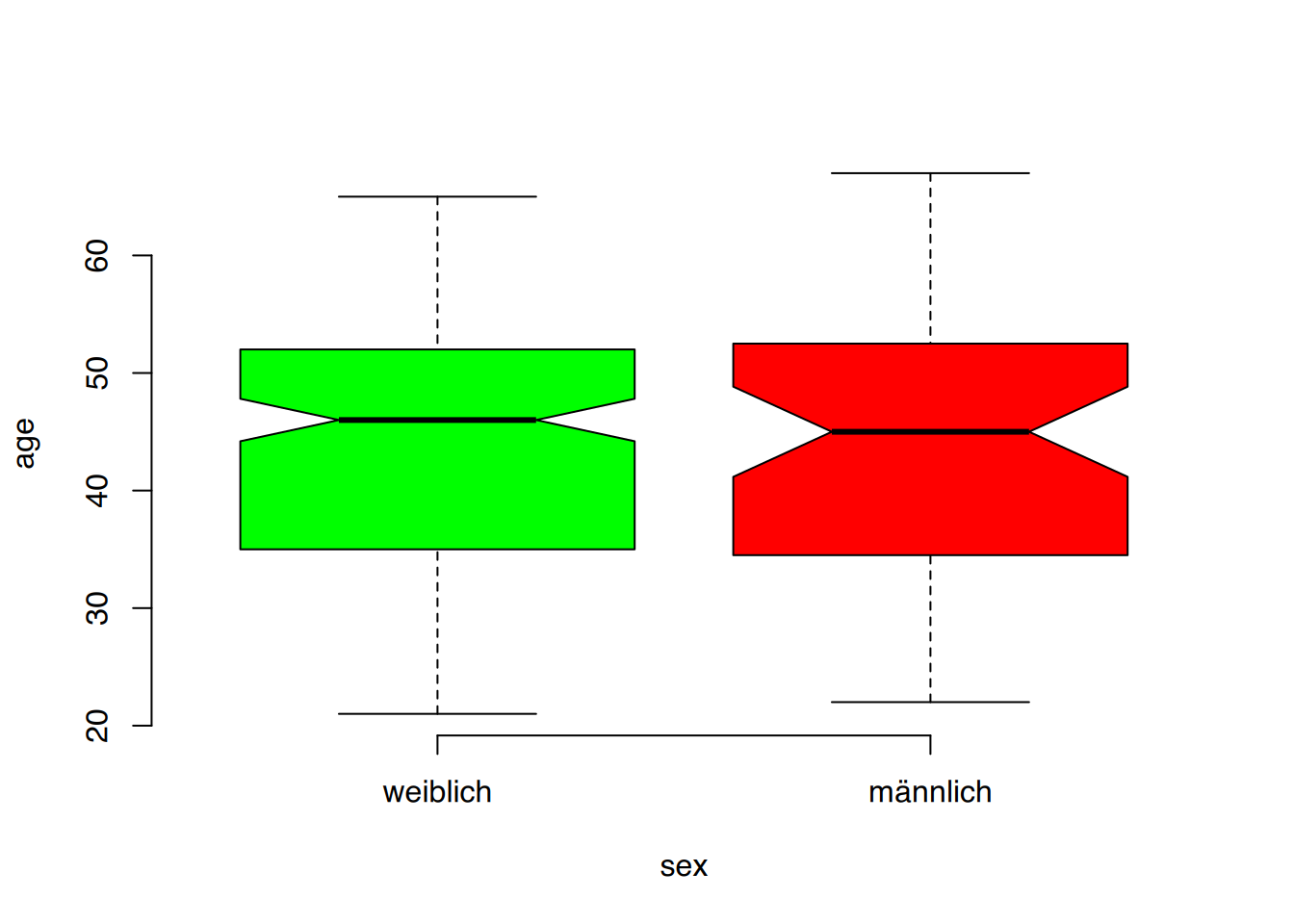
Weitere innere Gruppierungen können mit einem Sternchen * übergeben werden.
boxplot(age~sex*timework, data=nw, col=c("blue", "skyblue"), notch = TRUE)## Warning in (function (z, notch = FALSE, width = NULL, varwidth = FALSE, :
## einige notches liegen außerhalb der hinges ('box'): evtl. notch=FALSE setzen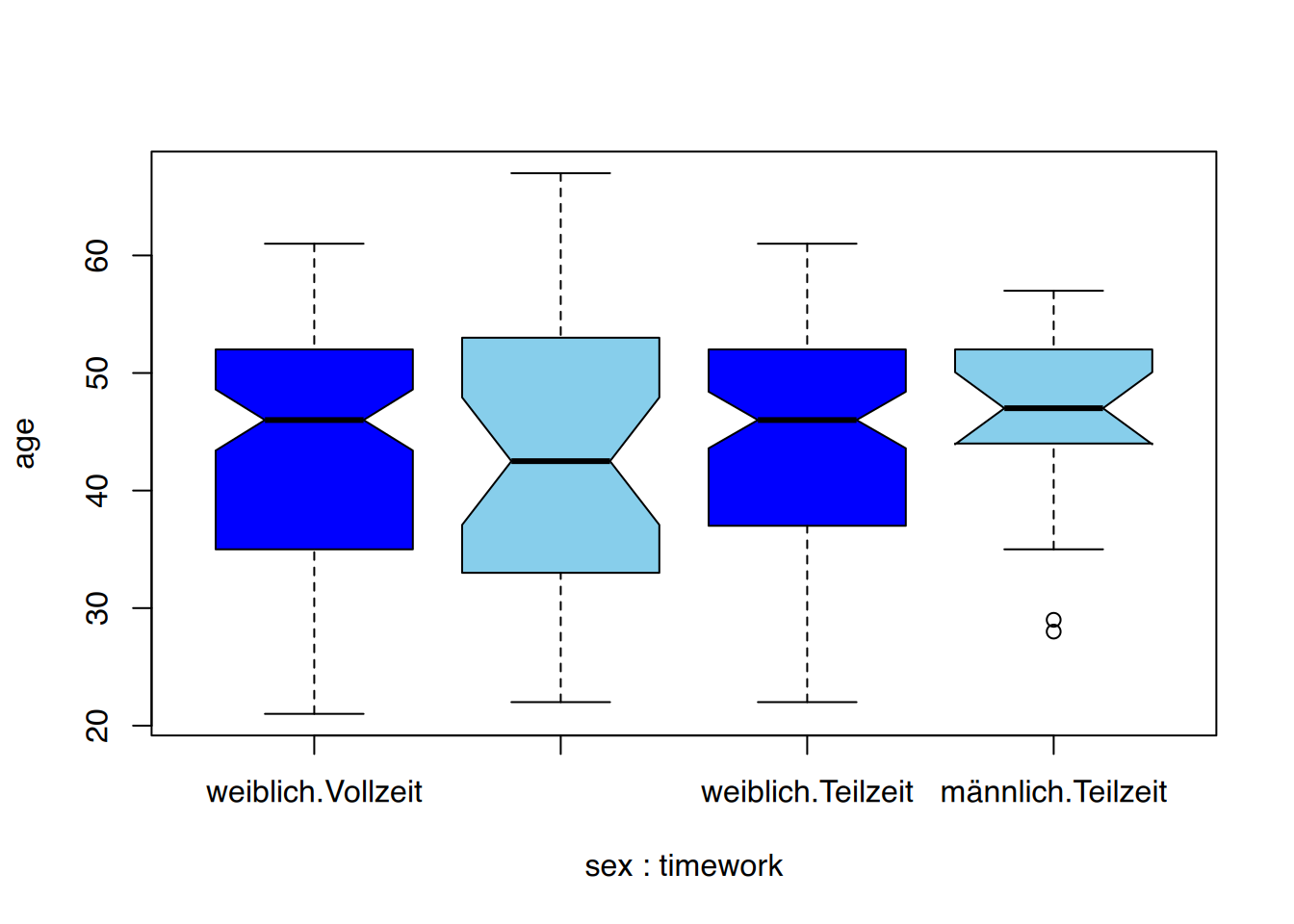
34.9 Polygone
Mit der Funktion polygon() können Polygone hinzugefügt werden. Ein Polygon wird so gezogen, als würden wir es mit einem Stift auf Papier zeichnen, ohne den Stift dabei abzusetzen. Das heisst, wir müssen “hin und zurück” zeichnen.
# erzeuge Testwerte
x <- sort(rnorm(100, mean=12, sd=2))
y <- sort(rnorm(100, mean=140, sd=15))
# Mittelwer als x-Koordinate
mx <- mean(x)
# Berechne Schnittpunkt mit Regressionsgeraden
ymx <- coef(lm(y~x))[1] + mx*coef(lm(y~x))[2]
plot(x,y, col="blue",
ylab="Körpergröße", xlab="Alter",
main="Ein Plot mit R base",
sub="Dieses Plot wurde aus Zufallswerten erstellt",
pch=13)
abline(lm(y~x), col="red")
# Füge Mittelwertlinie hinzu
lines(x=c(mx, mx), y=c(0, ymx), lty=3)
# füge Polygon hinzu
polygon(x=c(x, max(x), min(x)), y=c(y, 0, 0), col="snow2")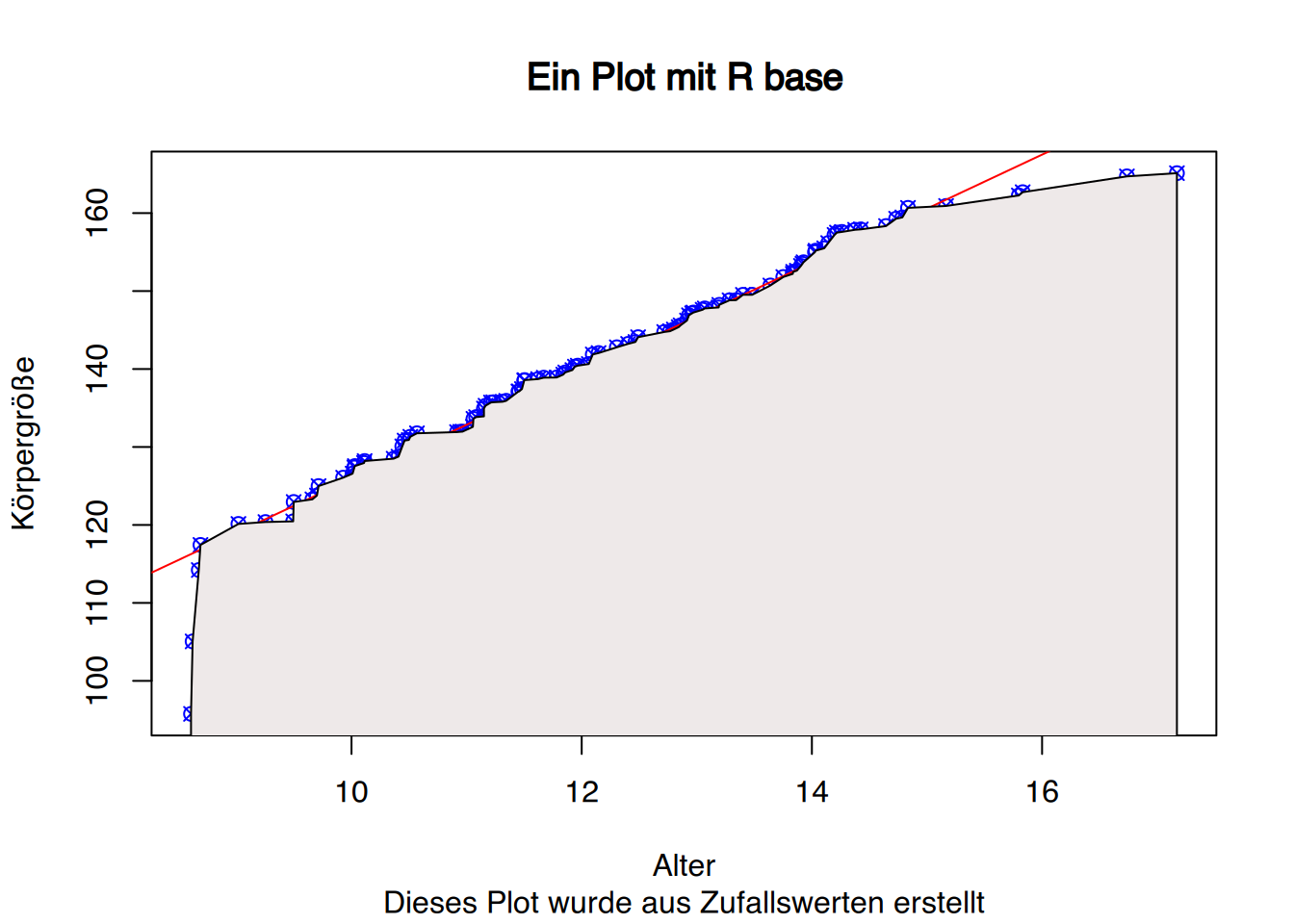
So kann ein Diagramm der Standardnormalverteilung erstellt werden, wobei beliebige Bereiche hervorgehoben werden können.
# erzeuge 10.000 Zufallswerte der Standardnormalverteilung
x <- sort(rnorm(10000))
# erzeuge die passenden Dichterwerte
y <- dnorm(x)
# beginne mit Plot
plot(x, y, ylab="Dichtefunktion", xlab="x", main="Die Standardnormalverteilung",
sub="Bereich von 1 Standardabweichung")
# füge gefülltes Polygon hinzu
polygon(x=c(x, max(x), min(x)), y=c(y, 0,0 ), col="snow2")
# füge 1 Standardabweichung hinzu
lines(x=c(-1, -1), y=c(0, dnorm(-1)), lty=3)
lines(x=c(1, 1), y=c(0, dnorm(1)), lty=3)
# füge Polygon für 1. Standardbweichung hinzu
xsd1 <- x[x<1 & x>-1]
ysd1 <- dnorm(xsd1)
polygon(x=c(xsd1, max(xsd1), min(xsd1)), y=c(ysd1, 0, 0 ), col="green") 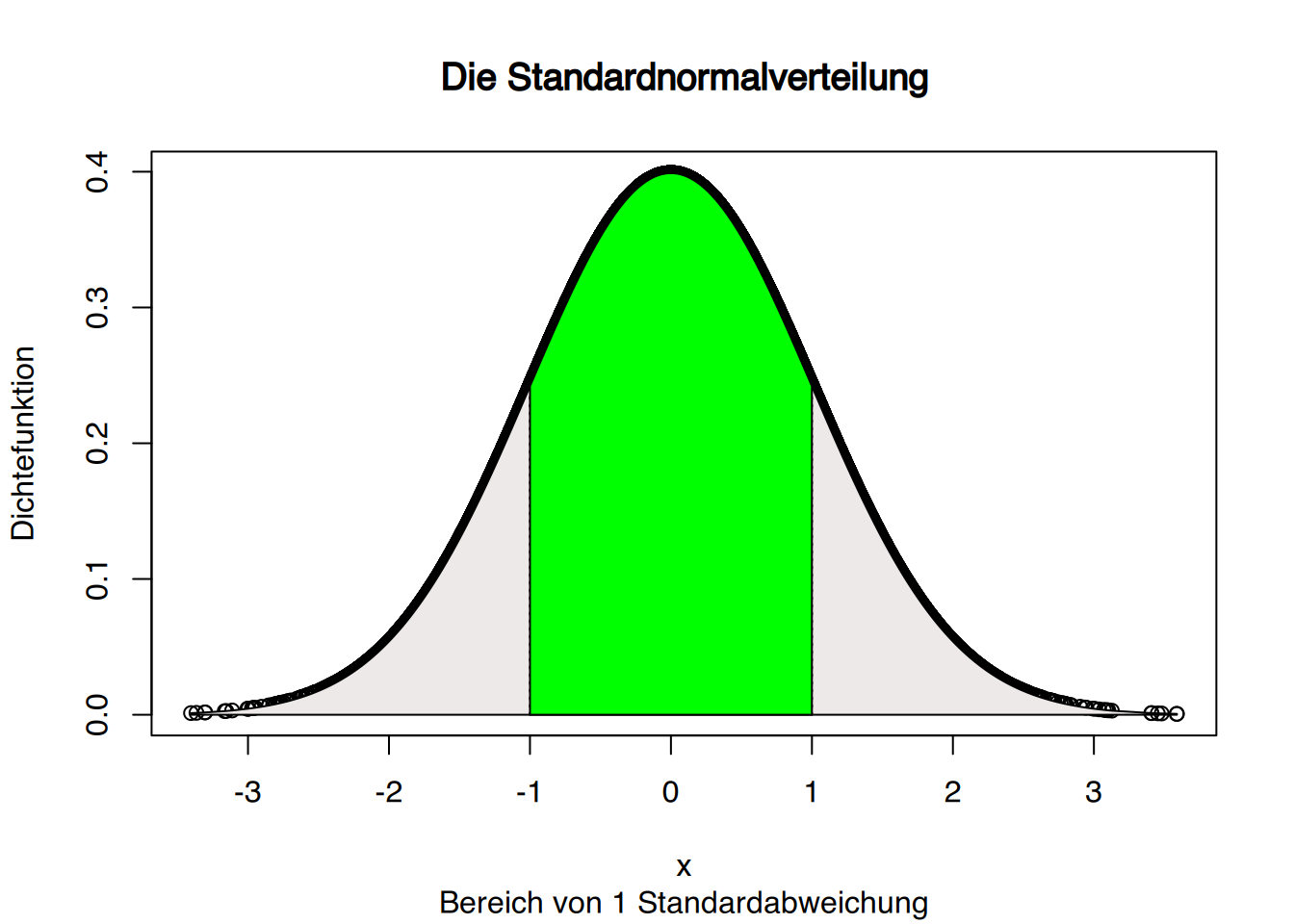
Der Bereich von zwei Standardabweichungen kann wie folgt hinzugefügt werden. Dies “übermalt” jedoch die grüne Fläche.
plot(x, y, ylab="Dichtefunktion", xlab="x", main="Die Standardnormalverteilung",
sub="Bereich von 2 Standardabweichungen")
# füge 2. Standardabweichung hinzu
lines(x=c(-2, -2), y=c(0, dnorm(-2)), lty=6)
lines(x=c(2, 2), y=c(0, dnorm(2)), lty=6)
# füge Polygon für 2. Standardabweichung
# dies übermalt das 1. Polygon
xsd2 <- x[x<2 & x>-2]
ysd2 <- dnorm(xsd2)
polygon(x=c(xsd2, max(xsd2), min(xsd2)), y=c(ysd2, 0, 0 ), col="blue") 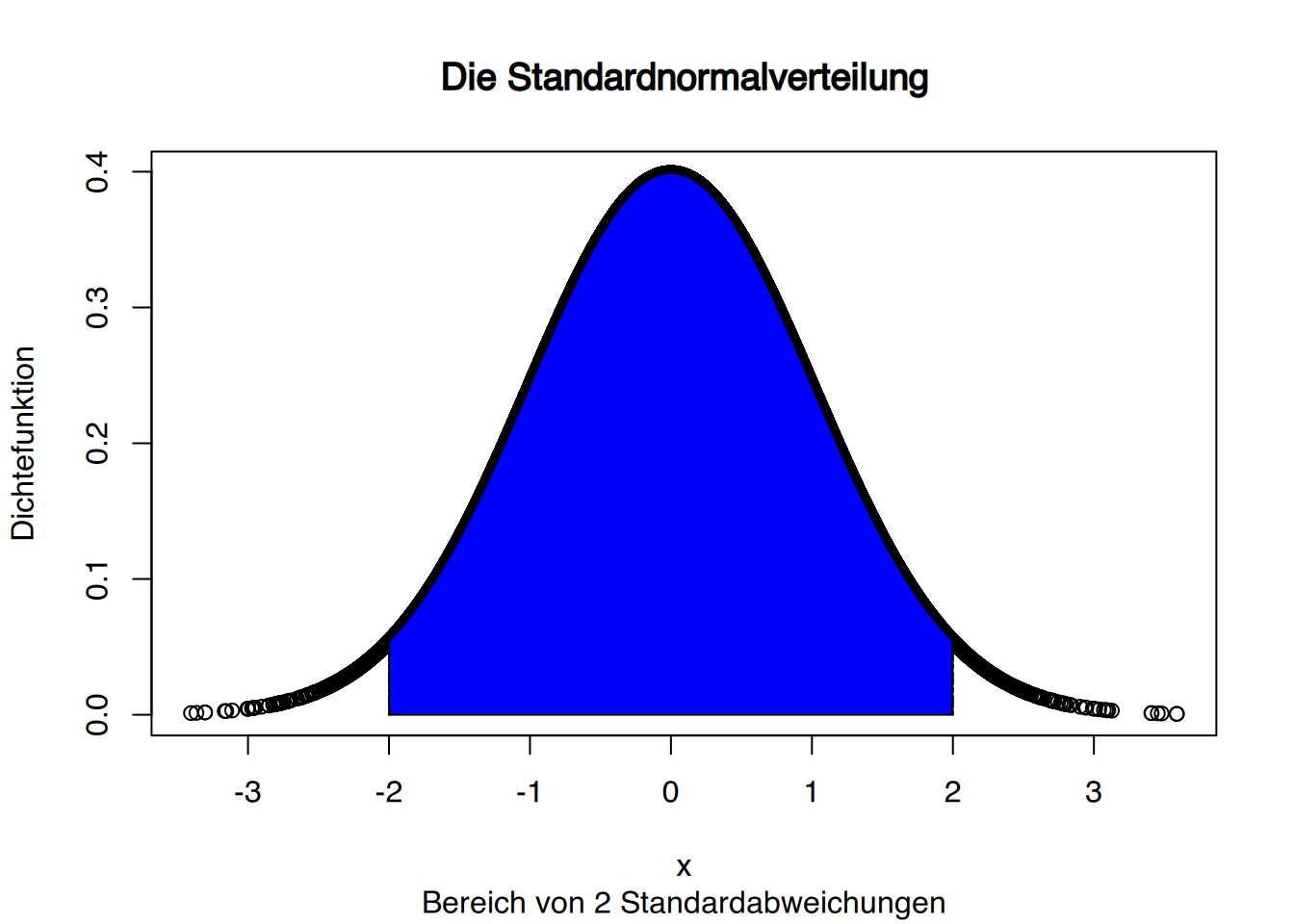
In Abschnitt 37.6 werden Polygone verwendet, um eine Übersichtsgrafik der BMI-Klassen in Abhängigkeit von Körpergröße und Körpergewicht zu erstellen.
34.10 QQ-Plots
Zur Überprüfung, ob eine Variable normalverteilt ist, werden häufig Quantil-Quantil-Diagramme (QQ-Plots) erzeugt. In R lassen sich diese mit den Funktionen qqnorm() und qqline() erstellen.
# QQ-Plot für "Alter" aus dem Datensatz "epa"
qqnorm(epa$age, col = "steelblue")
qqline(epa$age, col = "purple", lwd = 2)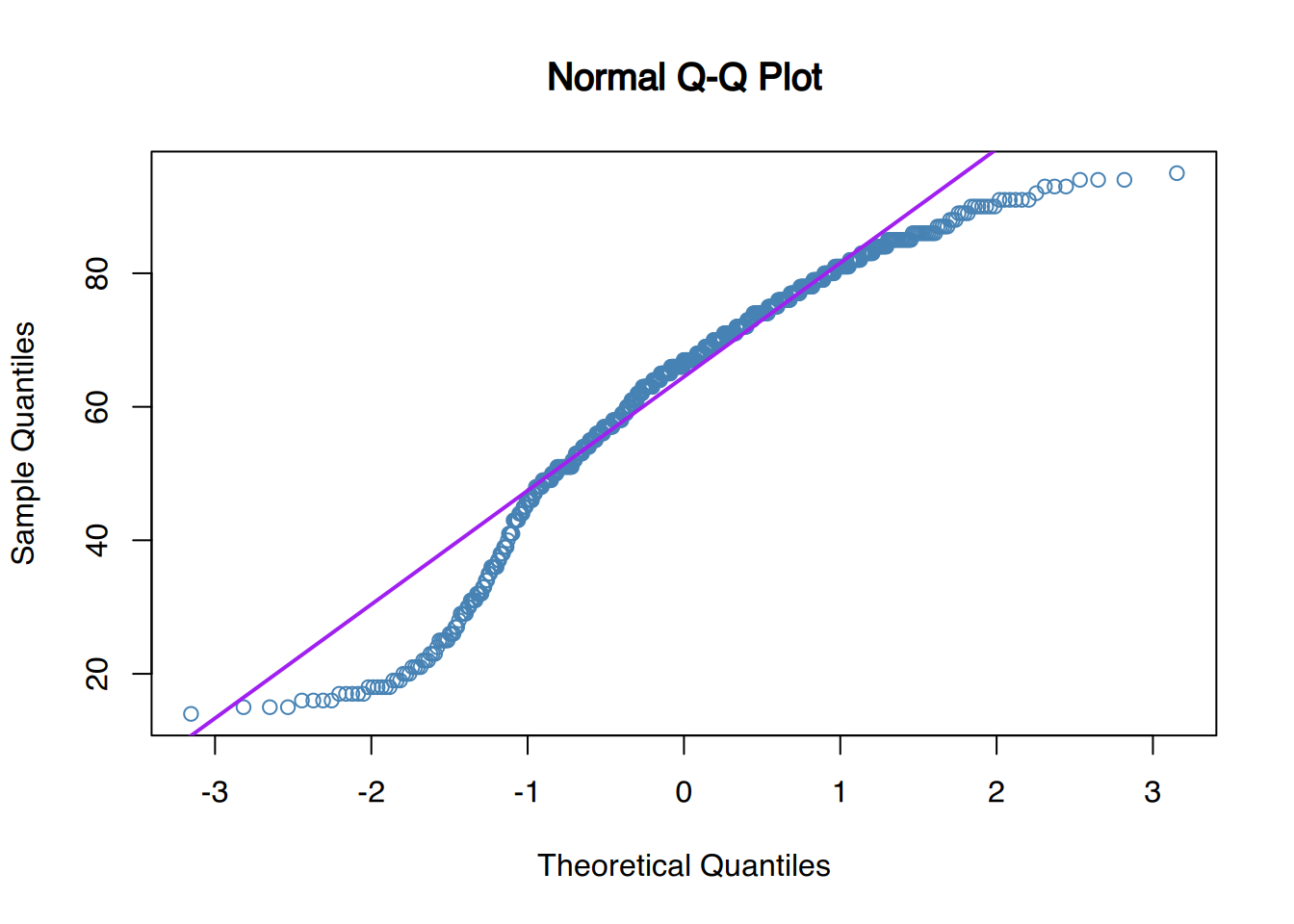
Zusätzlich steht die abgewandelte Funktion qqPlot() aus dem car-Paket zur Verfügung. Dieses zeichnet standardmäßig das Konfidenzintervall mit ein, und kann neben der Normalverteilung auch anderen Verteilungsformen prüfen.
car::qqPlot(epa$age, col="darkblue",
col.lines = "red")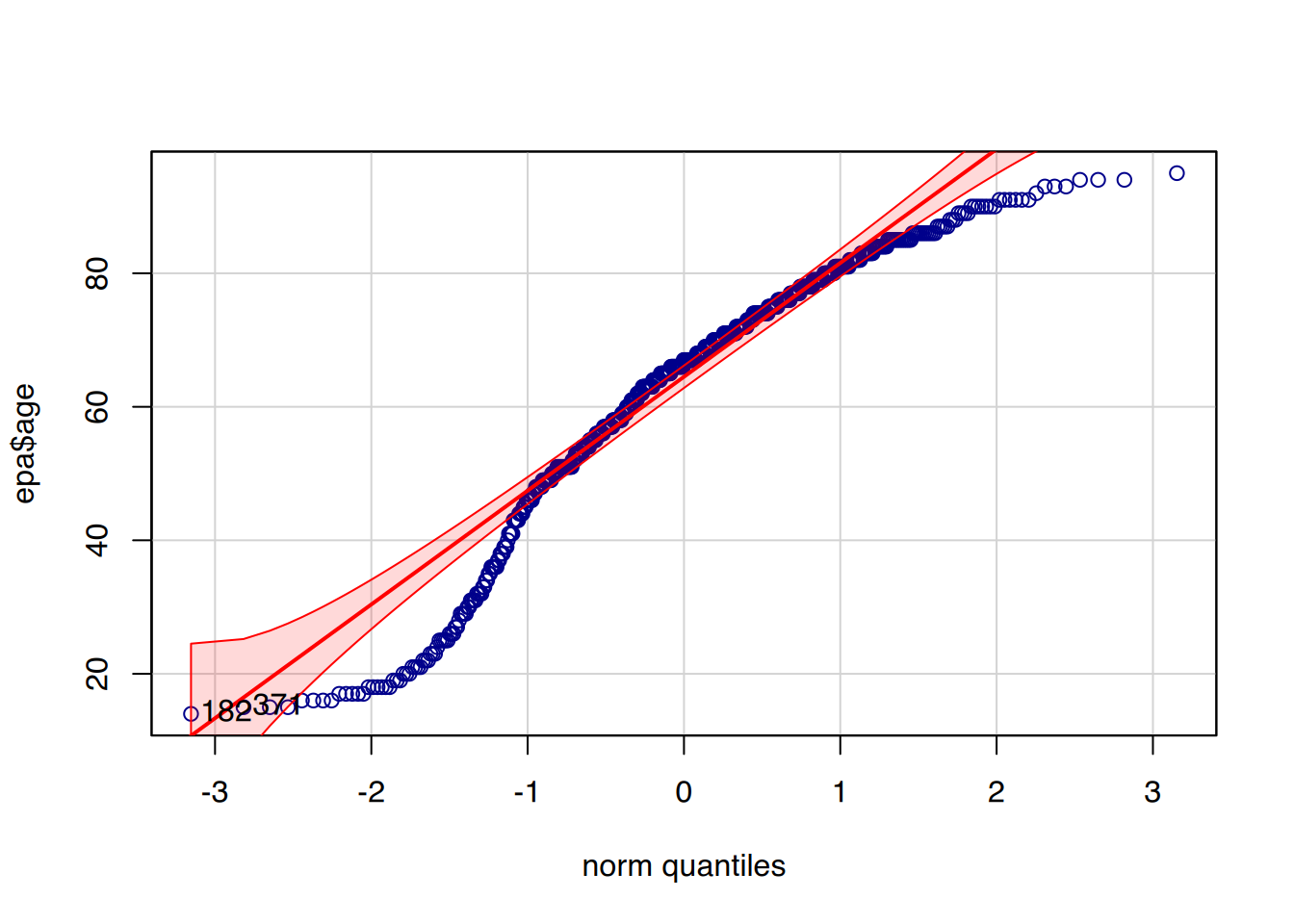
## [1] 182 37134.11 Legendenbox
Mit der Funktion legend() kann dem Plot eine Legendenbox hinzugefügt werden. Die ersten beiden Parameter legen dabei die X- und Y-Achsenpositionen der Box fest.
# Erzeuge ein paar Daten
x <-1:20
y1 <- x*x+1
y2 <- 2*y1
#
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
# Füge Linie hinzu
lines(x, y2, pch=18, col="blue", type="b", lty=2)
# Füge Legendenbox hinzu
legend(1, 95, legend=c("Männer", "Frauen"),
col=c("red", "blue"), lty=1:2, cex=0.8)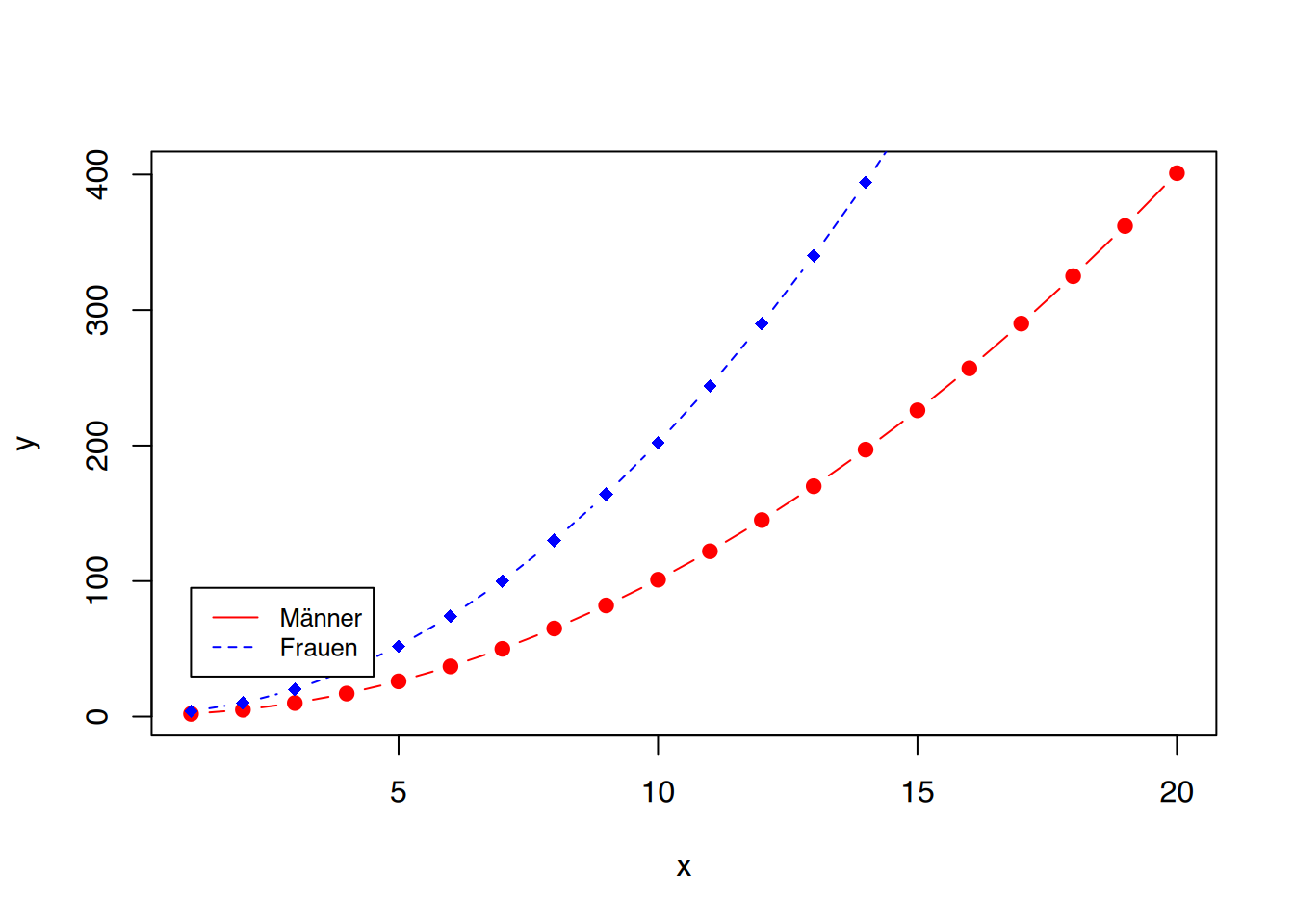
Zusätzlich nimmt legend() folgende Parameter entgegen:
title- Titel der Legendenboxtext.font- ein Integer der das Aussehen festlegt:1= normal2= fett3= kursiv4= fett und kursiv
bg- die Hintergrundfarbe der Legendenbox
box.lty- die Strichart des Legenrahmensbox.lwd- die Strichstärke des Rahmensbox.col- die Farbe des Rahmenshoriz- Box horizontal ausrichten (TRUE/FALSE)legend- Variablennamen der Objektelty- Strichart der Variablencol/fill- Füll- und Strichfarben der Variablen
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
lines(x, y2, pch=18, col="blue", type="b", lty=2)
# Füge Legendenbox hinzu
legend(1, 95, legend=c("Männer", "Frauen"),
col=c("red", "blue"), lty=1:2, cex=0.8,
bg="lightgreen", box.lty = 4, box.lwd = 2, box.col = "purple")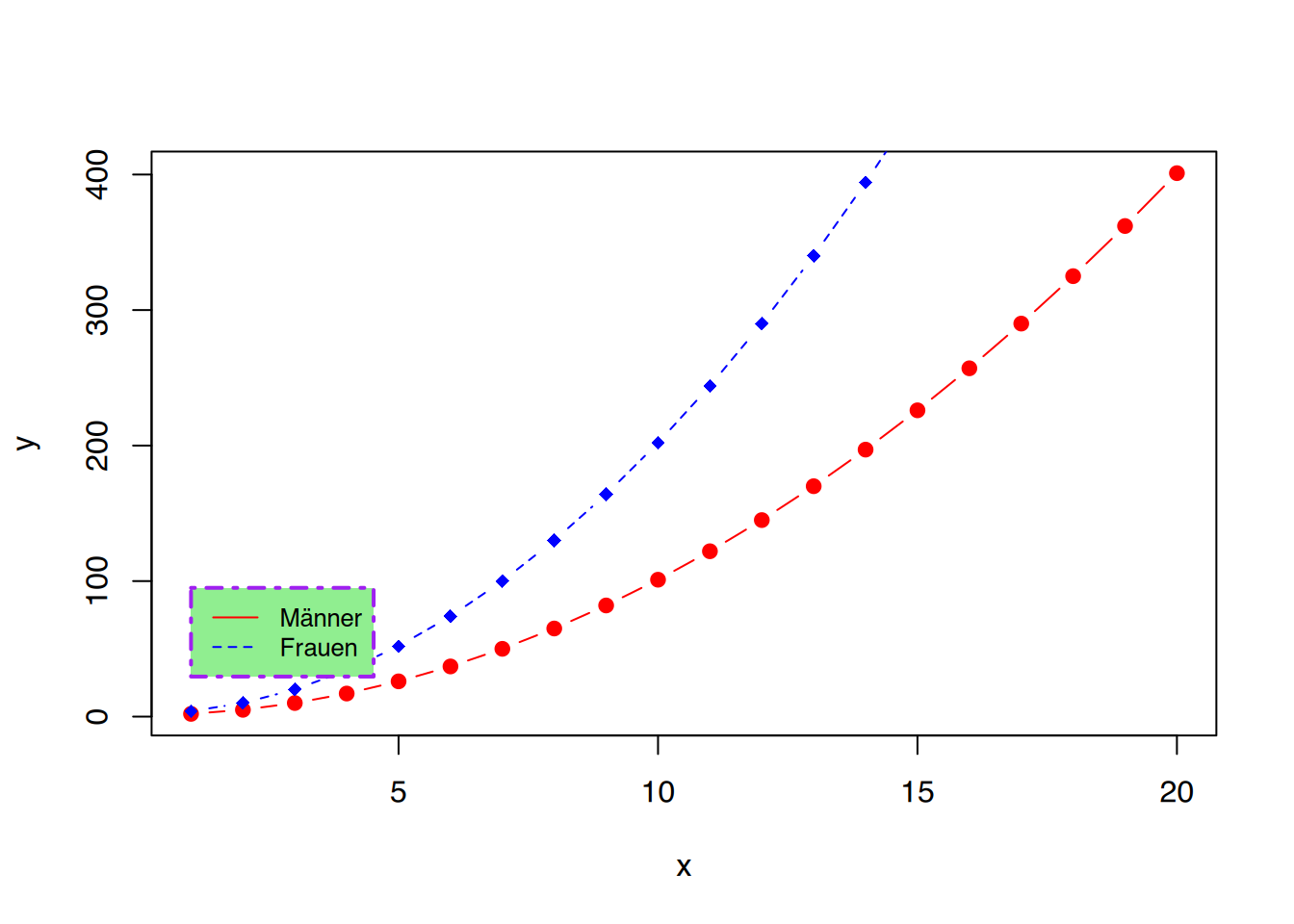
Die Position der Box kann auch mit den Keywords "bottomright", "bottom", "bottomleft", "left", "topleft", "top", "topright", "right" und "center" bestimmt werden.
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
lines(x, y2, pch=18, col="blue", type="b", lty=2)
# Füge Legendenbox hinzu
legend("topleft", legend=c("Männer", "Frauen"),
col=c("red", "blue"), lty=1:2, cex=0.8,
bg="lightgreen", box.lty = 4, box.lwd = 2, box.col = "purple")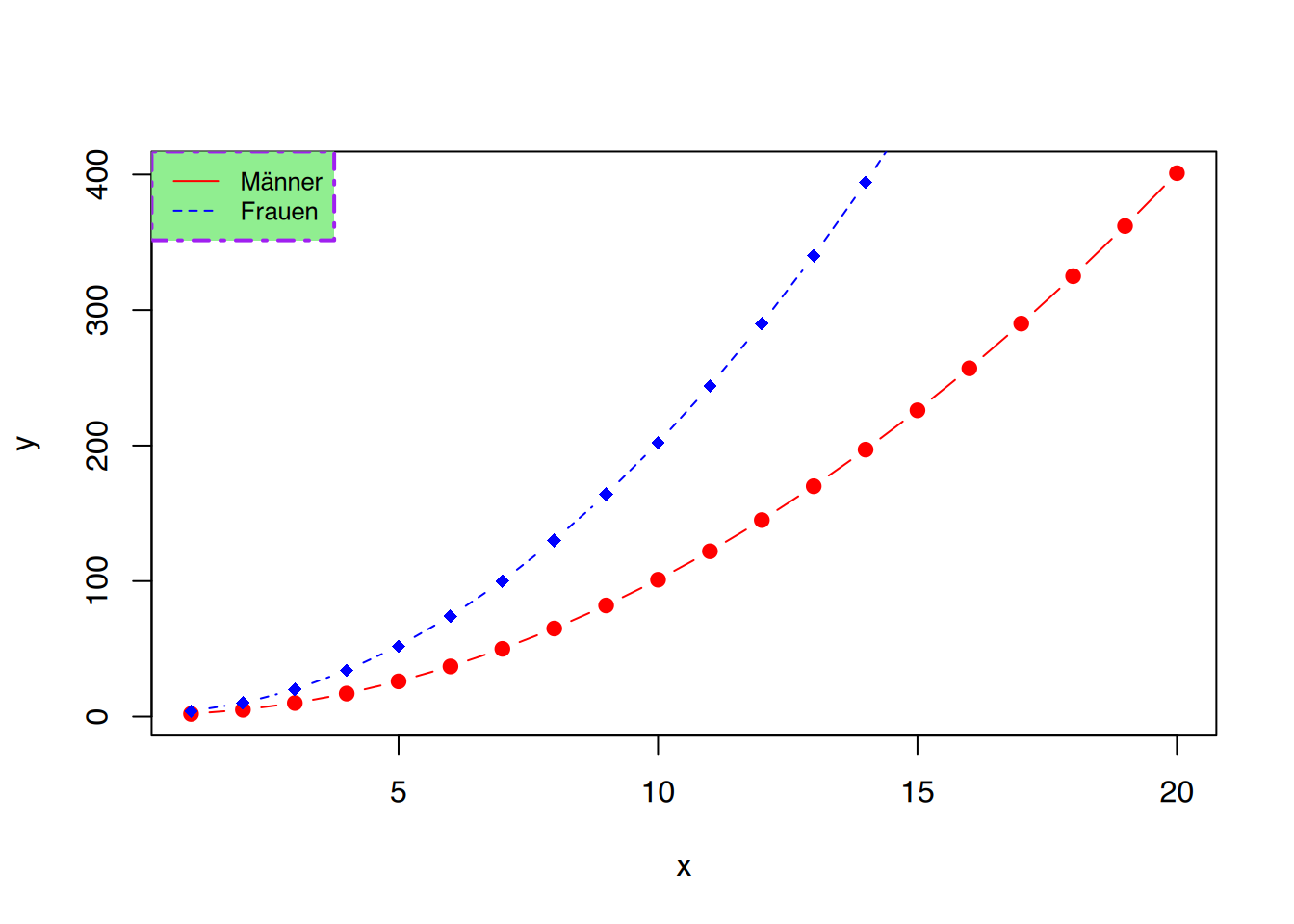
Mit dem Parameter horiz=TRUE werden die Variablennamen horizontal aneinandergereiht.
plot(x, y1, type="b", pch=19, col="red", xlab="x", ylab="y")
lines(x, y2, pch=18, col="blue", type="b", lty=2)
# Füge Legendenbox hinzu
legend("topleft", legend=c("Männer", "Frauen"),
col=c("red", "blue"), lty=1:2, cex=0.8,
bg="lightgreen", box.lty = 4, box.lwd = 2, box.col = "purple",
horiz=TRUE)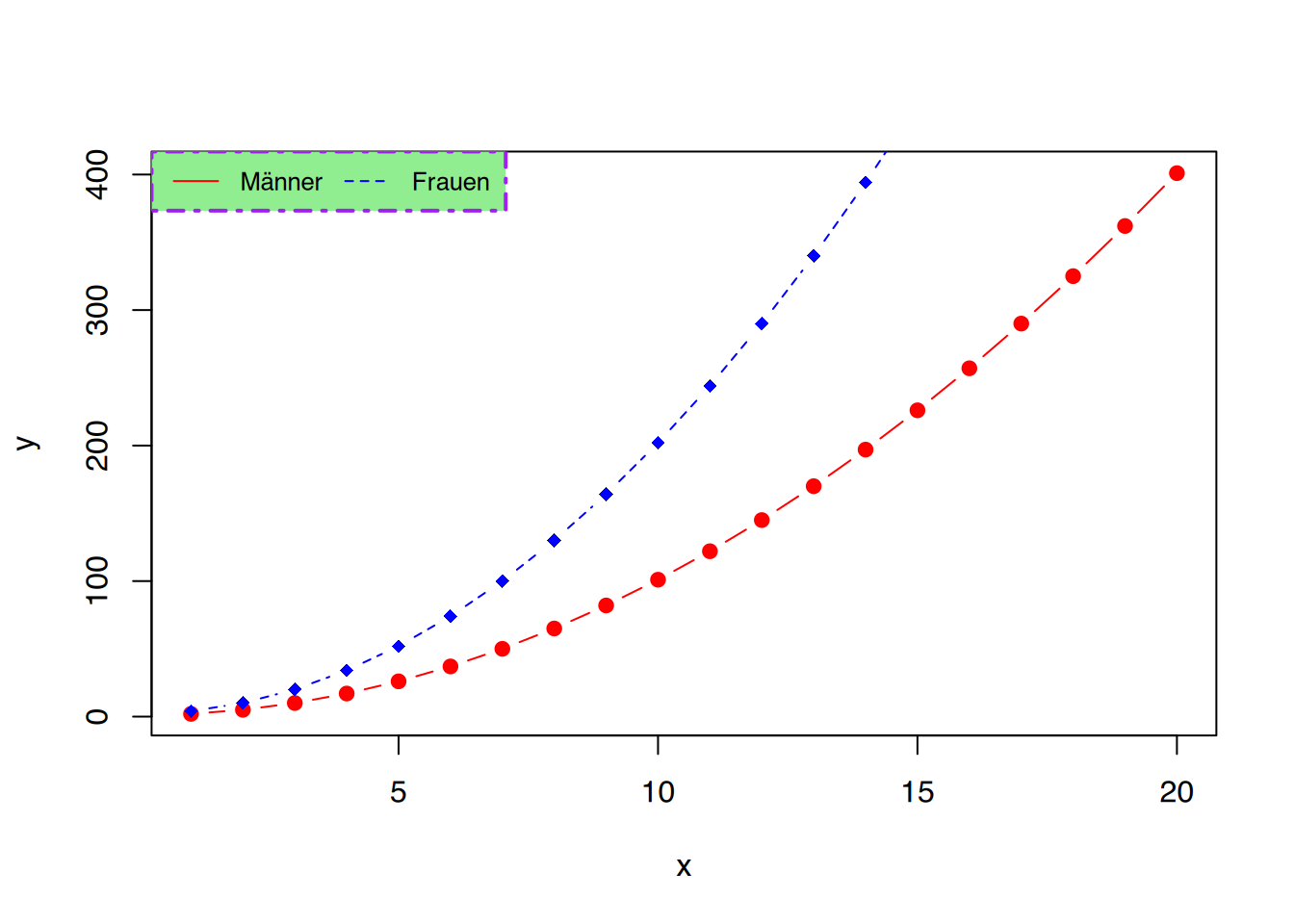
34.12 Diagramme speichern
Zum Speichern der Diagramme bietet die Formate jpeg, png, bmp und tiff an. Und genau so heissen auch die jeweiligen Speicherfunktionen. Dieser Befehl speichert das Diagramm im Format png:
# Speichere nachfolgendes als PNG
png("Testplot.png", width = 500, height = 500, units = "px", pointsize = 12)
# erzeuge Diagramm
pie(c(60,40))
# speichern abschließen
dev.off()Wie Sie sehen wird zunächst die Funktion png() aufgerufen. In ihr wird der Dateiname des Diagramms sowie dessen Dimensionen angegeben. Anschließend folgt der Aufruf der Plotbefehle, in diesem Falle pie(). Mit dem Befehl dev.off() (für device off ) wird das Speichern abgeschlossen. Die eigentlichen Diagrammbefehle müssen also zwischen der Speicherfunktion (png()) und dev.off() stehen.
Soll das Diagramm als jpeg gespeichert werden, lautet die Befehlskette entsprechend:
# Speichere nachfolgendes als JPEG
jpeg("Testplot2.jpg", width = 500, height = 500, units = "px", pointsize = 12)
# erzeuge Diagramm
barplot(c(10,40,20), names.arg=c("Dritter", "Erster", "Zweiter"))
# speichern abschließen
dev.off()Genau so funktionieren auch die Funktionen bmp() und tiff(), aber diese Formate verwendet heute eigentlich niemand mehr.
https://www.shirin-glander.de/2017/09/moving-my-blog-to-blogdown/↩︎