# ggplot2 installieren
install.packages("ggplot2", dependencies=T)
# ggplot2 aktivieren
library(ggplot2)35 Diagramme mit ggplot()
Das Zusatzpaket ggplot (für grammar of graphics plot) ist Hadley Wickhams (2012) R-Implementation der Grammar of Graphics von Leland Wilkinson (2005). Es ist eines der ersten Pakete des Tidyverse (welches damals noch nicht so hieß). Die Funktion zum plotten heisst ggplot(), das Installationspaket in R lautet jedoch ggplot2.
Unter https://ggplot2-book.org ist eine detaillierte Anleitung in englischer Sprache von Hadely Wickham et al. (2022) verfügbar.
Wenn Sie zuvor das Tidyverse (siehe Kapitel Kapitel 24) aktiviert haben…
library(tidyverse)… wird das ggplot2-Paket ebenfalls aktiviert.
Da ggplot aus dem Tidyverse stammt, ist es von Vorteil, wenn die zu verabreitenden Datensätze dem Prinzip “ein Fall pro Zeile” (tidy data bzw. long table) folgen. Das bedeutet, dass jede Beobachtung (auch Wiederholungen) in einer eigenen Zeile steht, und die jeweiligen Variablen durch die Spalten repräsentiert werden.
35.1 Satzbau
Die Idee einer Grammatik für Diagramme ist, dass jedes Diagramm aus den selben Komponenten (Satzteilen) aufgebaut werden kann. Benötigt werden:
- Daten (möglichst im Tidy Data Format)
- Aesthetische bzw. visuelle Zuordnungen der Daten zu den Komponenten des Plots
- Geometrische Objekte, die im Plot erscheinen sollen
- Statistische Transformation der Daten, bevor sie geplottet werden
- Coordinates (Koordinaten), um die Lage der geometrischen Objekte zu bestimmen
- Scales (Skalen), um den Wertebereich der Datenzuordnung zu bestimmen
- Facetten, um den Plot in Teil-Diagramme zu gruppieren
Diese Komponenten werden in ggplot() mit dem + Zeichen aneinander gereiht.
# sinnbildlich
ggplot(daten) +
visuelle Zuordnung +
Geometrische Objekte +
statistische Transformationen +
Koordinaten +
Skalen +
FacettenDie enthaltenen Funktionen folgen dabei dieser Komponentenstruktur:
Geome (Flächen, Linien, Punkte, usw) beginnen mit
geom_, z.B.:gemo_area(),gemo_density(),gemo_smooth(),gemo_histogram(),gemo_point(),gemo_jitter(),gemo_line(),gemo_boxplot(),gemo_col(),gemo_text()
statistische Funktionen (Häufigkeiten, Mittelwerte, Dichte, usw.) beginnen mit
stat_, z.B.:stat_count(),stat_density(),stat_function(),stat_summary()
Koordinatensystemfunktionen beginnen mit
coor_, z.B.:coord_flip(),coord_trans()
Funktionen zum Manipulieren des grundlegenden Aussehens (Theme), beginnend mit
theme_, z.B.:theme(),theme_bw(),theme_classic()
In RStudio ist ein “Cheatsheet” (siehe Abschnitt 30.1) zu ggplot verlinkt. Hier finden Sie (fast) alle verfügbaren Komponenten zusammengefasst. Klicken Sie in der Menüzeile von RStudio auf Help \(\rightarrow\) Cheatsheets \(\rightarrow\) Data Visualization with ggplot2.
35.2 Erstes Plot
Mit dem Datensatz jgsbook::pf8 erzeugen wir nun unser erstes ggplot.
# Lade Datensatz
load(url("https://www.produnis.de/R/data/pf8.RData"))
# oder
pf8 <- jgsbook::pf8
# Tidyverse aktivieren!
library(tidyverse)
# unser erstes Plot
ggplot(pf8)
“Wie Sie sehen, sehen Sie nichts.”
Wir sehen deshalb nichts, weil wir noch nicht bestimmt haben, was (welche Variablen) wie (Punkte, Linien, Boxplots) wo (x-Achse, y-Achse) geplottet werden soll. Wir müssen diese “Aesthetics” erst definieren.
35.2.1 Aesthetics
Angenommen, uns interessiert das Gewicht sowie das Alter der Probanden. Mit der Funktion aes() können wir die Daten visuell zuordnen. Das Alter soll auf der x-Achse liegen, das Gewicht auf der y-Achse.
# erster Plot mit "Aesthetics"
ggplot(pf8) +
aes(x = Alter, # x-Achse
y = Gewicht) # y-Achse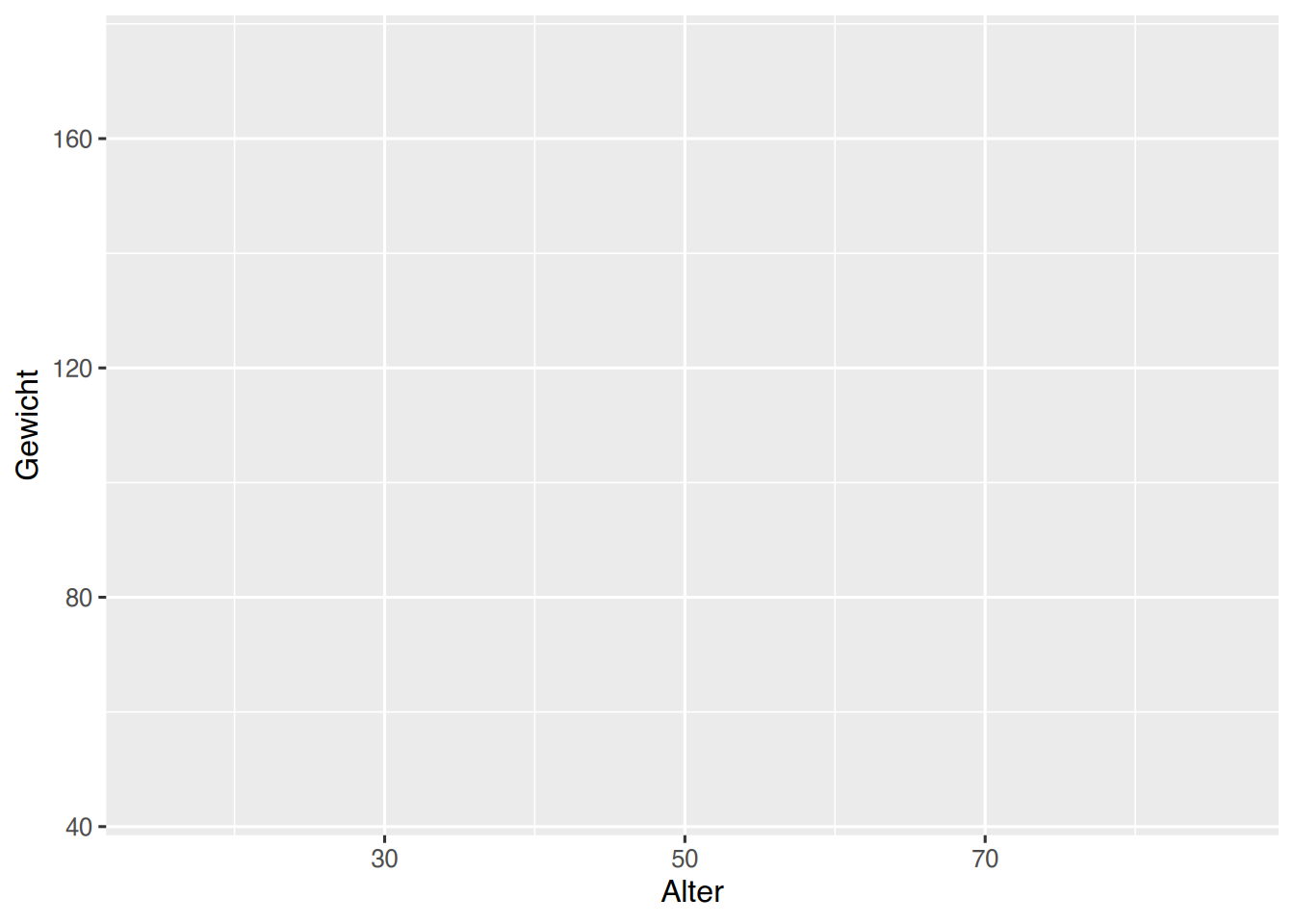
Ggplot hat die Achsen entsprechend beschriftet und die Wertebereiche anhand der vorhandenen Daten (Minimum, Maximum) ausgewählt.
35.2.2 Geome
Zur Darstellung von Gewicht und Alter werden geometrische Objekte (Geome) benötigt. Diese werden über die Funktionen geom_*() erzeugt, wobei das Sternchen durch die gewünschte gemoetrischen Form ersetzt werden muss.
Die 6 meist-genutzten Geome sind sicherlich:
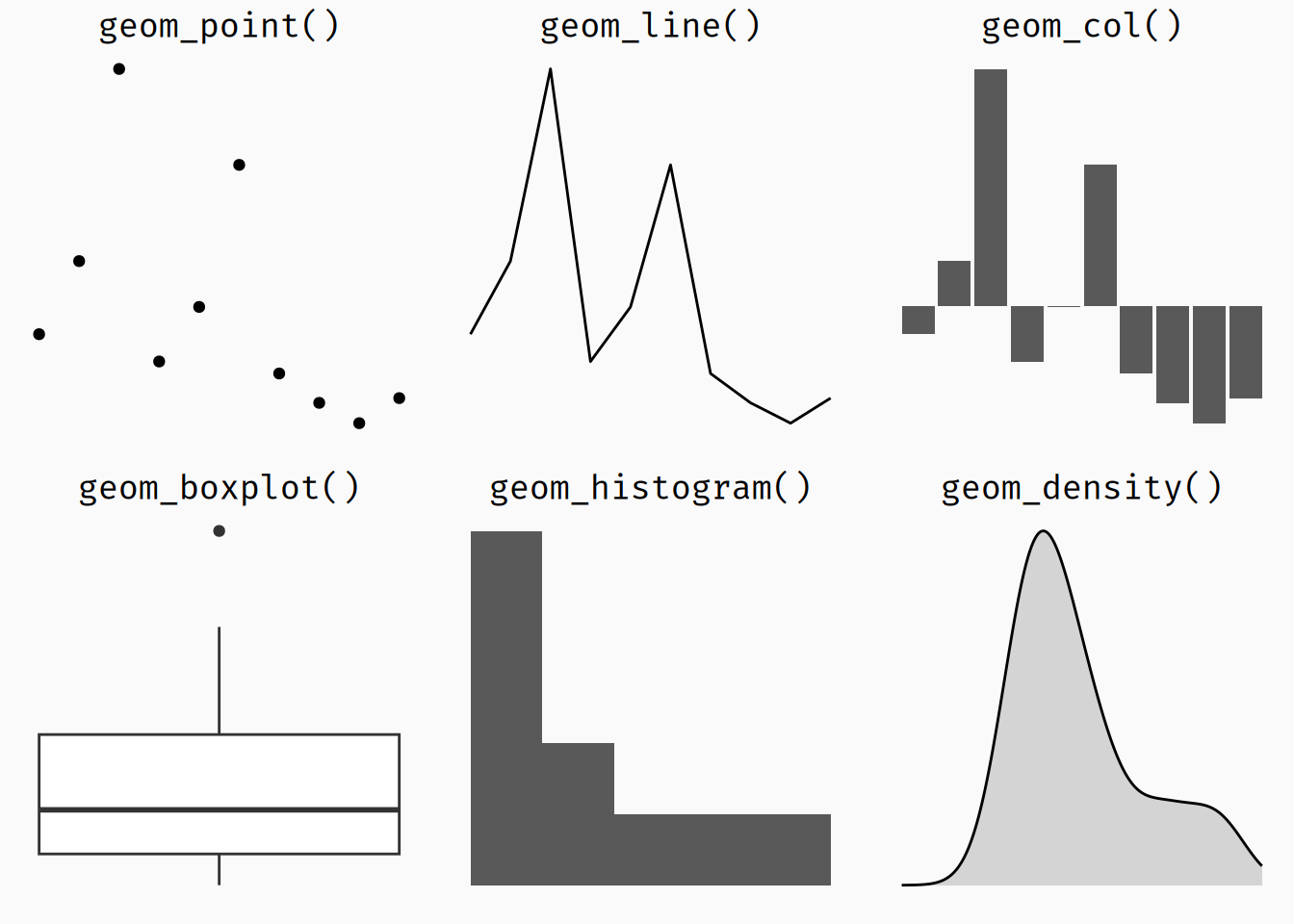
Insgesamt sind 53 Geome verfügbar:
## [1] "geom_abline" "geom_area" "geom_bar"
## [4] "geom_bin_2d" "geom_bin2d" "geom_blank"
## [7] "geom_boxplot" "geom_col" "geom_contour"
## [10] "geom_contour_filled" "geom_count" "geom_crossbar"
## [13] "geom_curve" "geom_density" "geom_density_2d"
## [16] "geom_density_2d_filled" "geom_density2d" "geom_density2d_filled"
## [19] "geom_dotplot" "geom_errorbar" "geom_errorbarh"
## [22] "geom_freqpoly" "geom_function" "geom_hex"
## [25] "geom_histogram" "geom_hline" "geom_jitter"
## [28] "geom_label" "geom_line" "geom_linerange"
## [31] "geom_map" "geom_path" "geom_point"
## [34] "geom_pointrange" "geom_polygon" "geom_qq"
## [37] "geom_qq_line" "geom_quantile" "geom_raster"
## [40] "geom_rect" "geom_ribbon" "geom_rug"
## [43] "geom_segment" "geom_sf" "geom_sf_label"
## [46] "geom_sf_text" "geom_smooth" "geom_spoke"
## [49] "geom_step" "geom_text" "geom_tile"
## [52] "geom_violin" "geom_vline"In unserem Beispiel möchten wir das Gewicht sowie das Alter der Probanden als Punktwolke darstellen. Das geeignete Geom ist geom_point().
# unser erstes Plot, mit "Aesthetics" und "Geom"
ggplot(pf8) +
aes(x = Alter,
y = Gewicht) +
geom_point()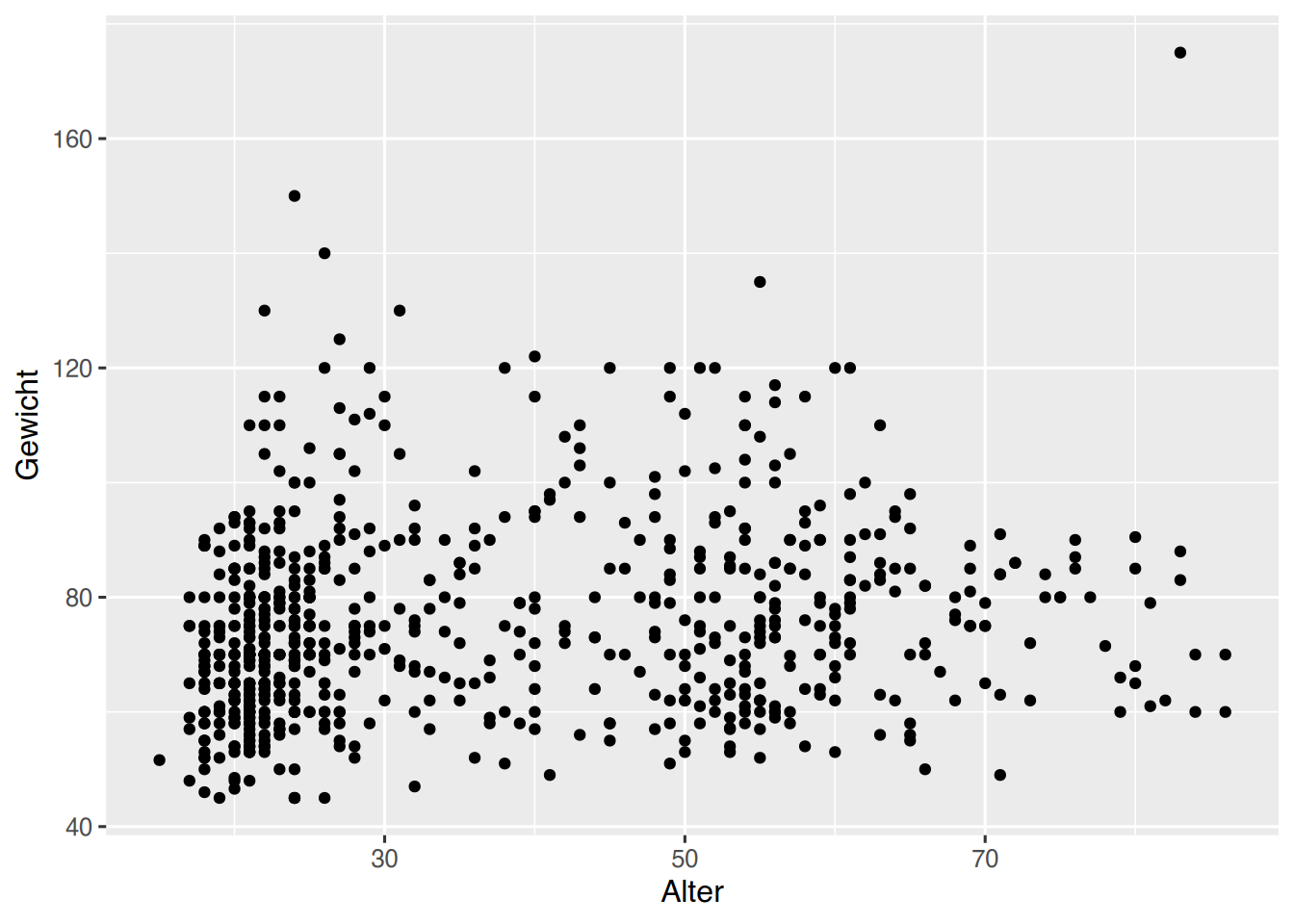
35.2.3 mehr Aesthetics
Angenommen, wir möchten das Gewicht sowie das Alter der Probanden als Punktwolke darstellen, aber graphisch zwischen den Geschlechtern unterscheiden. Hierzu erweitern wir dies aes()-Funktion um den Parameter color.
# unser erstes Plot, mit “Aesthetics” und “Geom”,
# farblich nach Geschlecht
ggplot(pf8) +
aes(x = Alter,
y = Gewicht,
color = Geschlecht) +
geom_point()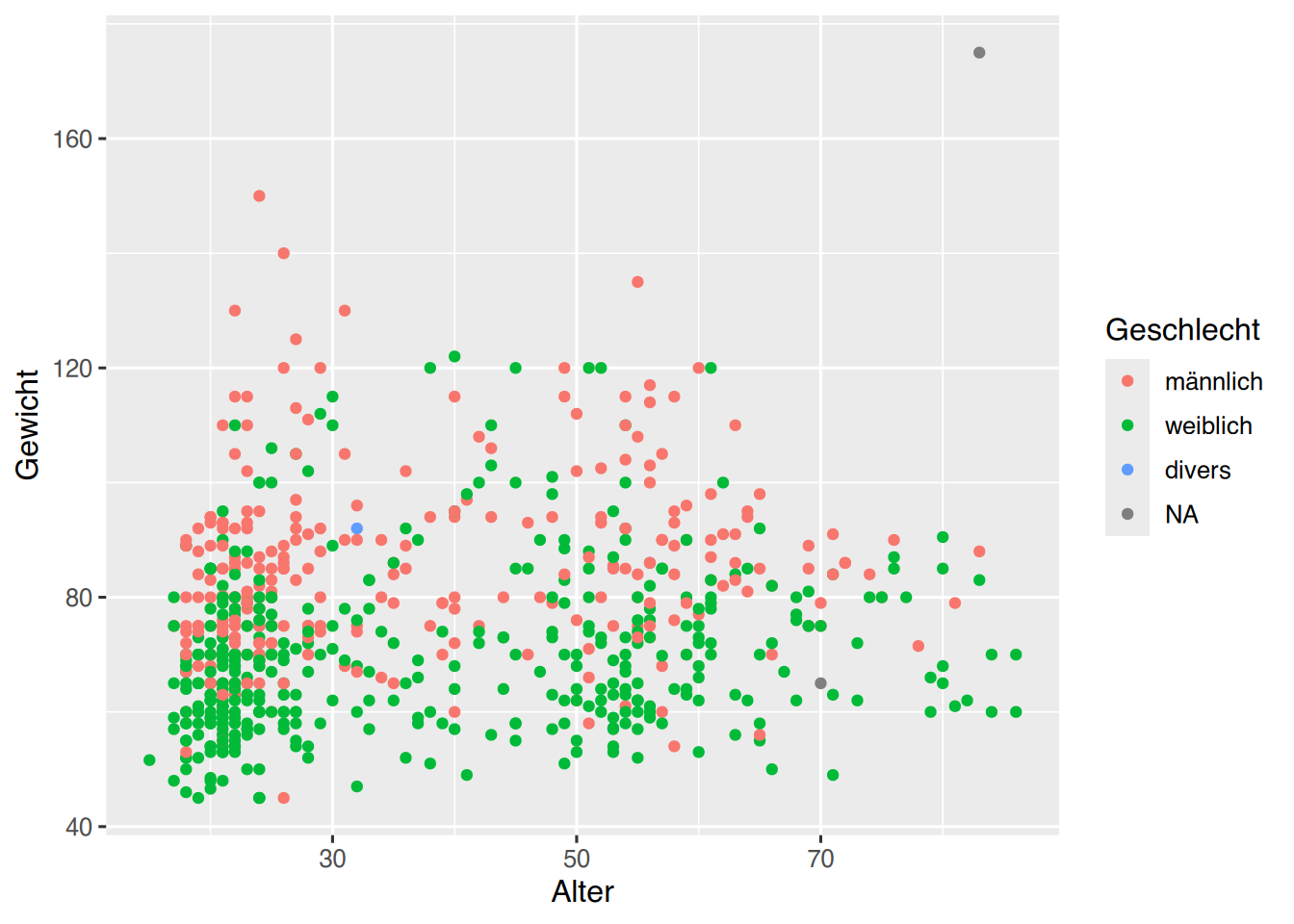
Ggplot stellt die Werte in unterschiedlichen Farben pro Geschlechterkategorie dar. Ebenso wurde das Plot um eine Legendenbox erweitert, in welcher die Farbzuordnungen pro Geschlechtskategorie angezeigt ist.
35.2.4 mehr Geome
Über das + Zeichen können beliebig viele Geome dem Plot hinzugefügt werden. Dabei werden die aesthetics automatisch an die Geome weitergegeben, sie können aber auch in jedem Geom per aes() neu definiert werden.
In unserem Beispiel möchten wir nun eine Regressionsgerade mit Konfidenzintervall einzeichnen. Dies kann mit geom_smooth() umgesetzt werden. Über den Parameter method können wir angeben, dass ein lineares Modell (“lm”) eingezeichnet werden soll.
# unser erstes Plot, mit “Aesthetics” und zwei “Geomen”
# und Regressionsmodell
ggplot(pf8) +
aes(x = Alter,
y = Gewicht,
color = Geschlecht) +
geom_point() +
geom_smooth(method="lm") 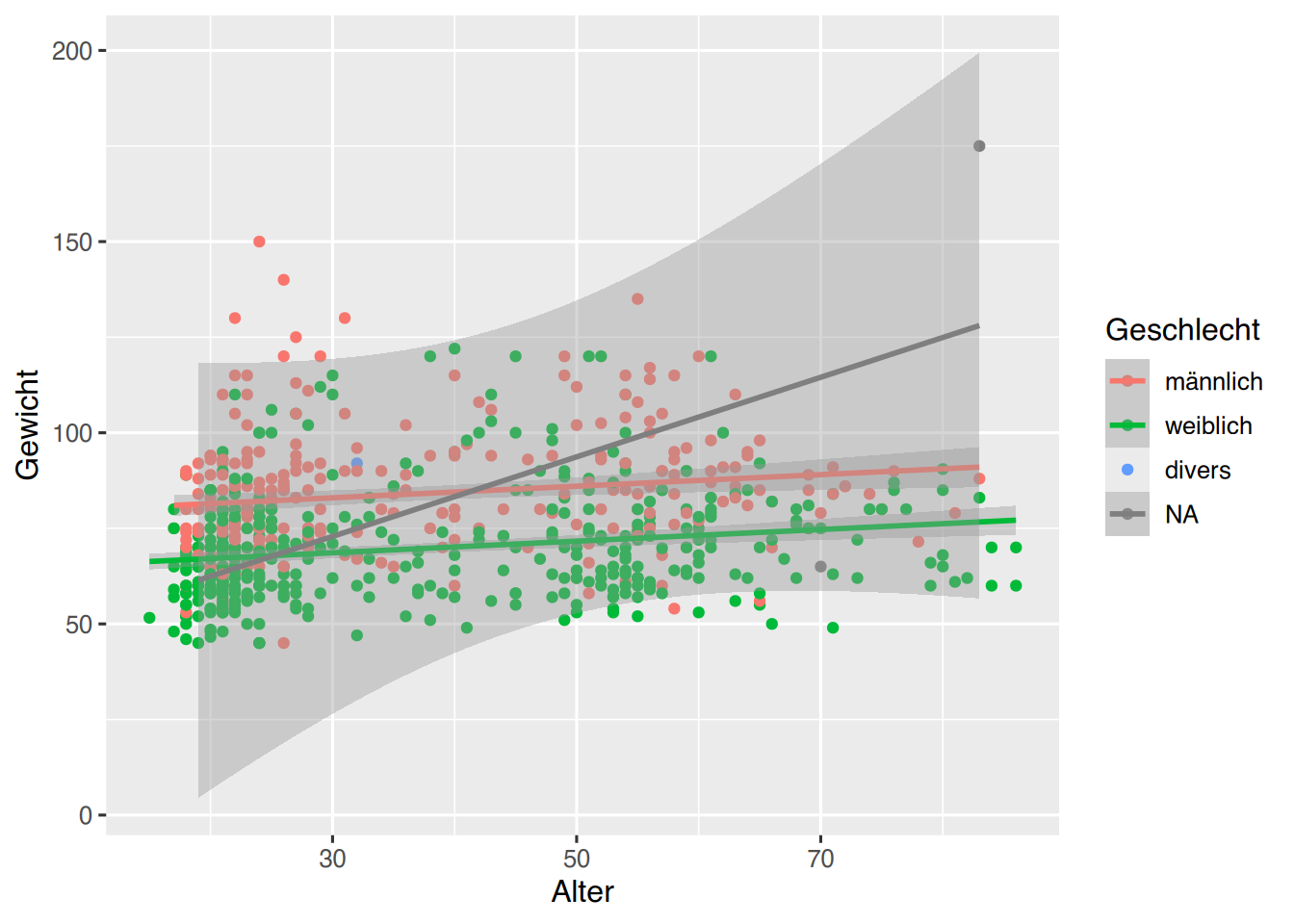
In der Variablen Geschlecht sind NA enthalten. Diese können wir mit der Funktion drop_na() aussortieren und direkt an ggplot() weiter-pipen.
# unser erstes Plot, mit “Aesthetics” und zwei “Geomen”
# und Regressionsmodell und
# OHNE NA
pf8 %>%
drop_na(Geschlecht) %>%
ggplot() +
aes(x = Alter,
y = Gewicht,
color = Geschlecht) +
geom_point() +
geom_smooth(method="lm")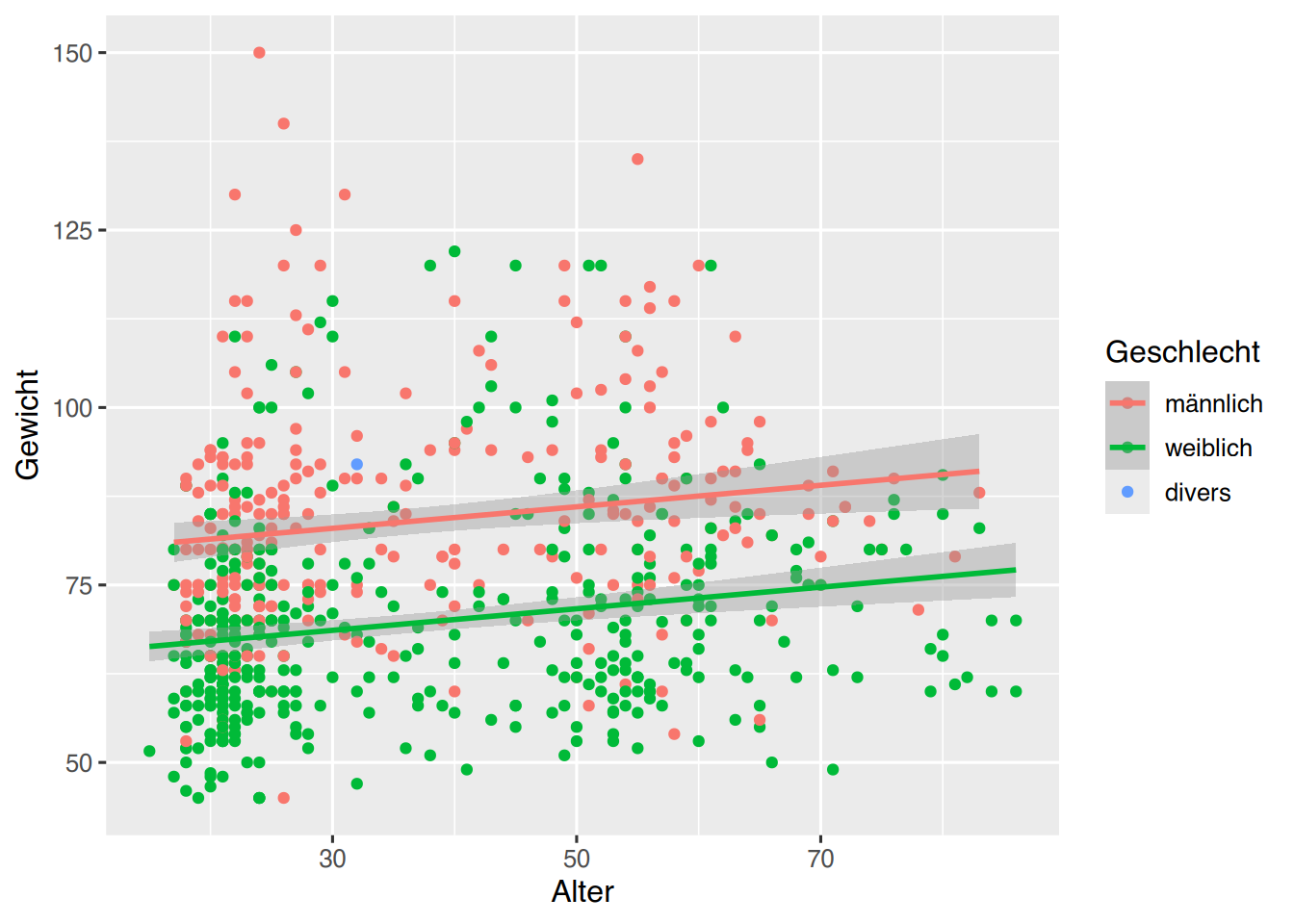
Es wird ausdrücklich empfohlen, sich über die Hilfeseiten, z.B. ?geom_smooth, mit den Funktionen vertraut zu machen. So erfährt man dort beispielsweise, dass der Konfidenzbereich per se=FALSE ausgeblendet werden kann.
# unser erstes Plot, mit “Aesthetics” und zwei “Geomen”
# und Regressionsmodell ohne Konfidenzbereich
# OHNE NA
pf8 %>%
drop_na(Geschlecht) %>%
ggplot() +
aes(x = Alter,
y = Gewicht,
color = Geschlecht) +
geom_point() +
geom_smooth(method="lm", se=FALSE)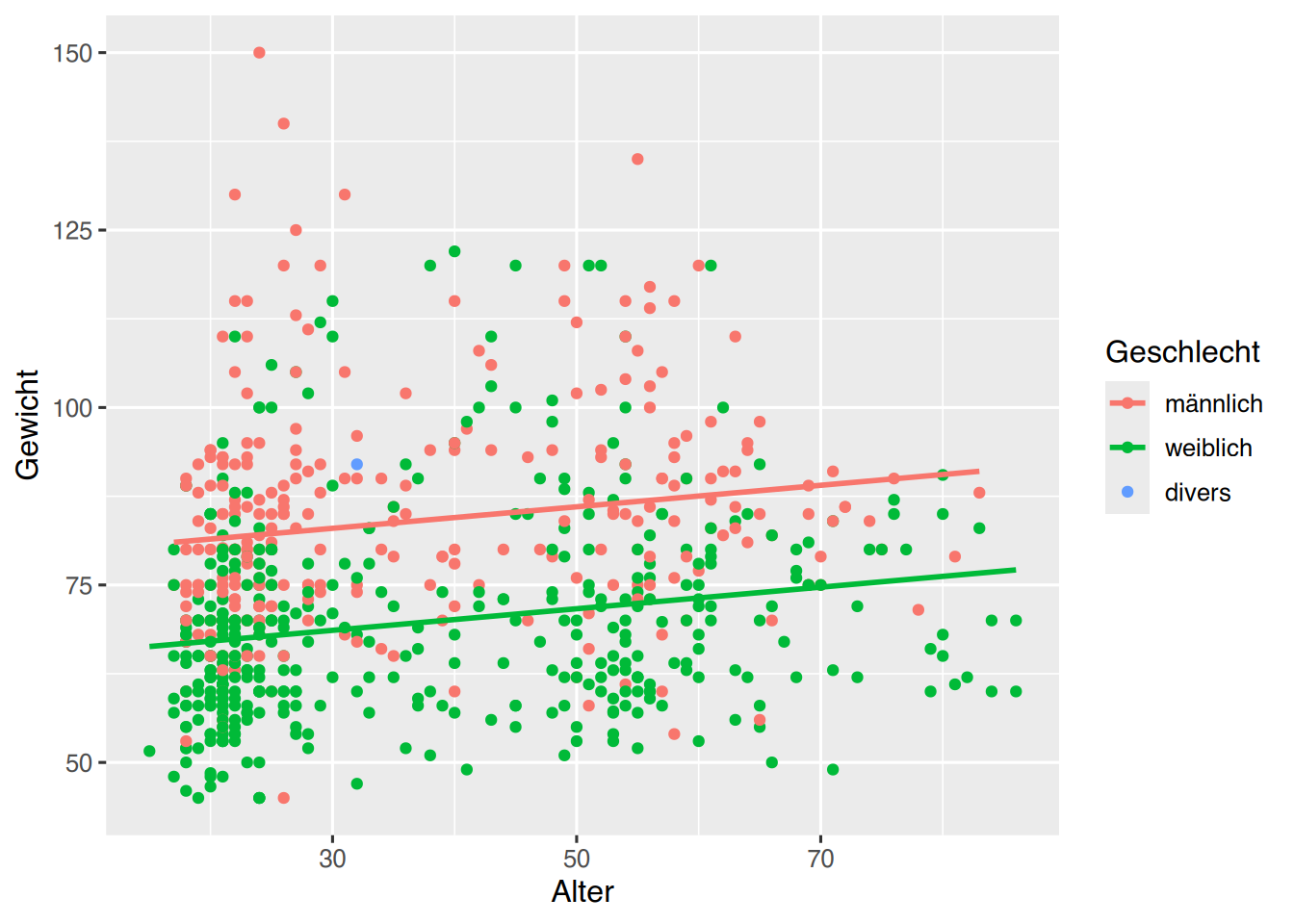
35.2.5 Facetten
Mit den Funktionen facet_wrap() und facet_grid() können Teil-Diagramme innerhalb des Gesamtplots erzeugt werden.
So können wir beispielsweise ein Plot pro Standort erstellen.
# unser erstes Plot, mit “Aesthetics”,
# “Geom” und Facetten
pf8 %>%
drop_na(Geschlecht) %>%
ggplot() +
aes(x = Alter,
y = Gewicht,
color = Geschlecht) +
geom_point() +
geom_smooth(method="lm") +
facet_wrap(~ Standort)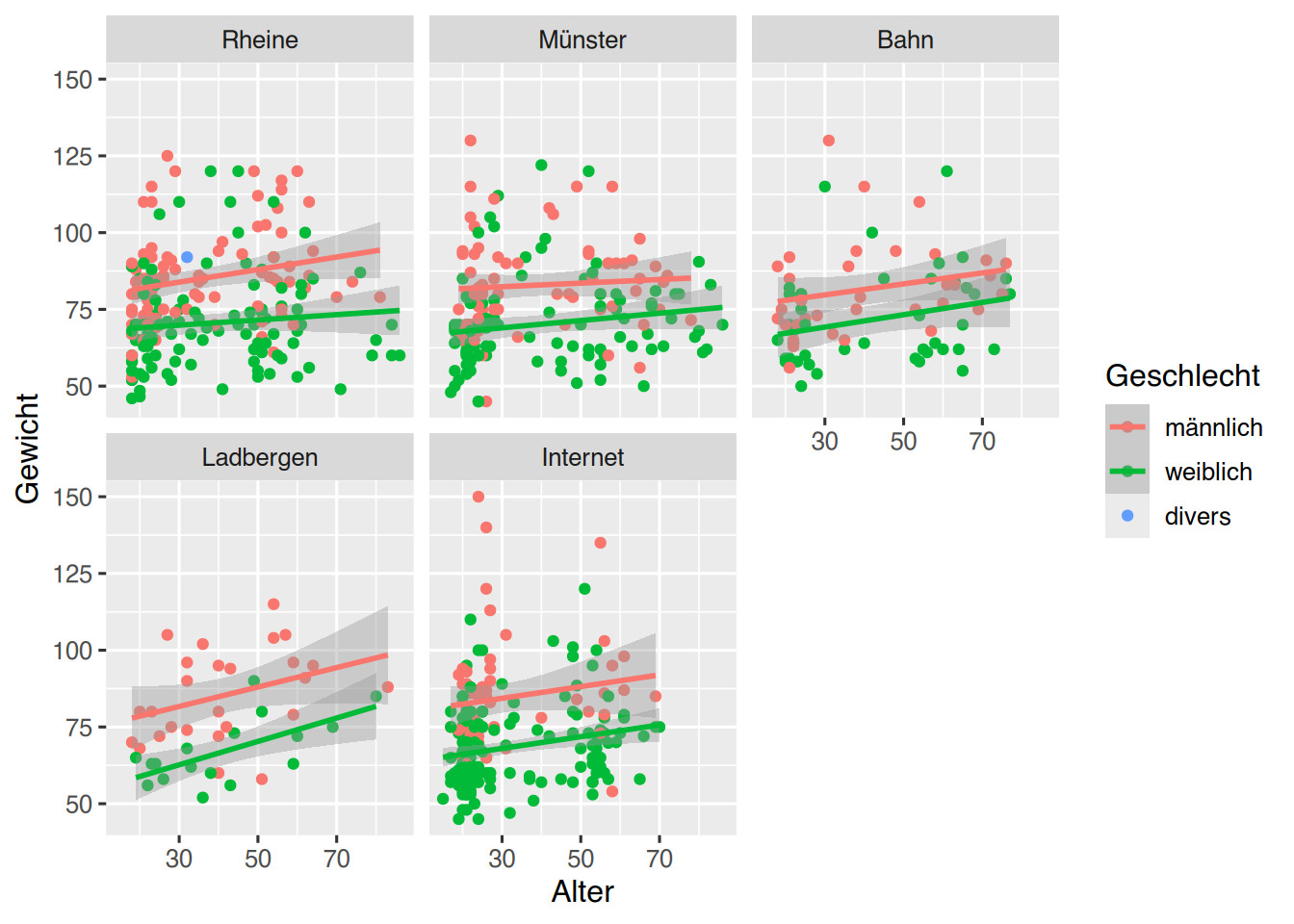
Oder wir erstellen ein Plot pro Rauchen-Kategorie.
# unser erstes Plot, mit “Aesthetics”,
# “Geom” und Facetten
pf8 %>%
drop_na(Geschlecht) %>%
ggplot() +
aes(x = Alter,
y = Gewicht,
color = Geschlecht) +
geom_point() +
geom_smooth(method="lm") +
facet_wrap(~ Rauchen)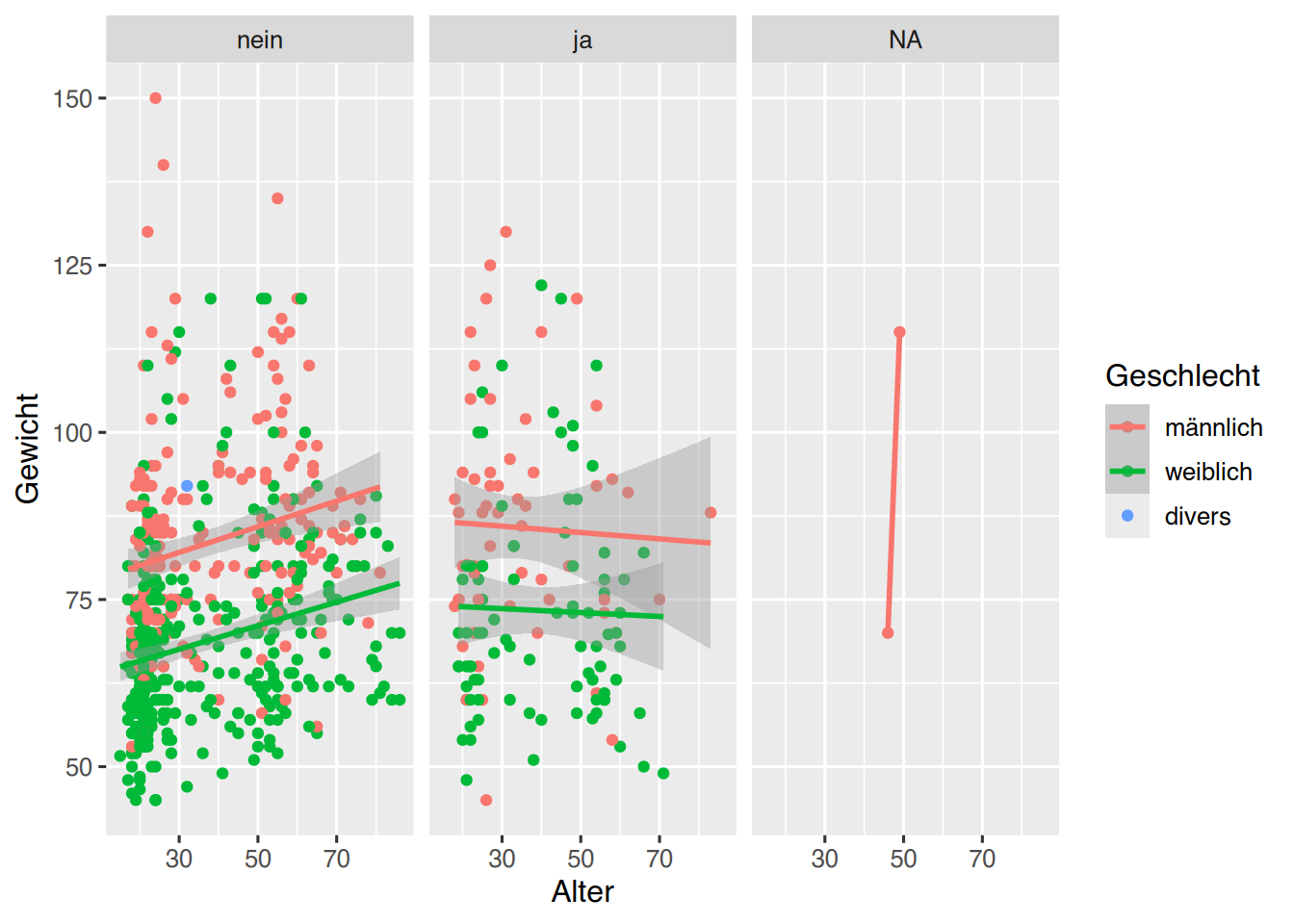
Mit der Funktion facet_grid() können Plot-Gitter erzeugt werden. So können wir beispielsweise ein Plot pro Standort und Rauchen erstellen.
# unser erstes Plot, mit “Aesthetics”,
# “Geom” und Facetten
pf8 %>%
drop_na(Geschlecht) %>%
ggplot() +
aes(x = Alter,
y = Gewicht,
color = Geschlecht) +
geom_point() +
geom_smooth(method="lm") +
facet_grid(Rauchen ~ Standort)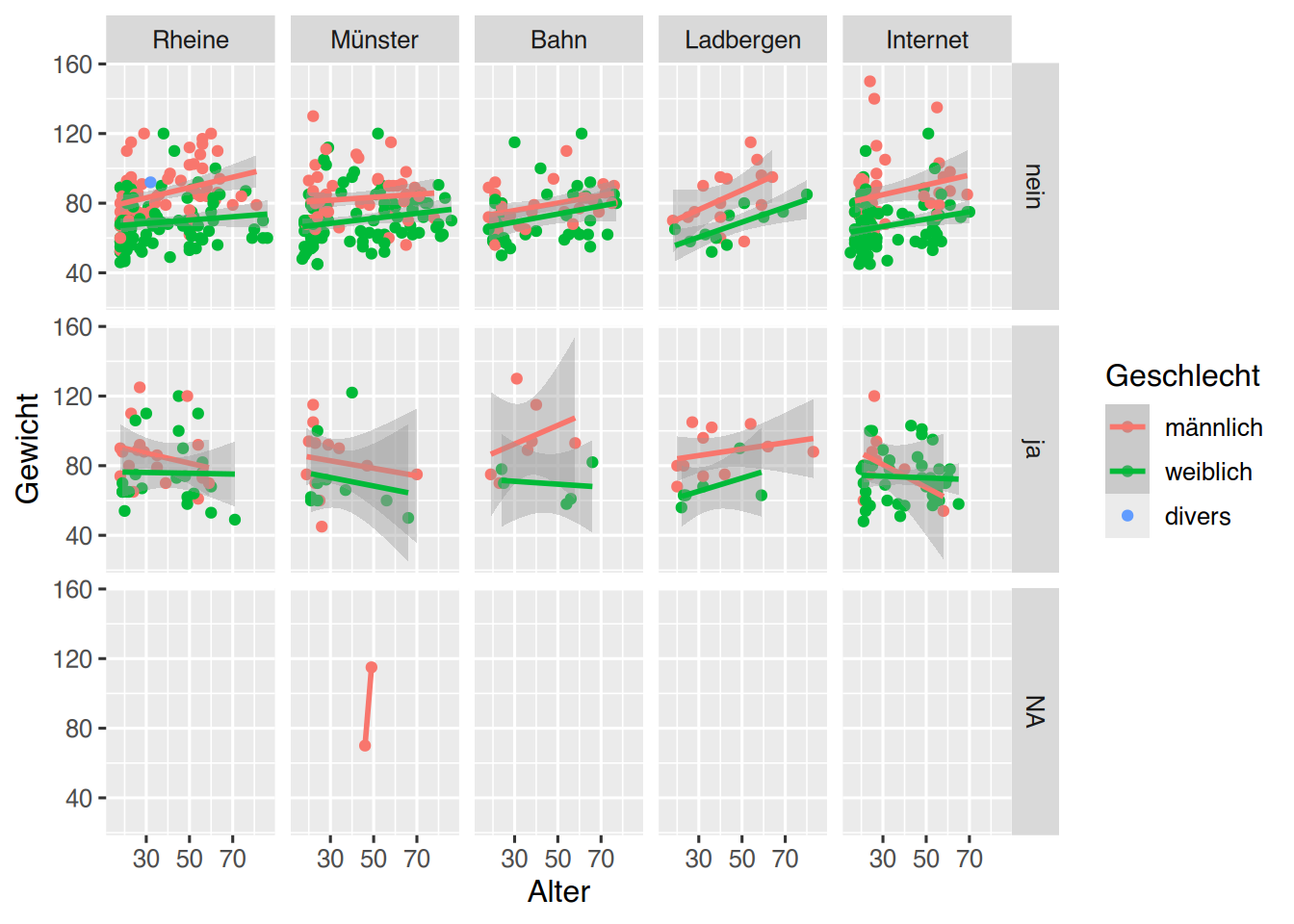
Mittels drop_na() können wir uns der NA-Zeile entledigen.
# unser erstes Plot, mit “Aesthetics”,
# “Geom” und Facetten
pf8 %>%
drop_na(Geschlecht, Rauchen) %>%
ggplot() +
aes(x = Alter,
y = Gewicht,
color = Geschlecht) +
geom_point() +
geom_smooth(method="lm") +
facet_grid(Rauchen ~ Standort)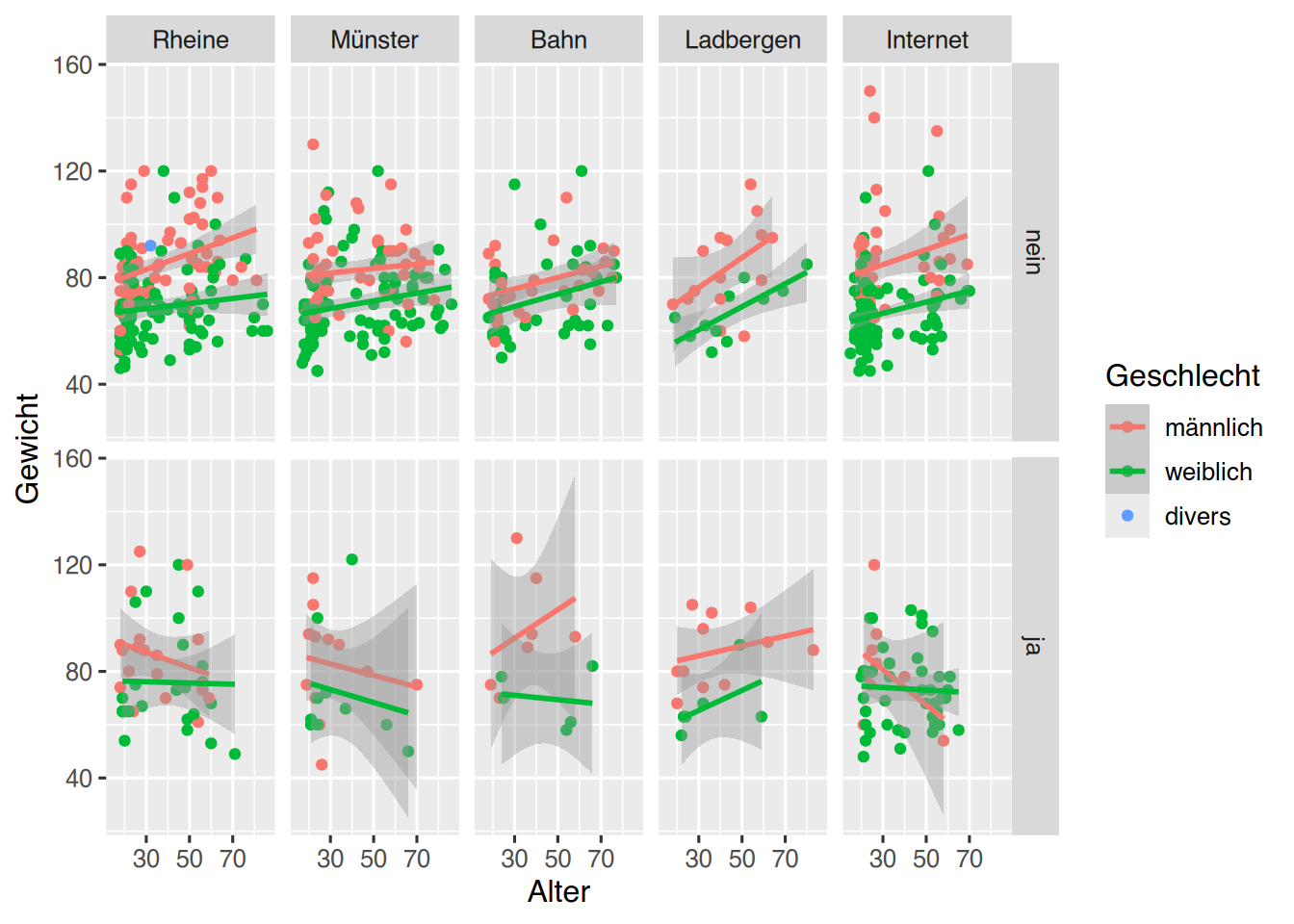
35.2.6 Geom-Parameter
Geome können mit verschiedenen Parametern beeinflusst werden. Welche Parameter zur Verfügung stehen, verrät die Hilfeseite der Geome. Die meisten reagieren auf:
geom_*(data, aes, stat, position, PARAMETER)
data: Geome übernehmen das Datenobjekt aus der
ggplot()-Funktion. Sie können aber auch eigene Daten verarbeitenaes: Geome erben die Zuordnungen aus der
aes()-Funktion. Sie können aber auch eigene aesthetics haben.stat: Geome können statistische Transformationen vornehmen, z.B.
stat='identity'.position: die Positionierung der Objekte lässt sich anpassen, z.B.
'dodge','stack','jitter','fill'.weitere PARAMETER sind:
color: (Rahmen)farbefill: Füllfarbesize: Objektgrößeshape: Objektformen

Die Parameter können auch innerhalb der `aes()`-Funktion verwendet werden, so dass anhand der Daten entschieden wird, welche Größe, Farbe und Form die Geome haben.linetype: Linientypen

35.3 Punktwolke
Punktwolken (Scatterplots) werden mit der Funktion geom_point() erzeugt. Wir plotten die Variablen Größe und Gewicht aus dem pf8-Datensatz.
ggplot(pf8) +
aes(x=Größe, y=Gewicht) +
geom_point()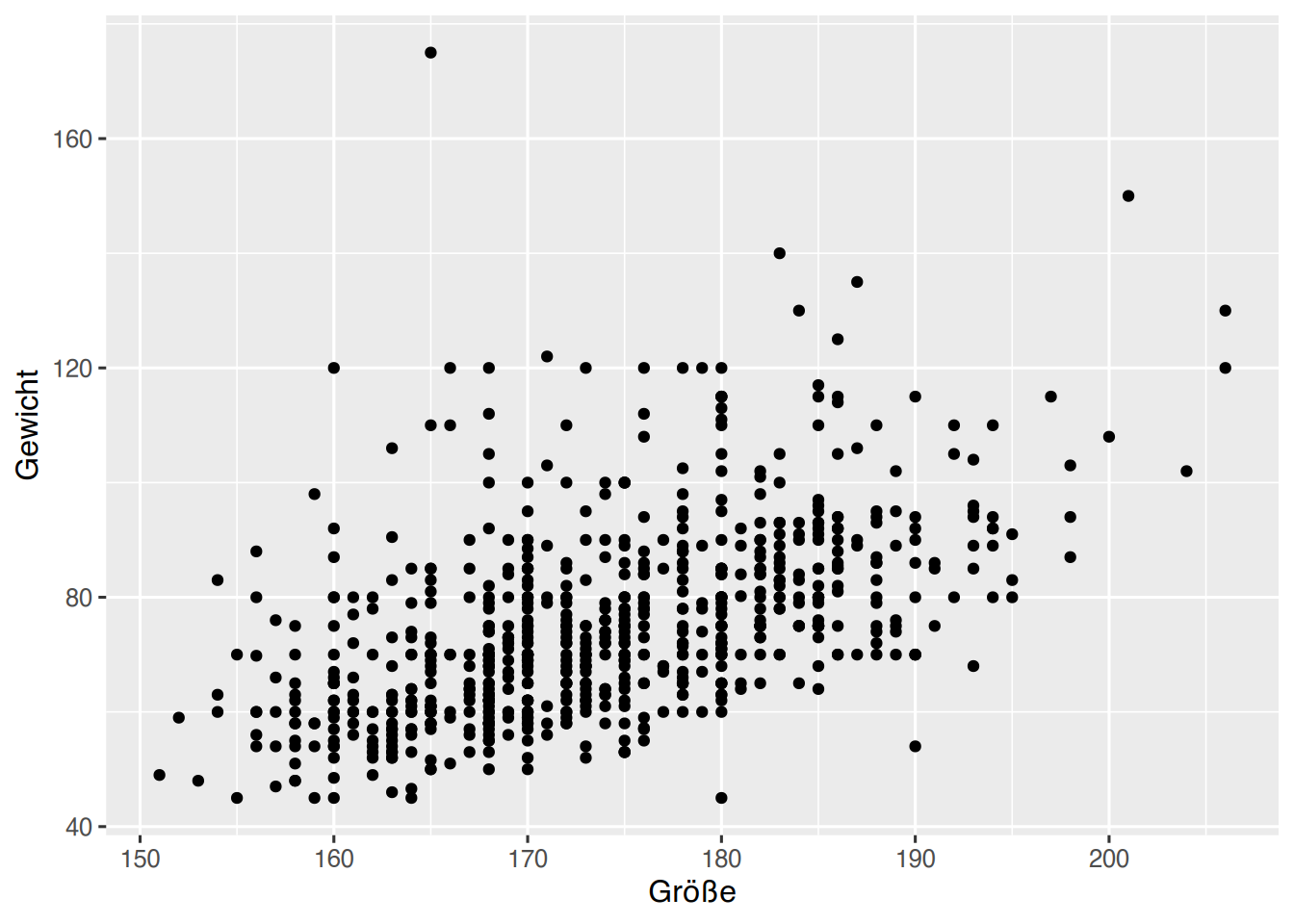
Über die Parameter color, shape und size können wir die Punkte manipulieren.
ggplot(pf8) +
aes(x=Größe, y=Gewicht) +
geom_point(color="blue",
shape=13,
size=10)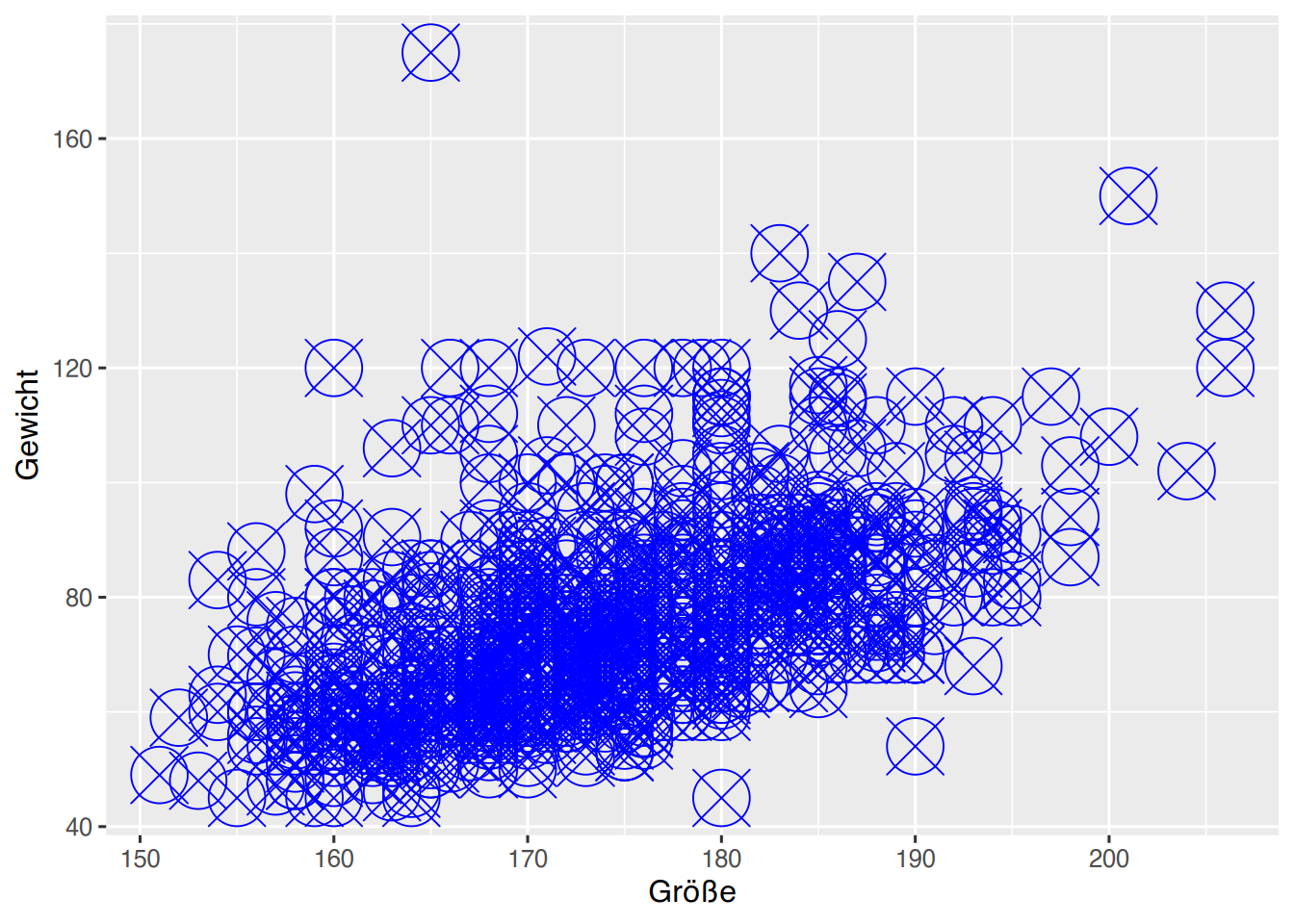
Die Parameter können auch als aesthetics verwendet werden, so dass anhand der Daten entschieden wird, welche Größe, Farbe und Form die Punkte haben.
ggplot(pf8) +
aes(x=Größe, y=Gewicht,
color=Geschlecht,
shape=Geschlecht,
size=Alter) +
geom_point()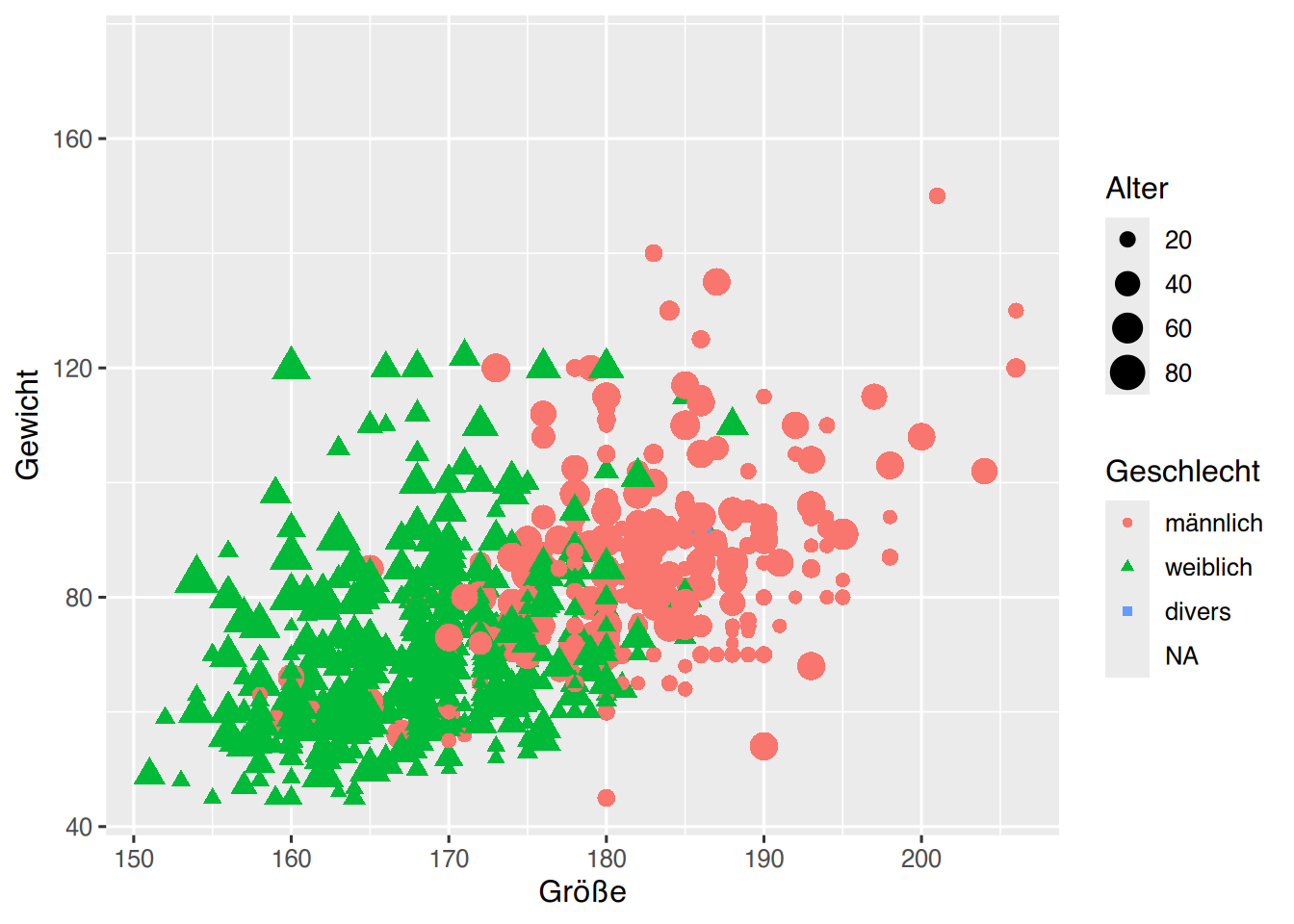
35.4 Liniendiagramm
Liniendiagramme werden mit der Funktion geom_line() erzeugt.
# Erzeuge ein paar Daten
df <- data.frame(Jahr=c(2020:2023),
Wert=c(3.2, 11, 31.1, 78.3))
# Plotte als Liniendiagramm
ggplot(df, aes(x=Jahr, y=Wert)) +
geom_line()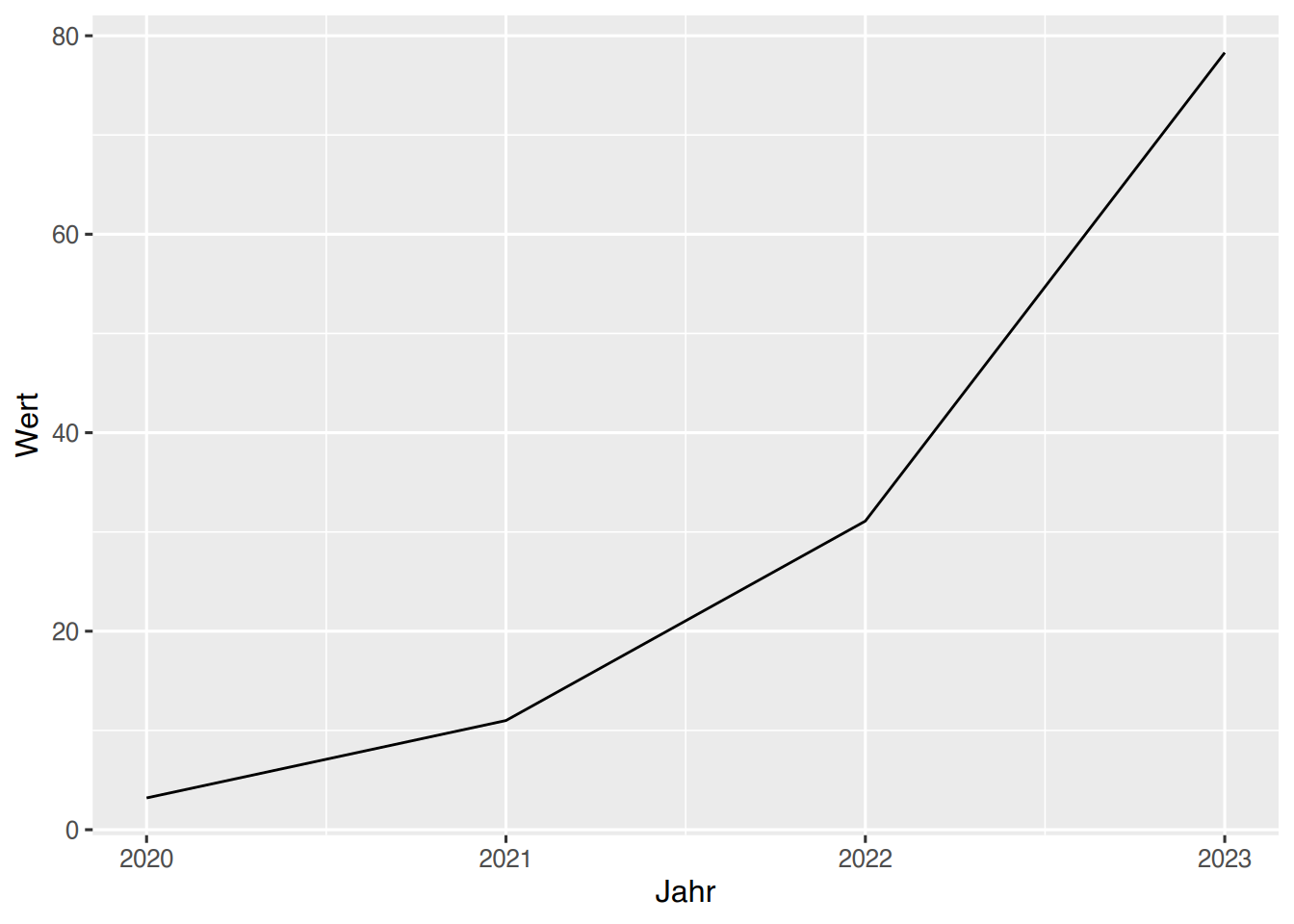
Über den Parameter linetype kann der Linientyp festgelegt werden.
ggplot(df, aes(x=Jahr, y=Wert)) +
geom_line(linetype = "dashed")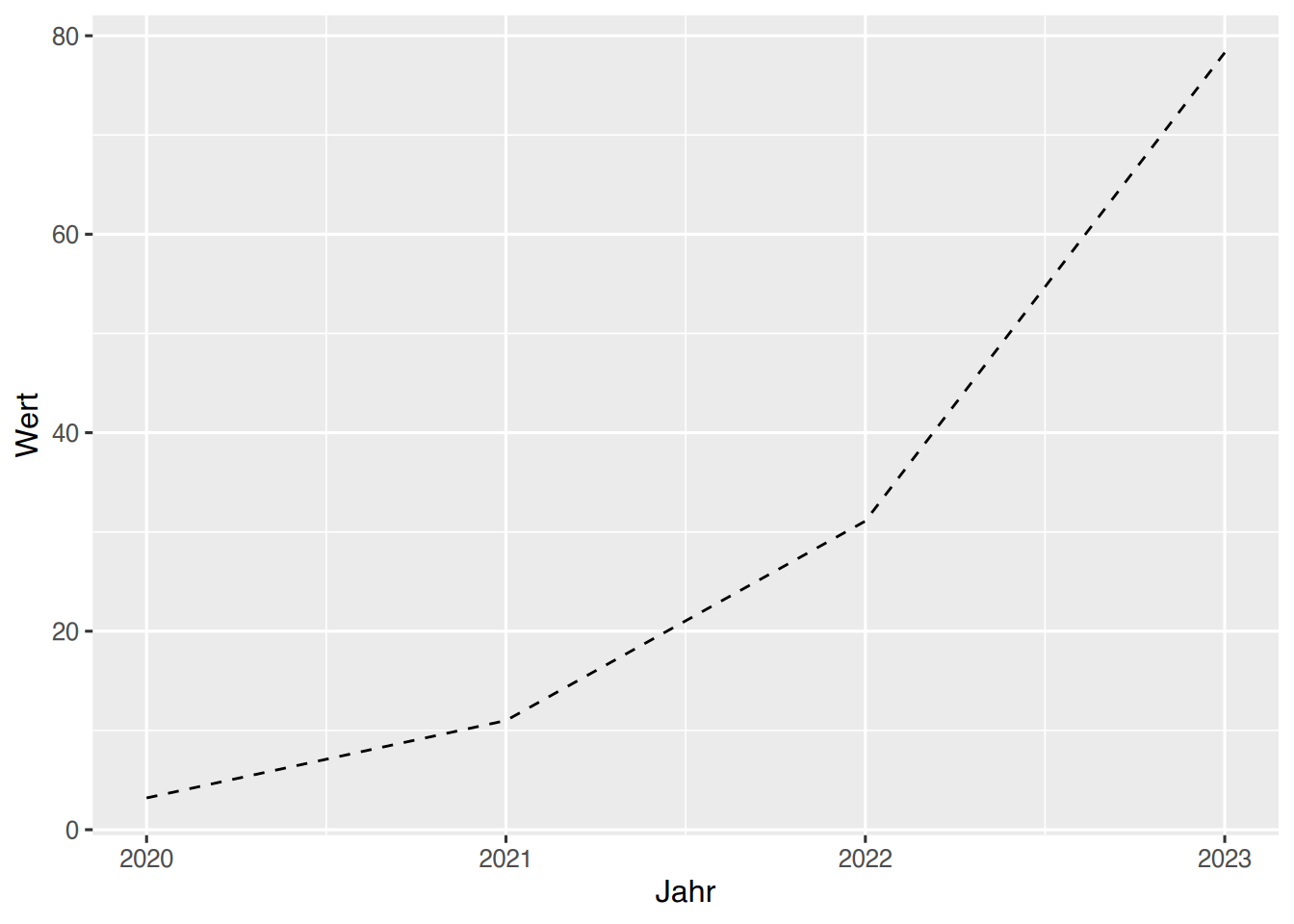
Sollen zusätzlich die Datenpunkte eingezeichnet werden (vgl. plot(type="b")), kann einfach das geom_point() angehängt werden.
ggplot(df, aes(x=Jahr, y=Wert)) +
geom_line(linetype = "dashed")+
geom_point()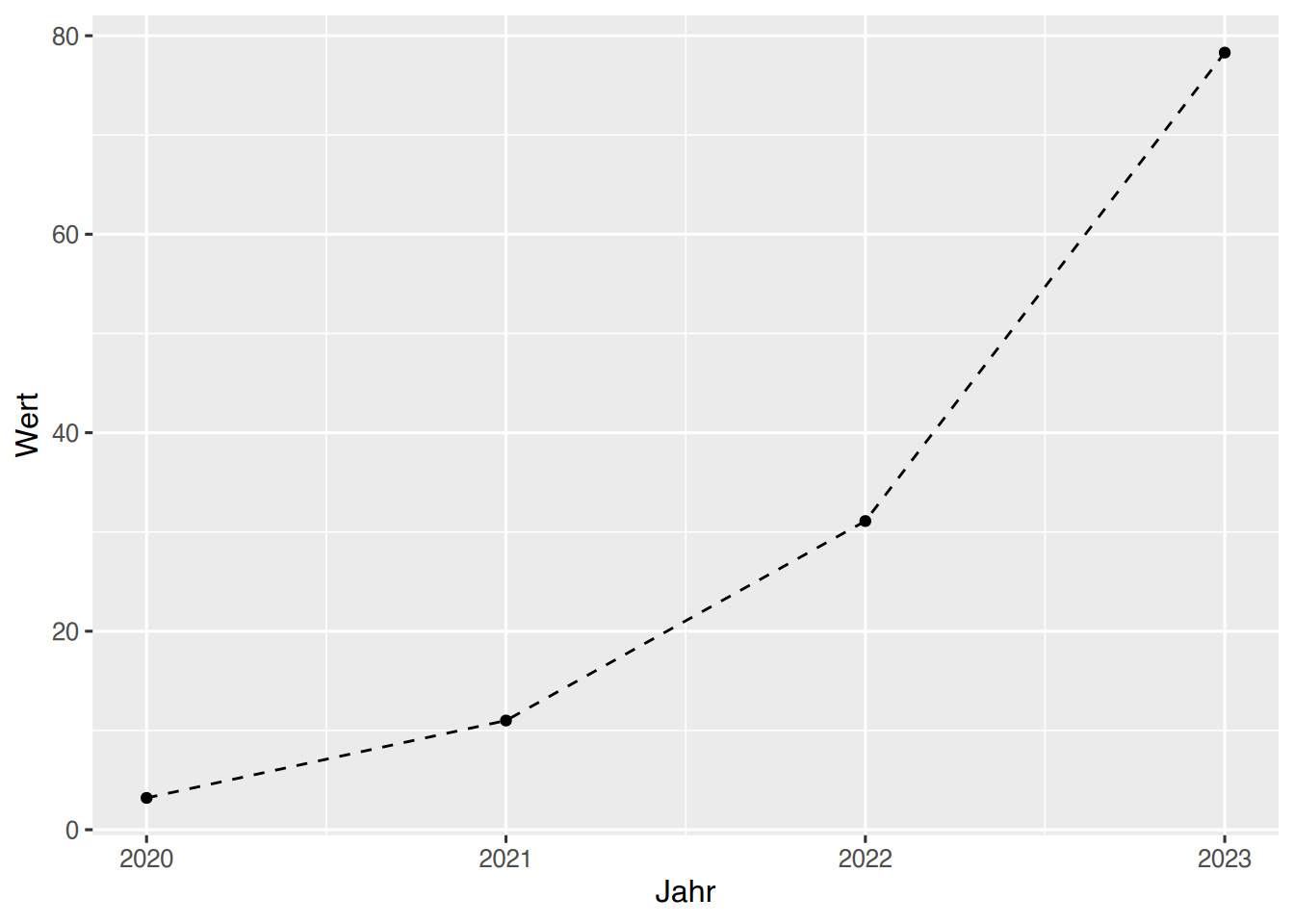
Farben können entweder per aes() oder direkt per col= gesetzt werden.
ggplot(df, aes(x=Jahr, y=Wert)) +
geom_line(color="red")+
geom_point(color="darkblue")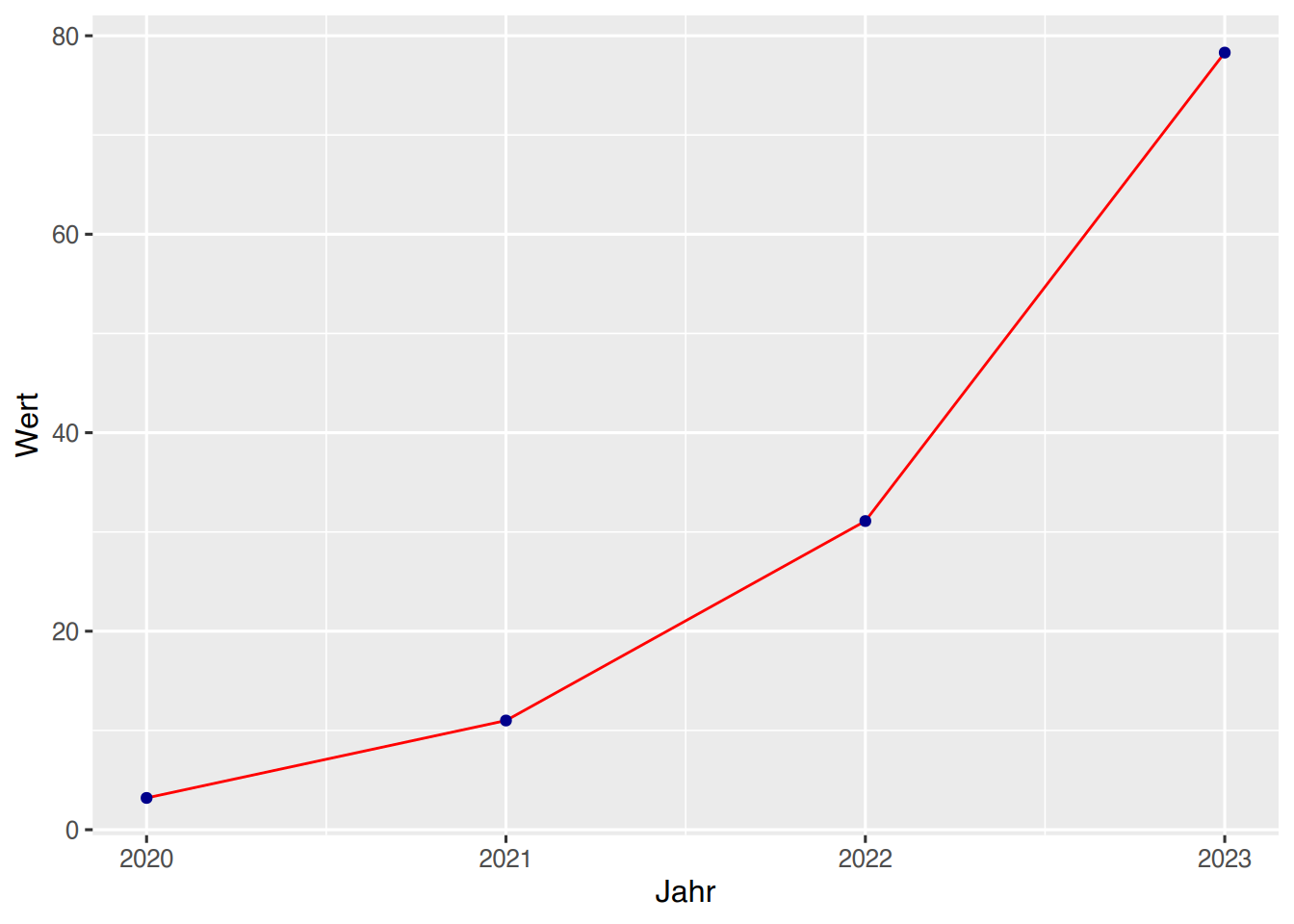
Liegen gruppierte Daten vor, kann die Farbe der Gruppen per aes() übergeben werden.
# erzeuge gruppierte Daten
df <- data.frame(Jahr=rep(c(2020:2023), 2),
Wert=c(3.2, 11, 31.1, 78.3,
4.1, 21, 53.5, 120),
Person=c(rep("Erika", 4), rep("Hans", 4)))
# Plotte mit unterschiedlichen Farben
ggplot(df, aes(x=Jahr, y=Wert, color=Person)) +
geom_line()+
geom_point()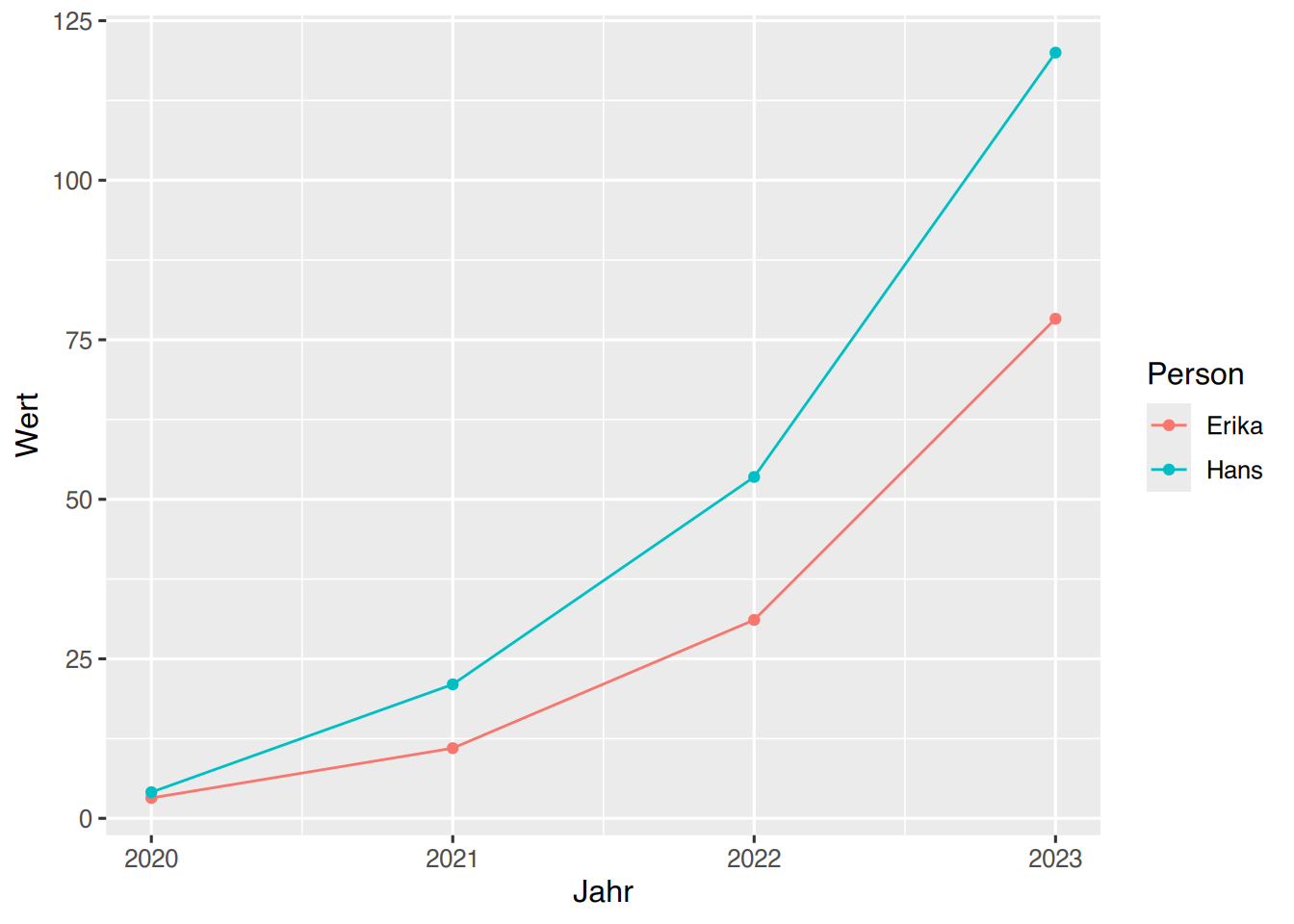
Dies funktioniert nicht nur mit Farben, sondern auch mit allen anderen Parametern.
ggplot(df, aes(x=Jahr, y=Wert, color=Person,
linetype=Person)) +
geom_line()+
geom_point()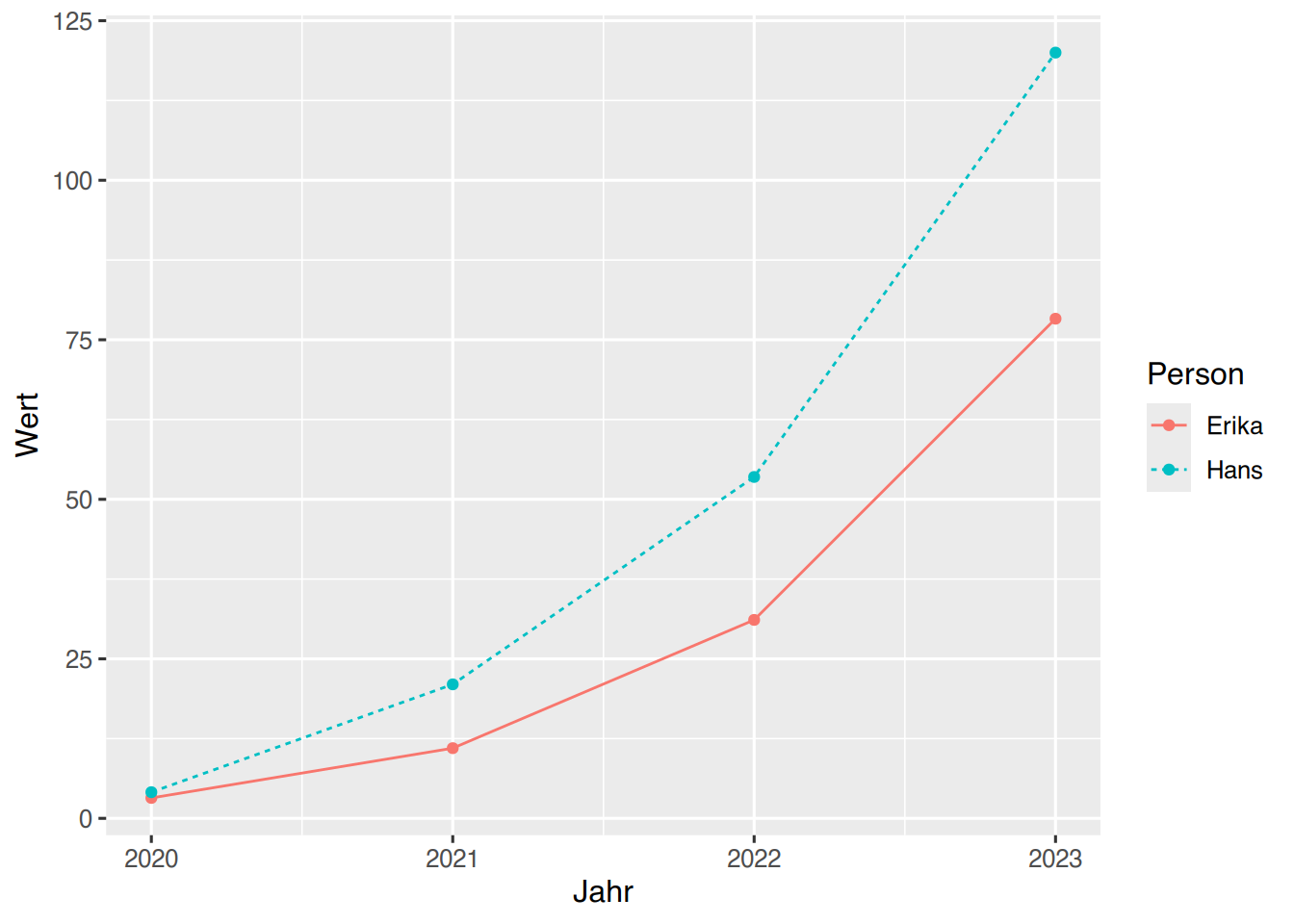
Innerhalb der Geome können die Parameter überschrieben werden, z.B. die Geom-Farbe.
ggplot(df, aes(x=Jahr, y=Wert, color=Person,
linetype=Person)) +
geom_line()+
geom_point(color="black")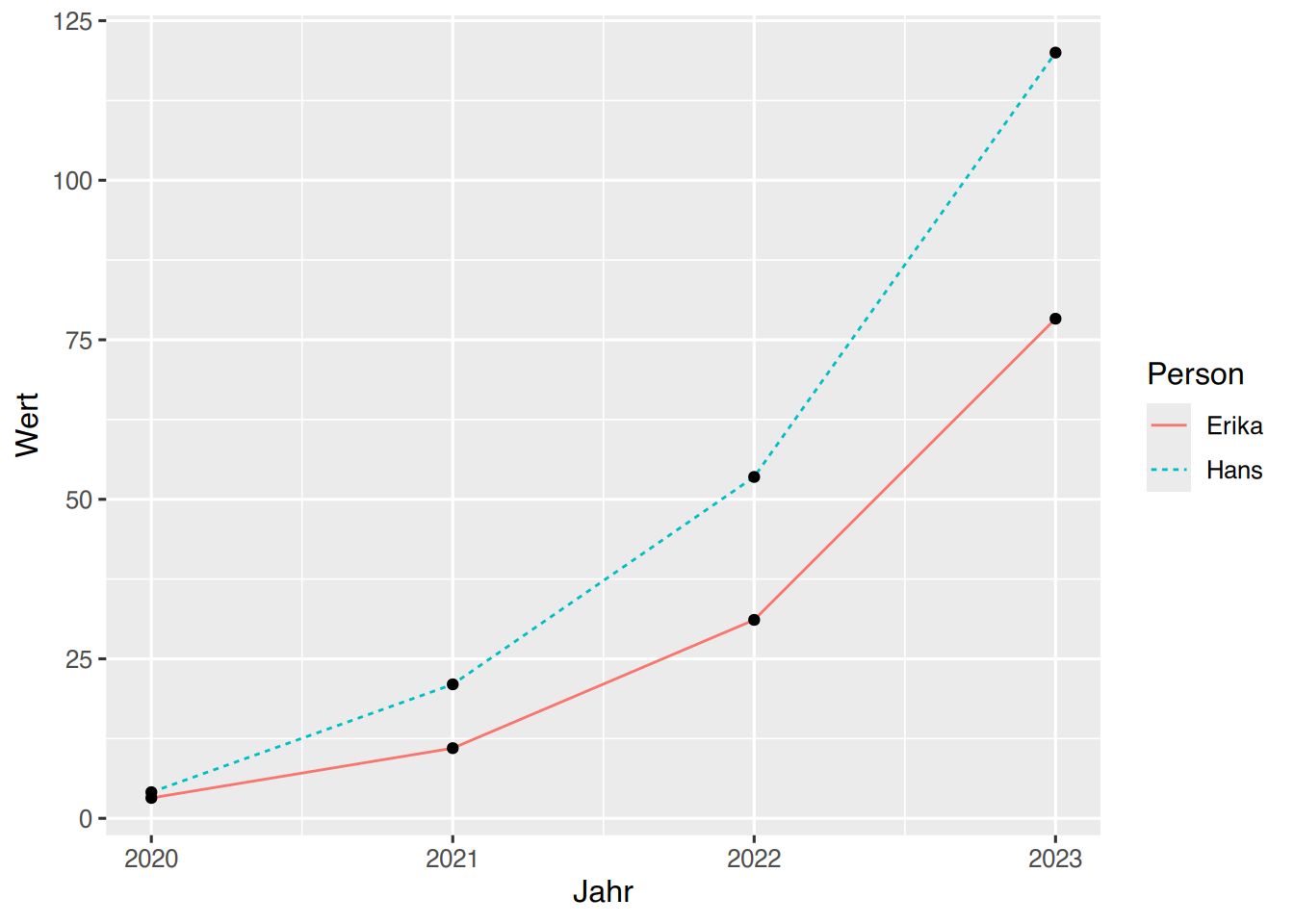
35.5 Histogramm
Histogramme werden mit der Funktion geom_histogram() erzeugt.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram()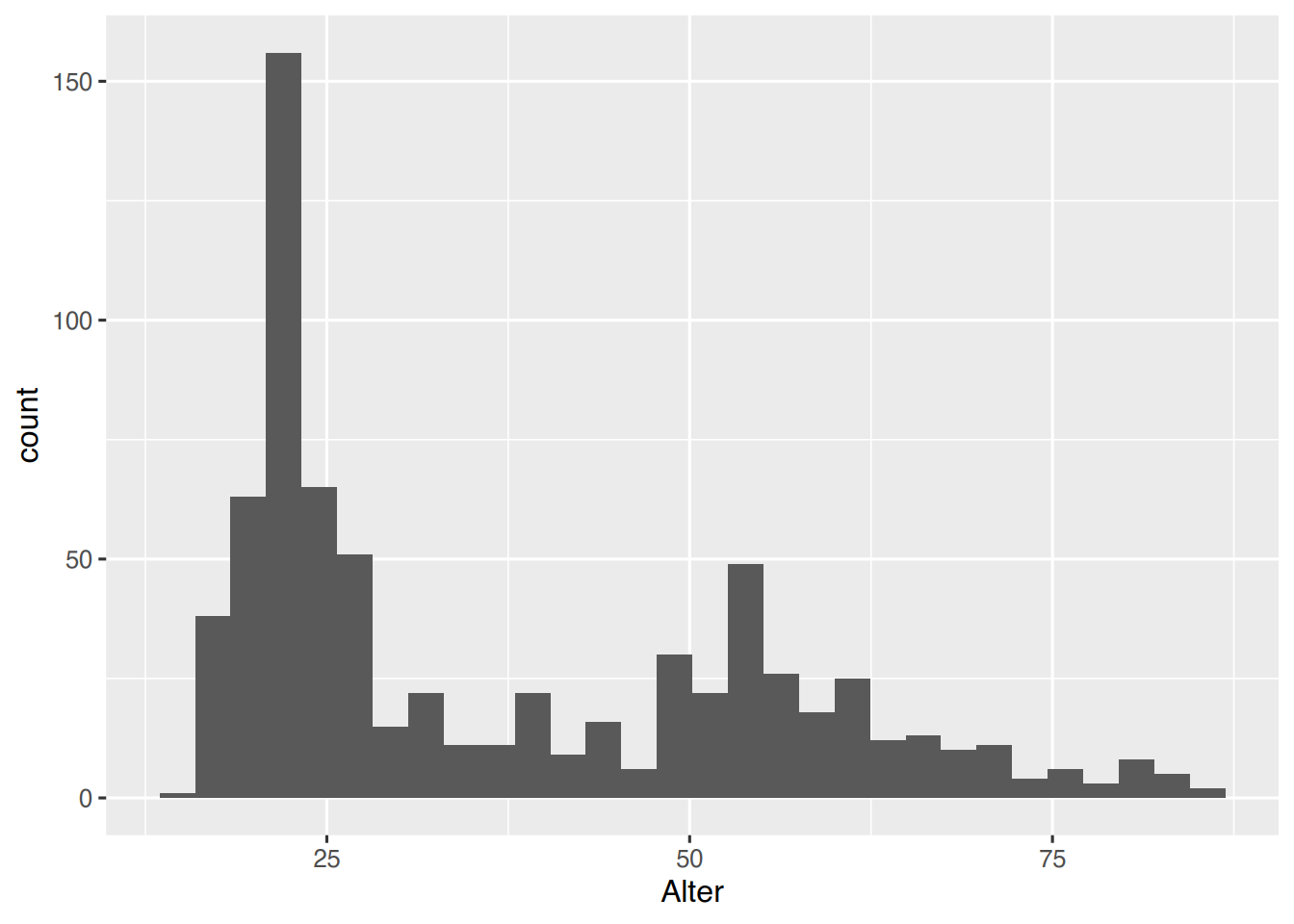
Mit den Parametern color und fill können wir das Histogramm etwas aufhübschen.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue", # Rahmenfarbe
fill="skyblue") # Füllfarbe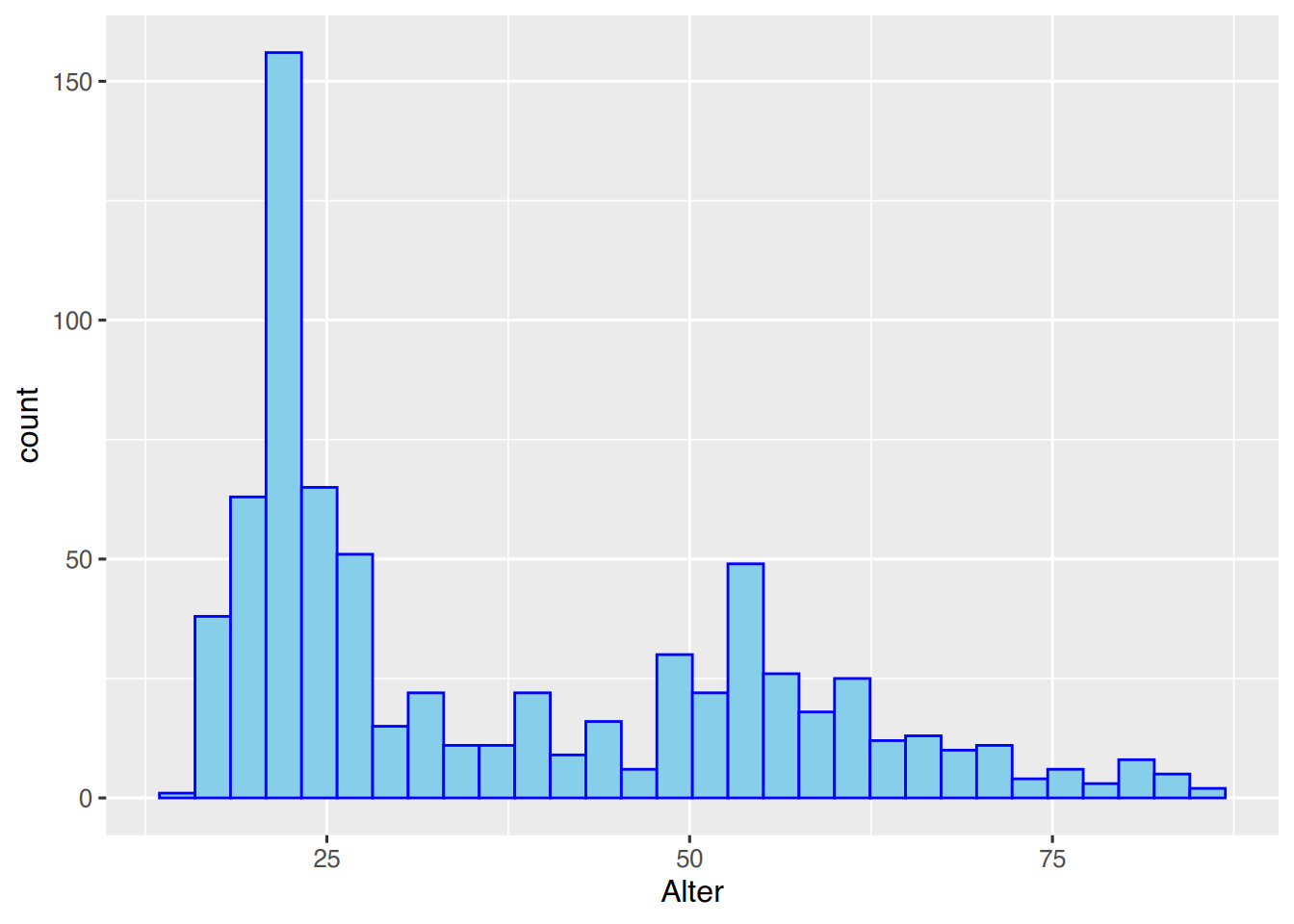
Über den Parameter binwidth kann die Säulenbreite bestimmt werden.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue", # Rahmenfarbe
fill="skyblue", # Füllfarbe
binwidth=10) # Klassenbreite 10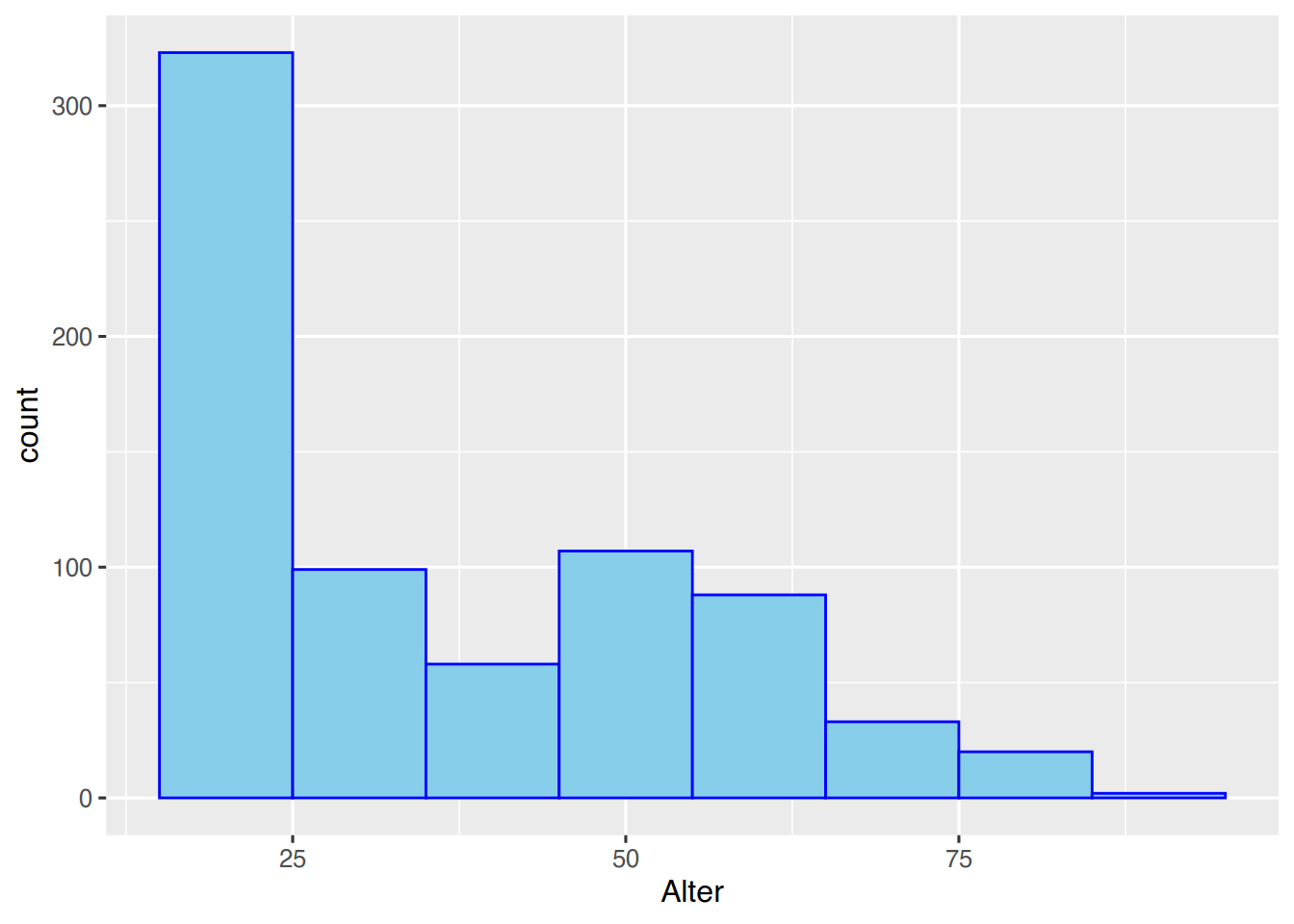
Mit dem Parameter bins kann die Anzahl der Säulen bestimmt werden.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue", # Rahmenfarbe
fill="skyblue", # Füllfarbe
bins=5) # 5 Säulen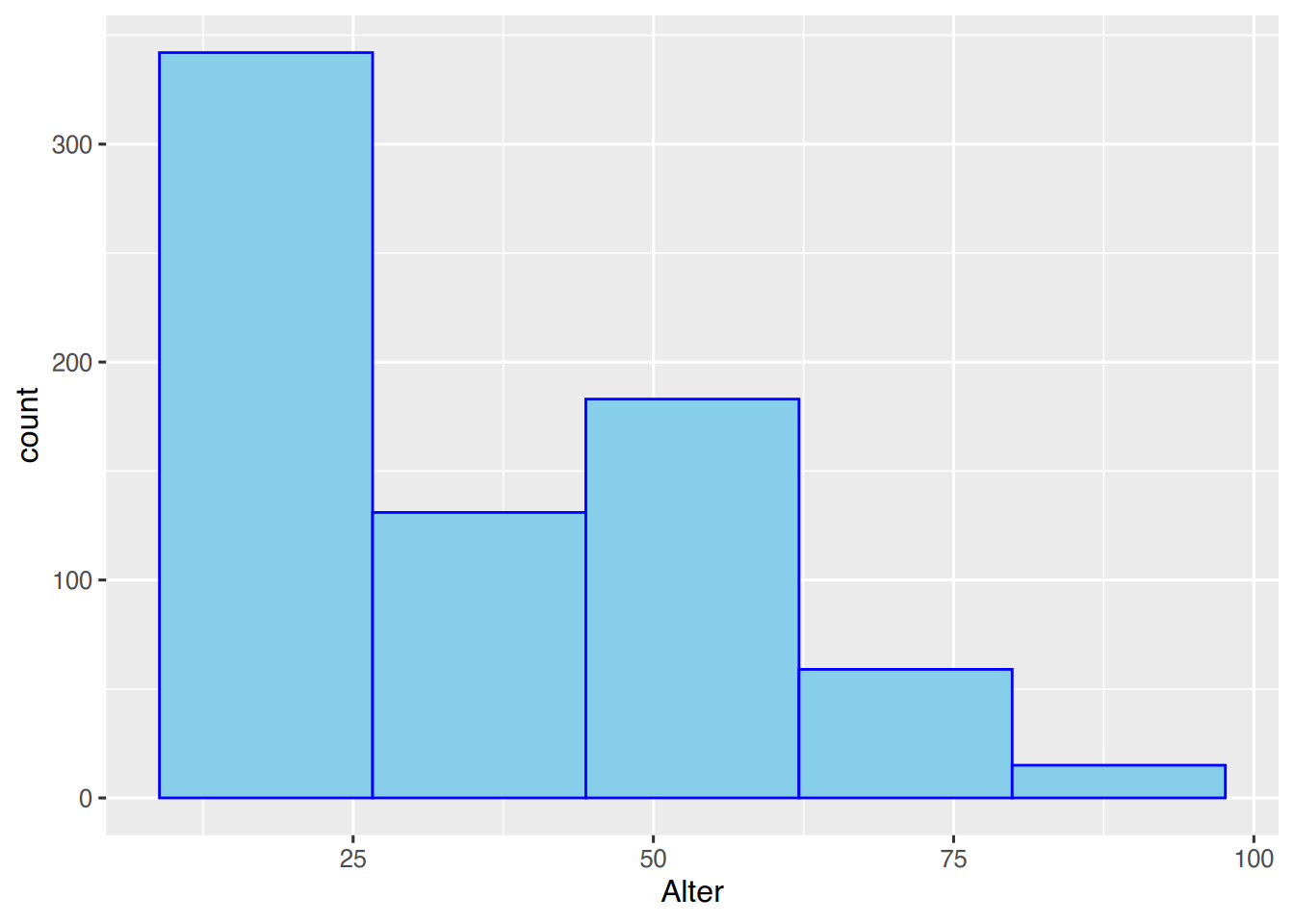
Mit der Funktion stat_bin() kann das “Originalhistogramm” hinzugefügt werden. So ist leicht zu erkennen, wie grob die Klassen zusammengefasst wurden.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue", # Rahmenfarbe
fill="skyblue", # Füllfarbe
bins=5) + # 5 Säulen
stat_bin(fill="darkblue")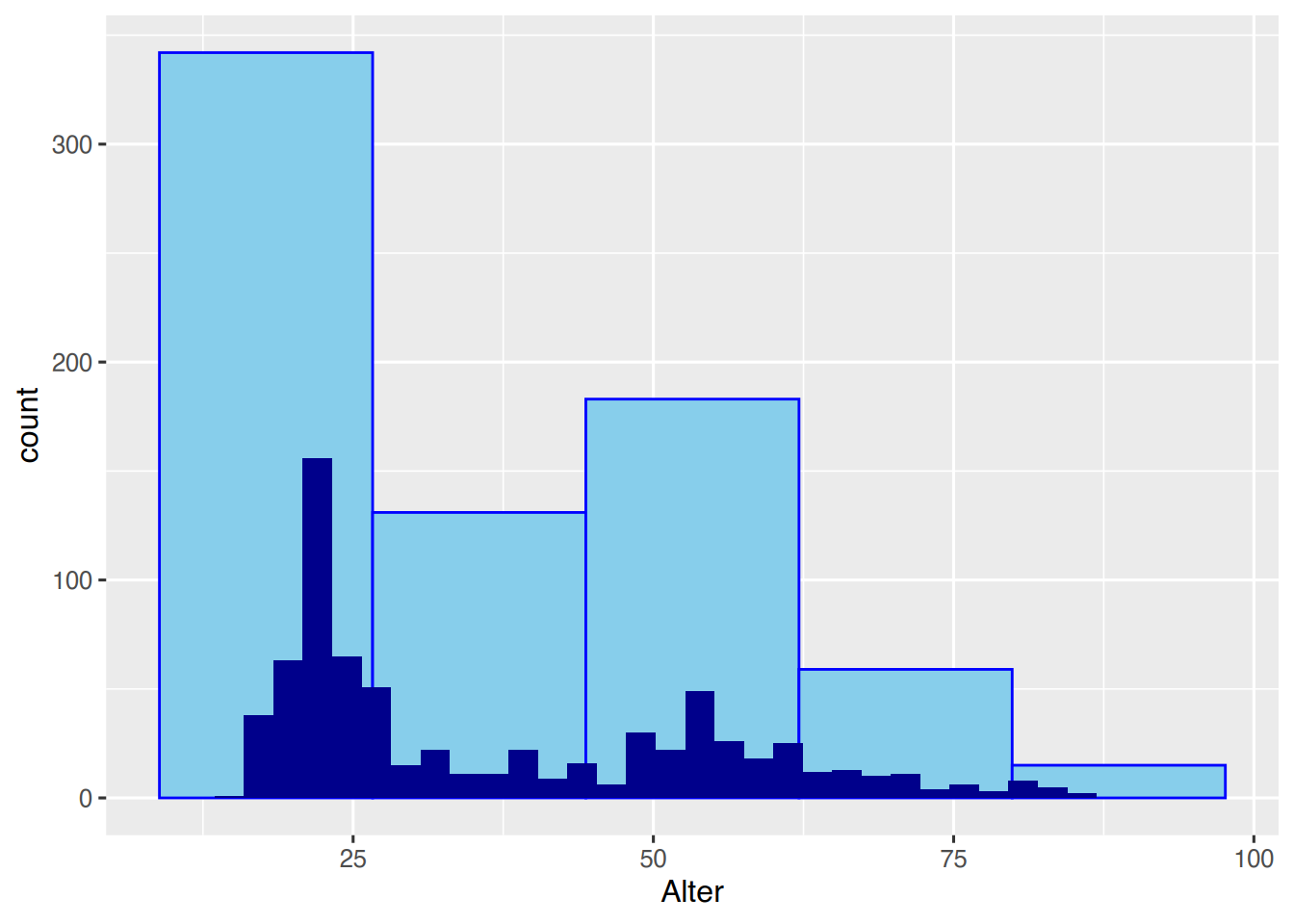
35.5.1 Histogramm und Dichteverteilung
Über die Aesthetic ..density.. können wir auf die Skala der Dichtefunktion wechseln.
ggplot(pf8) +
aes(x=Alter,
y=..density..) +
geom_histogram(color="blue",
fill="skyblue")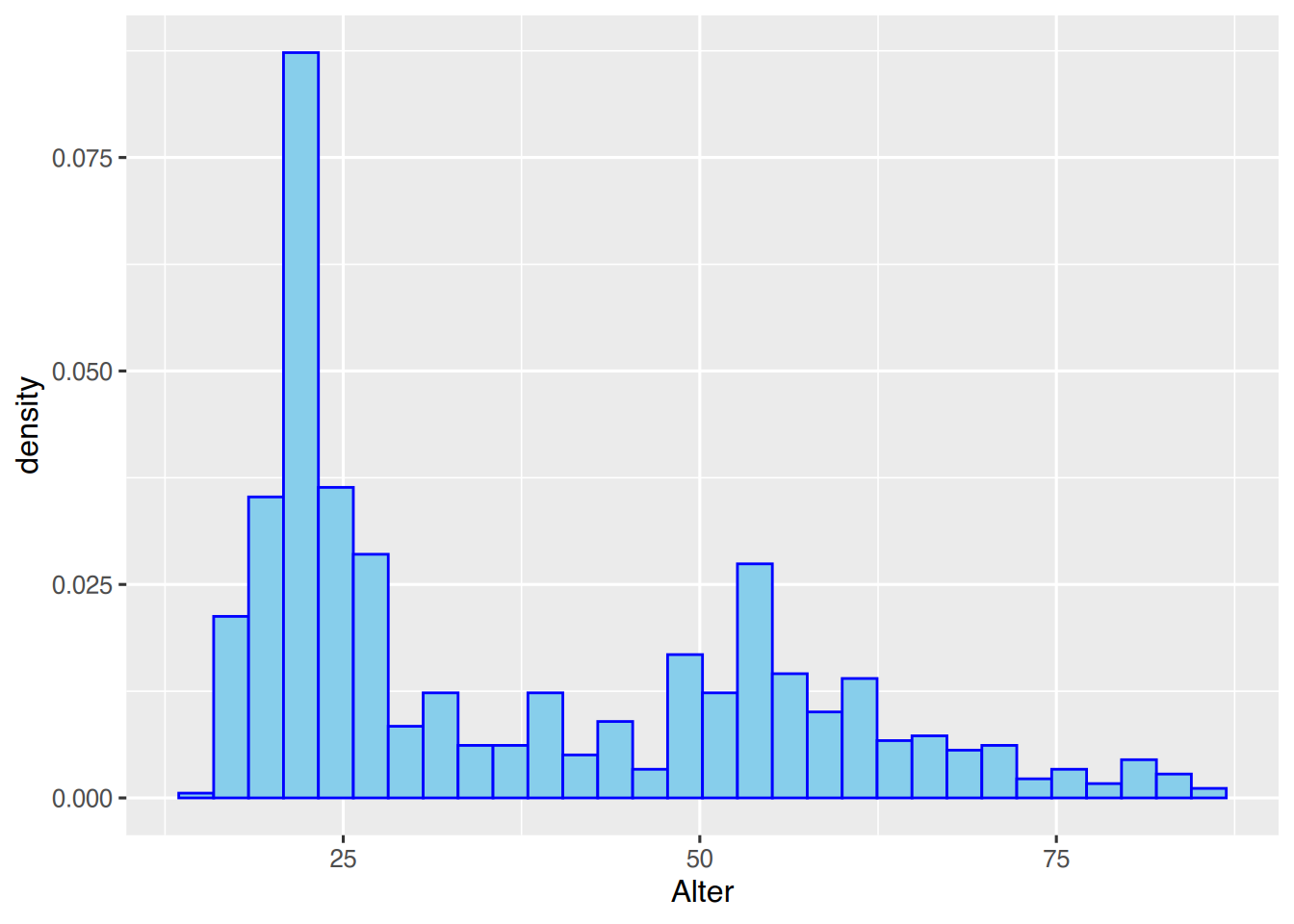
Beachten Sie die Achseneinträge der y-Achse.
Das funktioniert auch, wenn die aesthetics im geom_histogram() angegeben werden.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(aes(y=..density..),
color="blue",
fill="skyblue")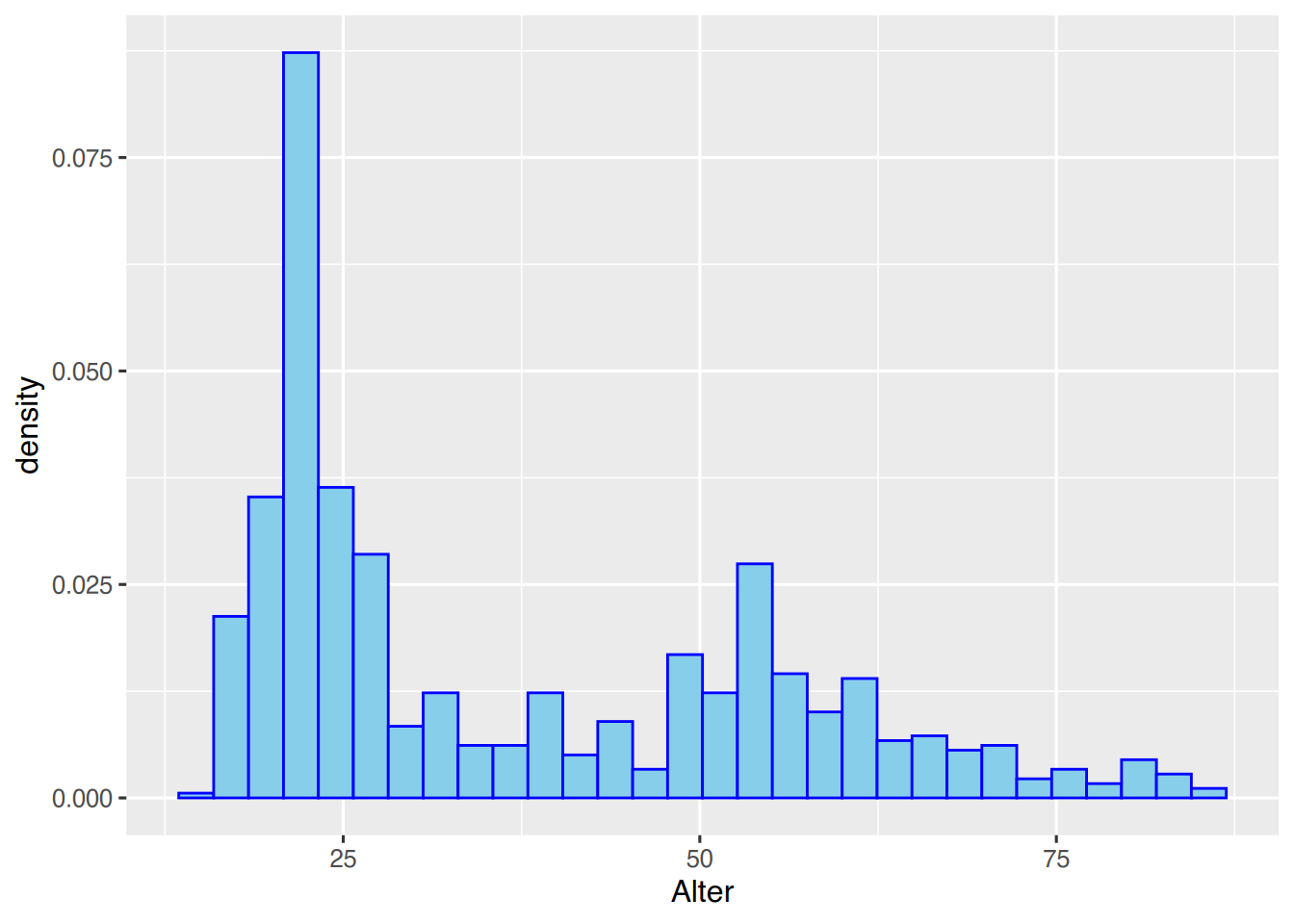
Die Dichtefunktion des Histogramms lässt sich nun mit der Funktion stat_density() einzeichnen.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(aes(y=..density..),
color="blue",
fill="skyblue") +
stat_density(geom="line",
color="red",
linetype="dotted")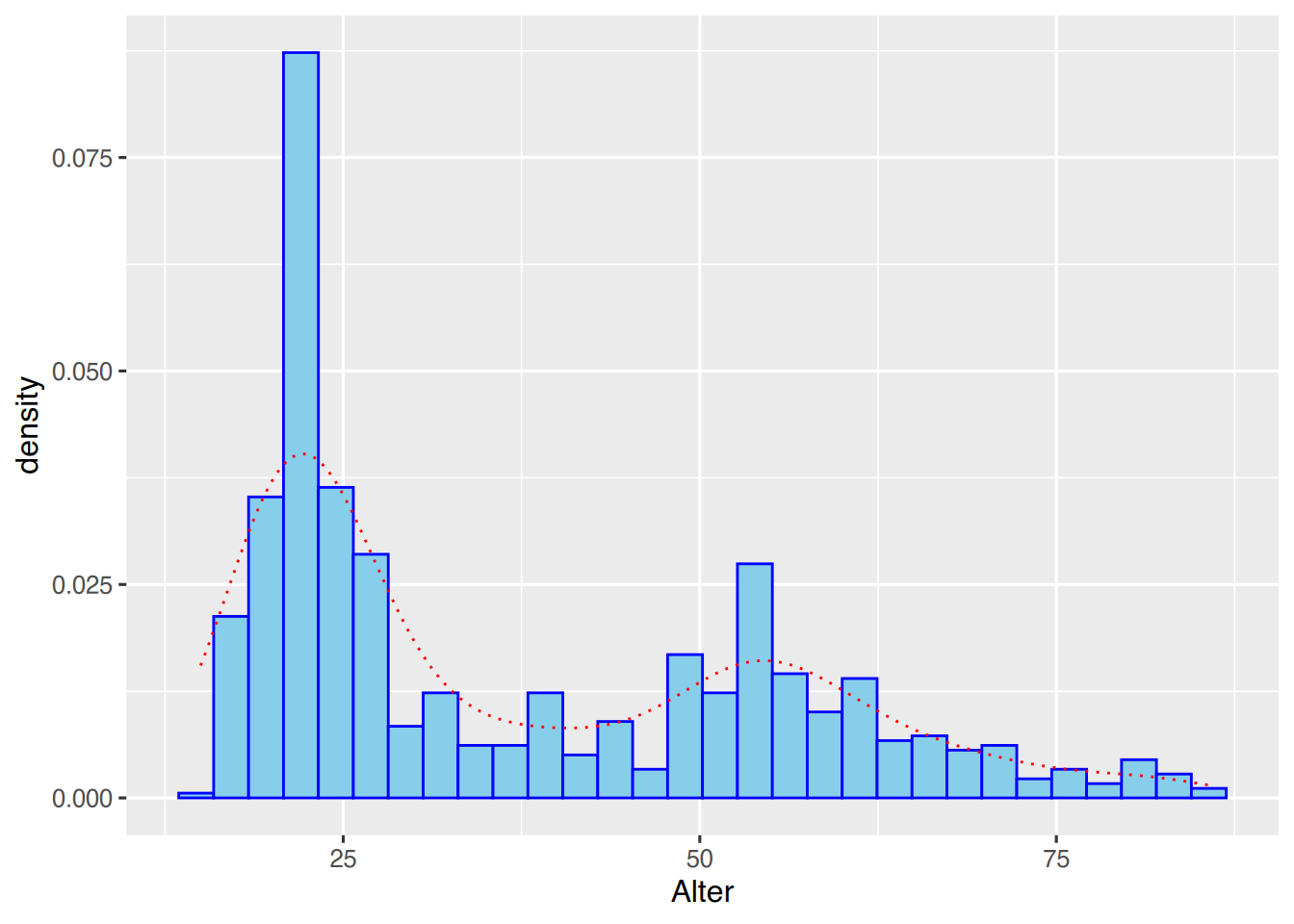
Wir wechseln von geom="line" auf geom="area":
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(aes(y=..density..),
color="blue",
fill="skyblue") +
stat_density(geom="area",
color="red",
fill="pink",
linetype="dotted")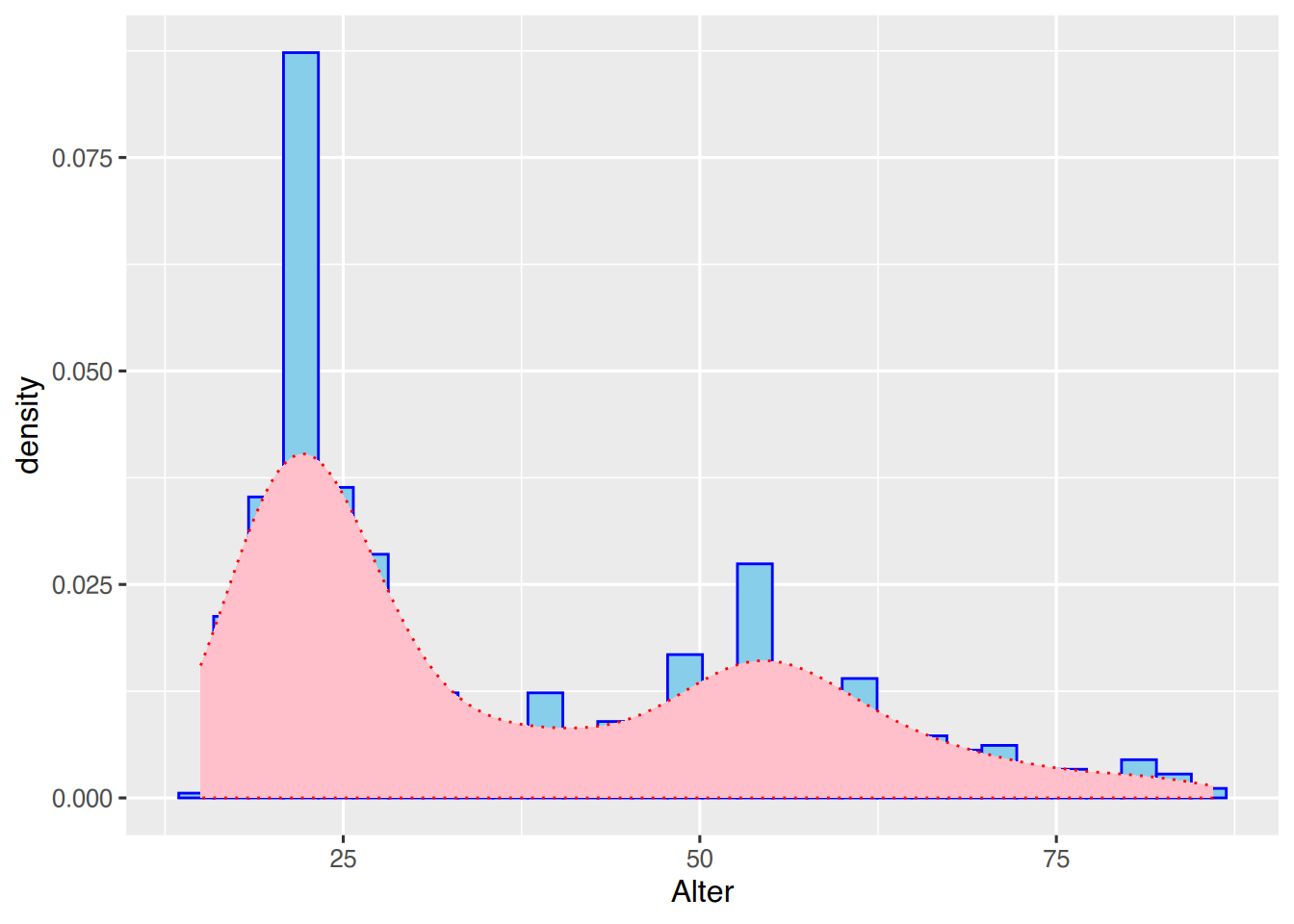
Über den Paramter alpha lässt sich die Durchsichtigkeit (so genannter Alphakanal) des Objekts eingeben.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(aes(y=..density..),
color="blue",
fill="skyblue") +
stat_density(geom="area",
color="red",
fill="pink",
linetype="dotted",
alpha=0.5)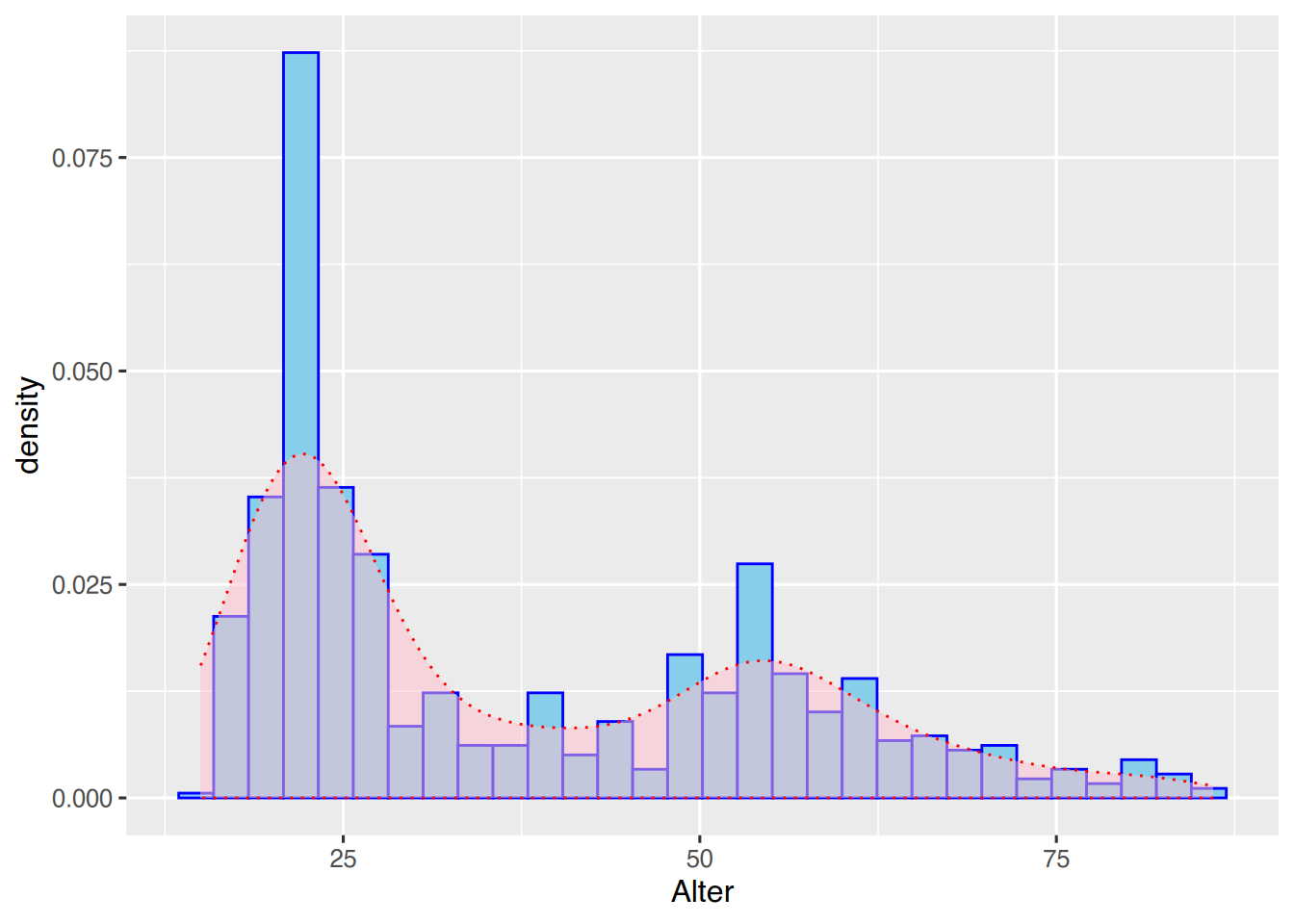
Mittels stat_function() und dnorm() lässt sich die Normalverteilung ergänzen. Hierfür bestimmen wir zunächst Mittelwert und Standardabweichung von pf8$Alter.
round(mean(pf8$Alter, na.rm=TRUE))## [1] 37round(sd(pf8$Alter, na.rm=TRUE))## [1] 18Die Werte werden nun wie folgt übertragen:
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(aes(y=..density..),
color="blue",
fill="skyblue") +
stat_density(geom="area",
color="red",
fill="pink",
linetype="dotted",
alpha=0.5)+
stat_function(fun=dnorm,
args=(c(mean=37,sd=18)),
color = "darkgreen",
linetype = "dashed")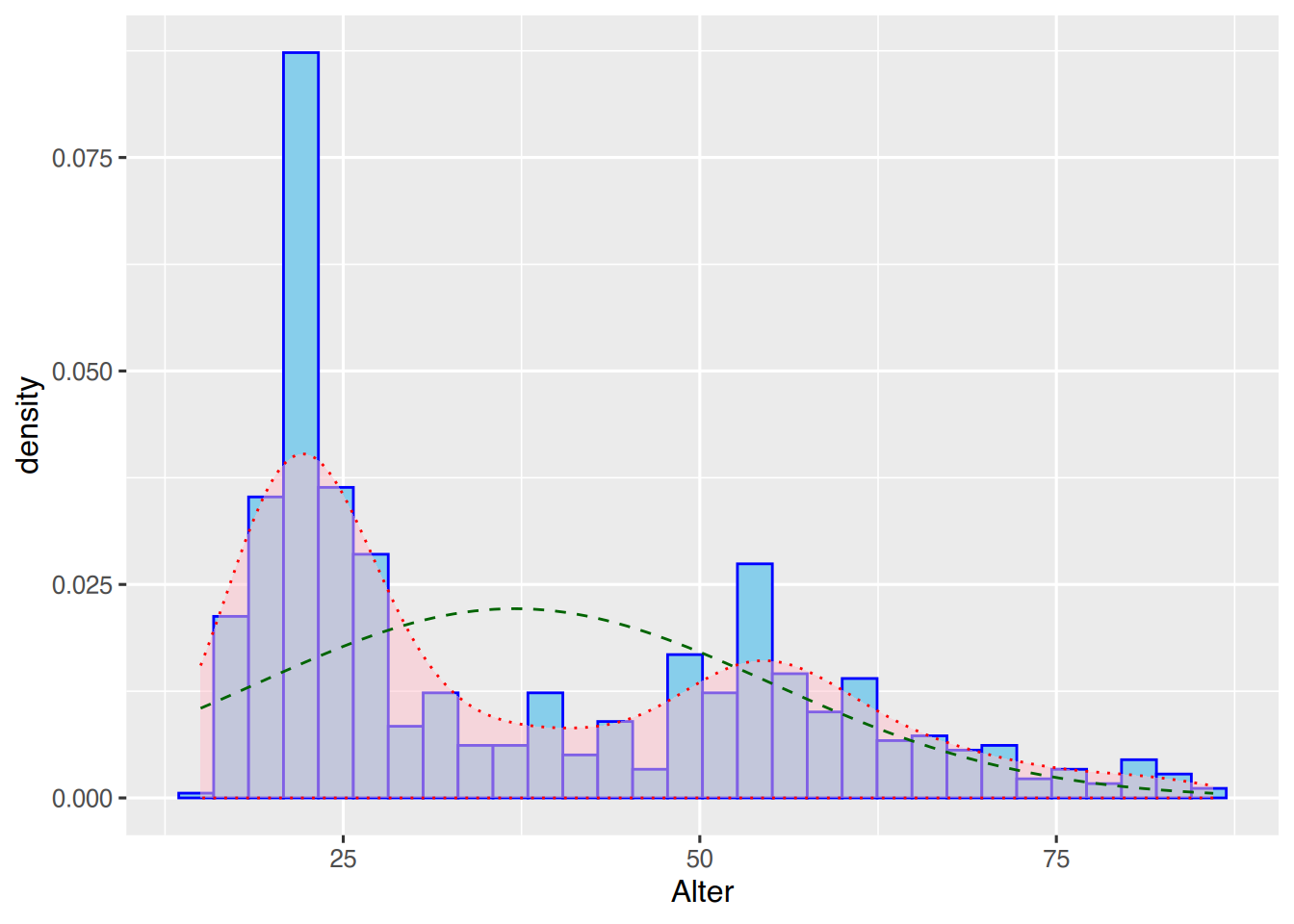
Es ist deutlich erkennbar, dass keine Normalverteilung vorliegt.
35.5.2 kumulierte Häufigkeiten
Für kumulierte Häufigkeiten nutzen wir innerhalb von aes() die Funktionen after_stat(count), um die Häufigkeiten entsprechen mittels cumsum() und sum() zu manipulieren.
# kumulierte absolute Häufigkeiten
ggplot(pf8) +
aes(x=Alter) + ylab("absolute Häufigkeiten") +
geom_histogram(aes(y=cumsum(after_stat(count))),
bins=5, fill="hotpink3", color="black") +
# füge Polygonzug hinzu
stat_bin(aes(y=cumsum(after_stat(count))),
bins=5,
geom="line", color="blue", linewidth=1.5) # oder geom="step"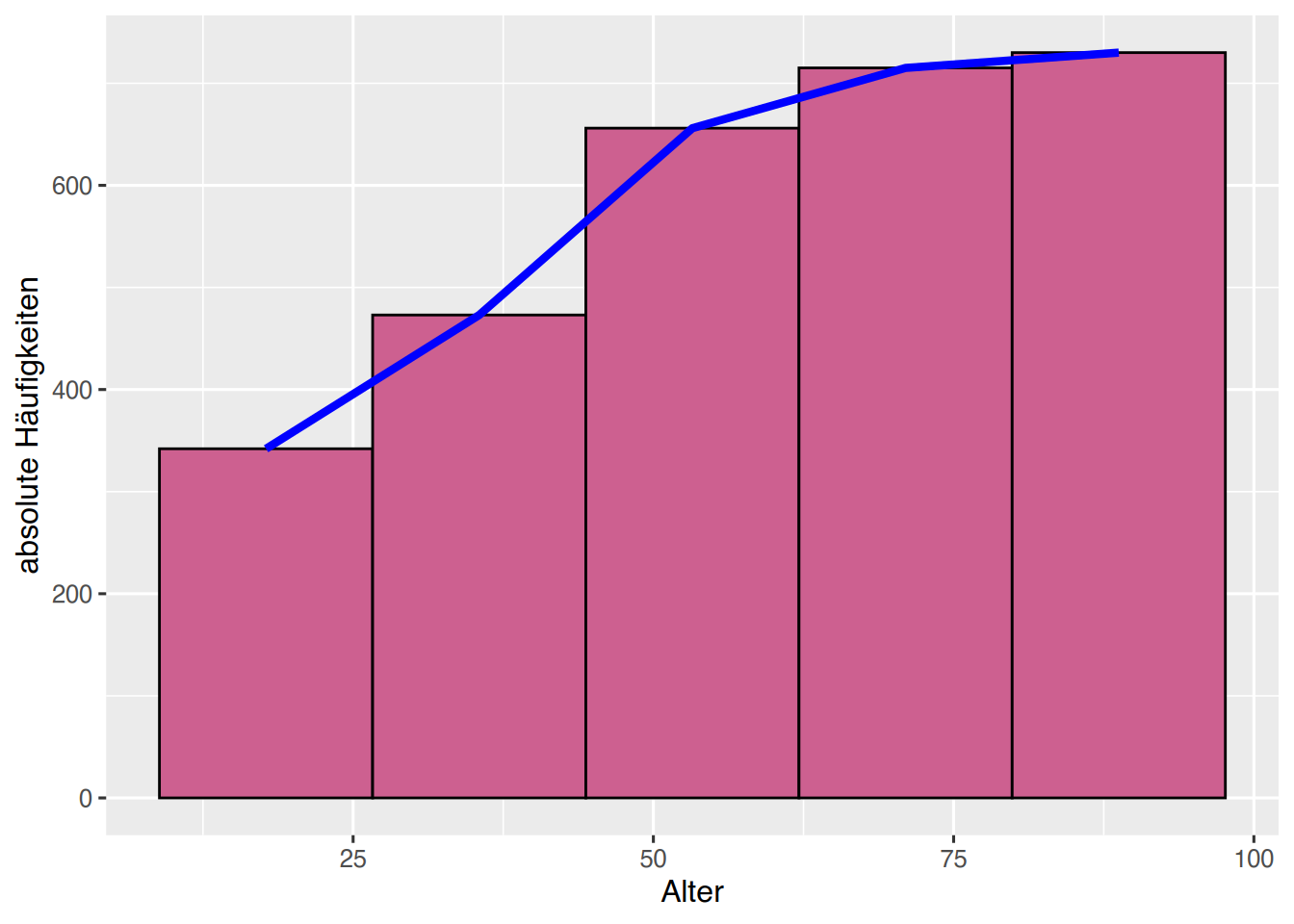
# kumulierte relative Häufigkeiten
ggplot(pf8) +
aes(x=Alter) +
ylab("relative Häufigkeiten")+
geom_histogram(aes(y=cumsum(after_stat(count)/sum(after_stat(count)))),
bins=5, fill="hotpink3", color="black") +
# füge Polygonzug hinzu
stat_bin(aes(y=cumsum(after_stat(count)/sum(after_stat(count)))),
bins=5,
geom="step", color="blue", linewidth=1.5) # oder geom="line"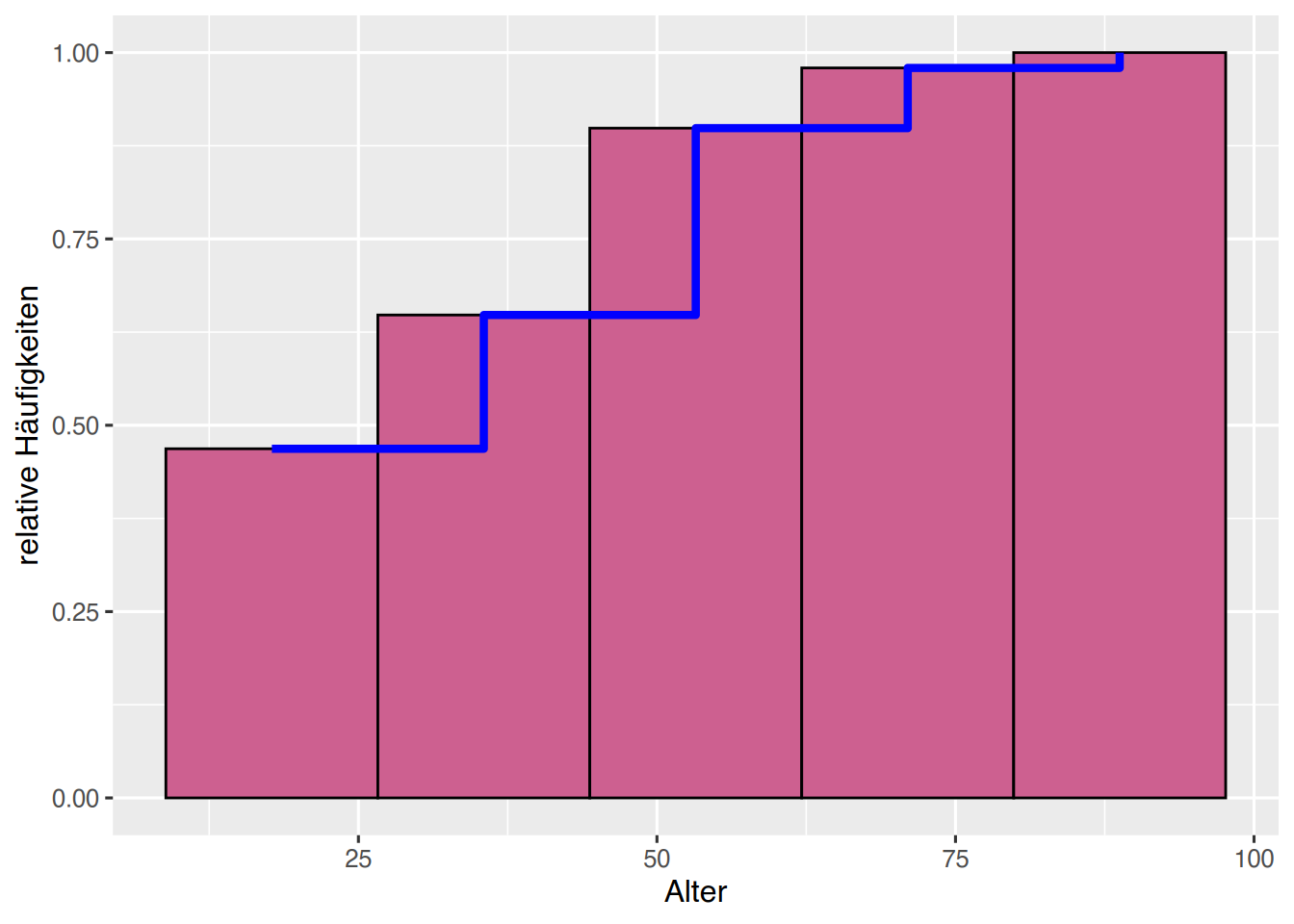
35.6 Säulendiagramme
Säulendiagramme werden mit den Funktionen geom_bar() und geom_col() erzeugt.
- Für
geom_bar()müssen die Variablen als Factor vorliegen. Die Größe der Säulen wird automatisch mittelsstat_count()berechnet. geom_col()erstellt die Größe der Säulen anhand der tatsächlichen Werte im Datenframe (vgl.stat_identity(); absolute Häufigkeit)
Die Variable pf8$SportWarum ist ein Faktor und soll mittels geom_bar() geplottet werden.
ggplot(pf8) +
aes(x=SportWarum ) +
geom_bar(color="blue",
fill="skyblue")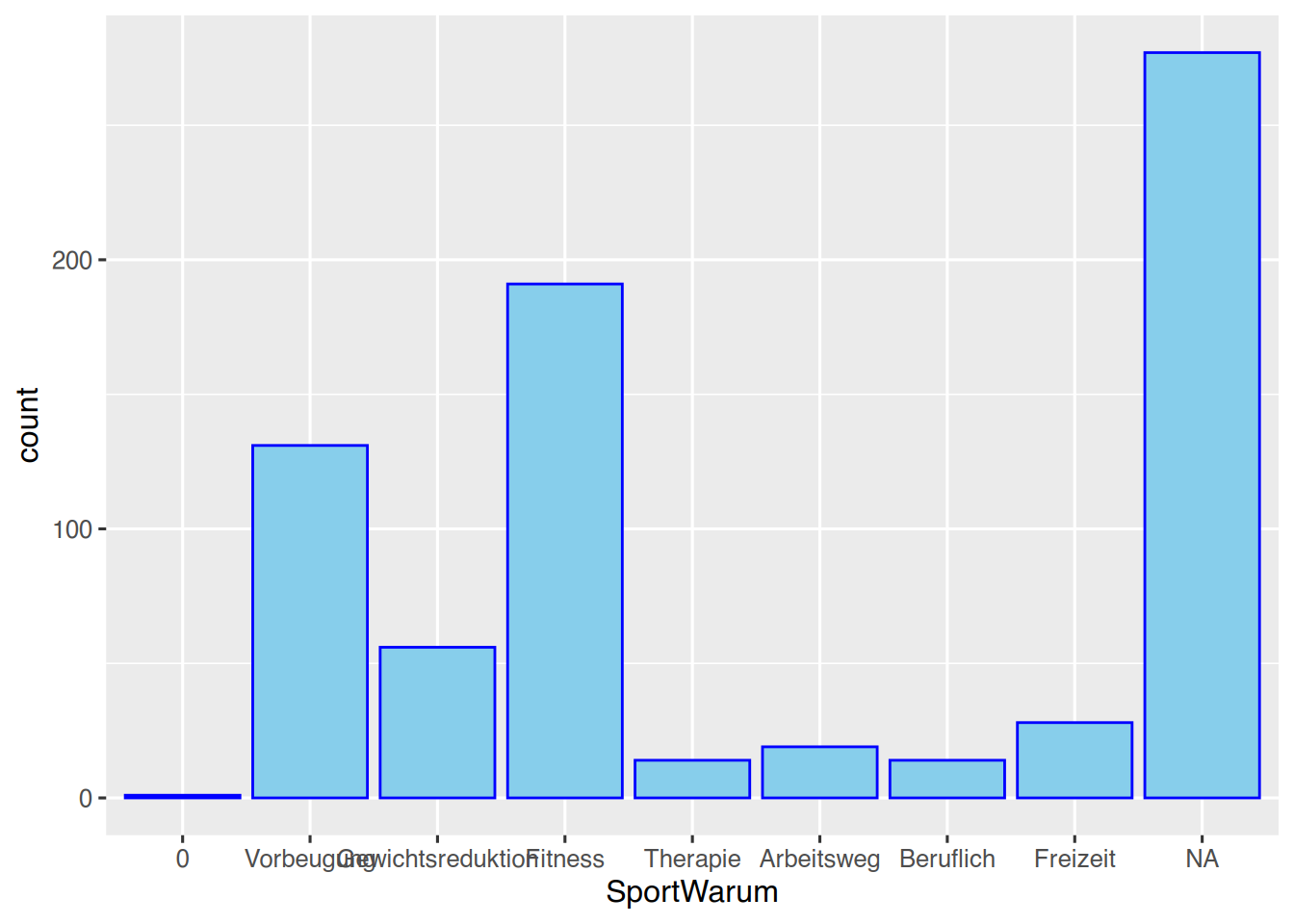
Mittels drop_na() kann der große NA-Balken entfernt werden.
pf8 %>%
drop_na(SportWarum) %>%
ggplot() +
aes(x=SportWarum ) +
geom_bar(color="blue",
fill="skyblue")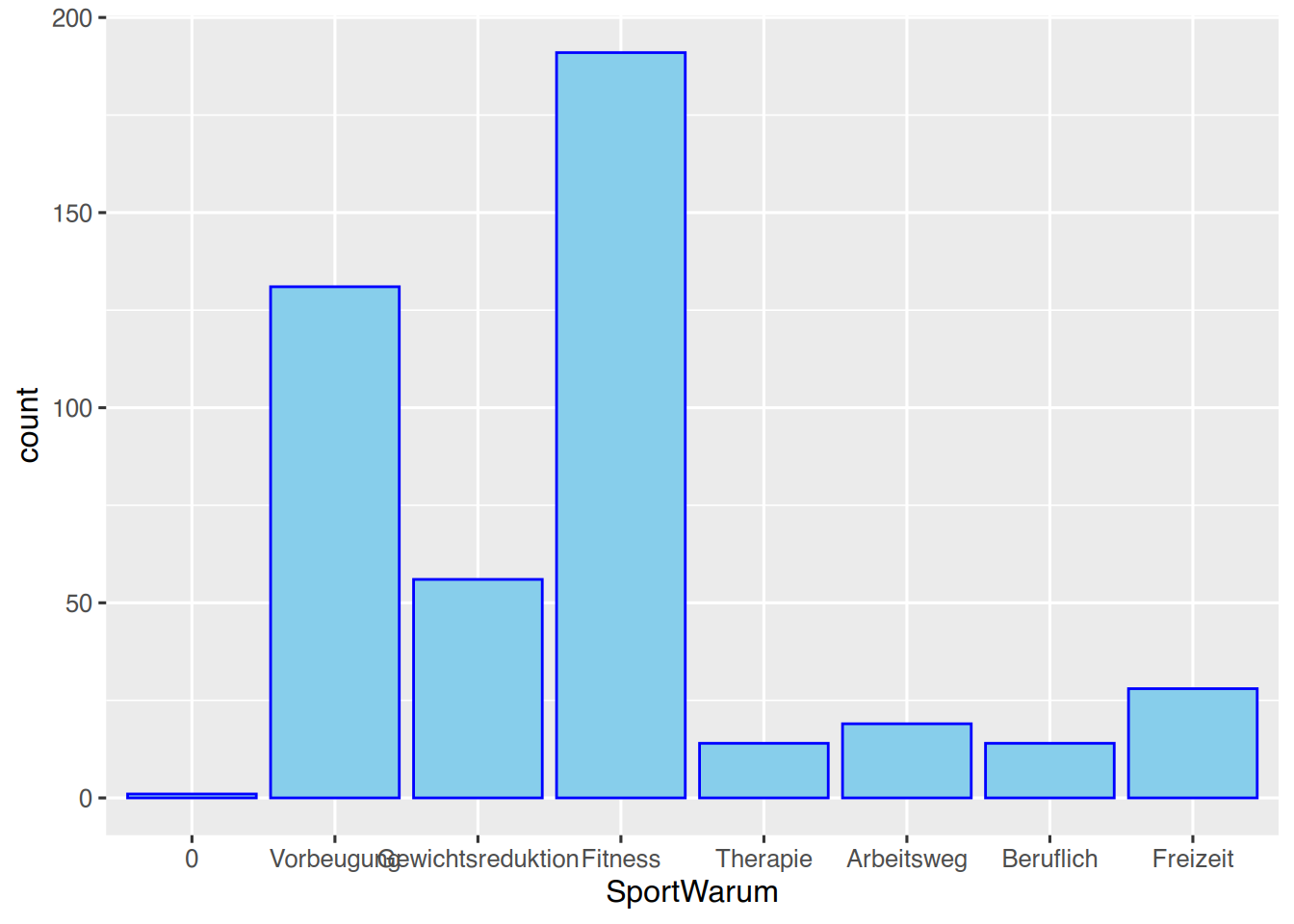
Mit Hilfe von der Funktionen fct_infreq() und fct_rev() können wir die Levels auf- und absteigend sortieren. Hierzu erzeugen wir eine neue Spalte S, welche wir in der aes()-Funktion auf die x-Achse legen.
# sortiere absteigend
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum)) %>%
ggplot() +
aes(x=S ) +
geom_bar(color="blue",
fill="skyblue")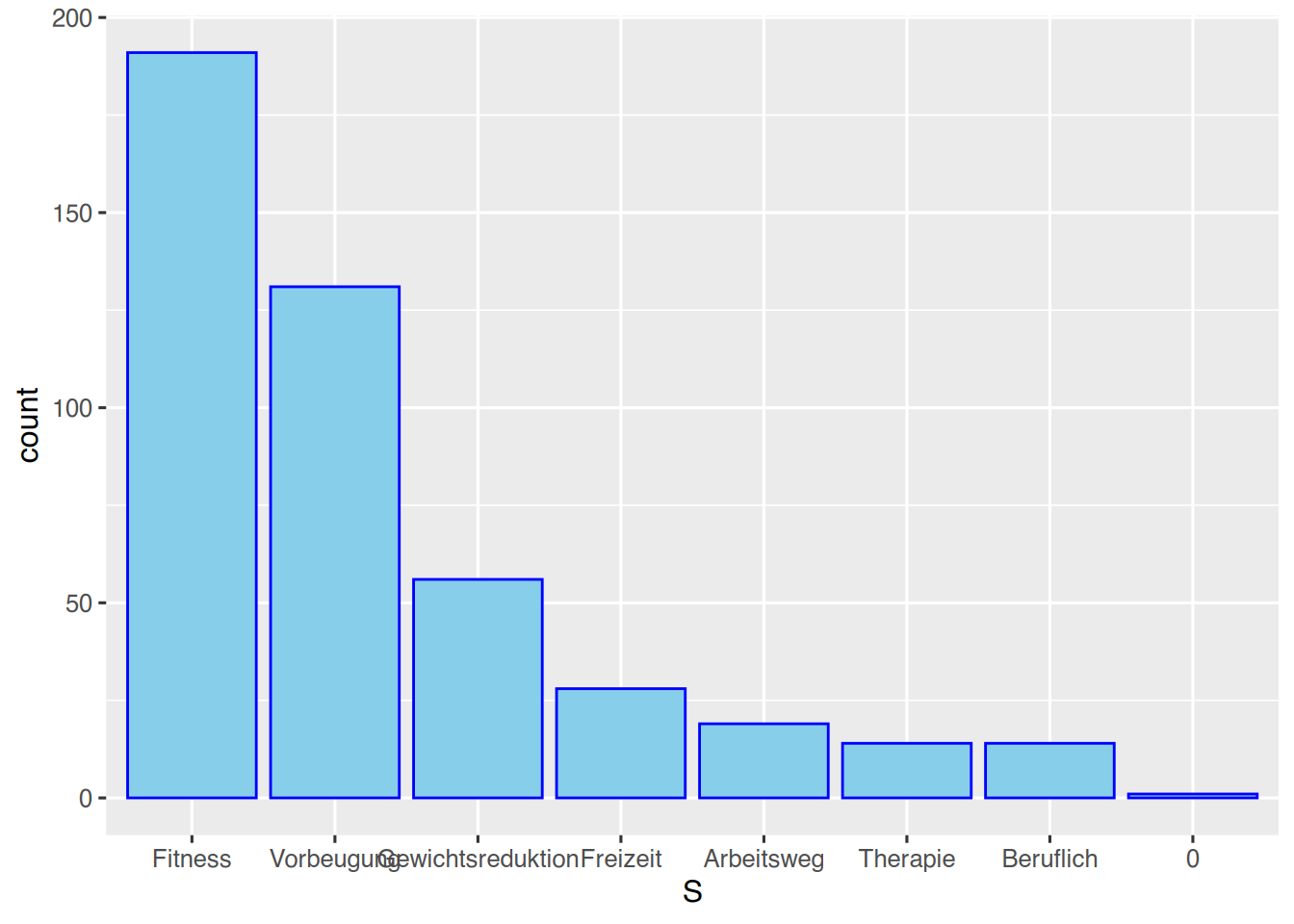
Um aufsteigend zu sortieren erweitert sich der mutate()-Aufruf wie folgt:
# sortiere aufsteigend
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum),
S = fct_rev(S)) %>%
ggplot() +
aes(x=S) +
geom_bar(color="blue",
fill="skyblue")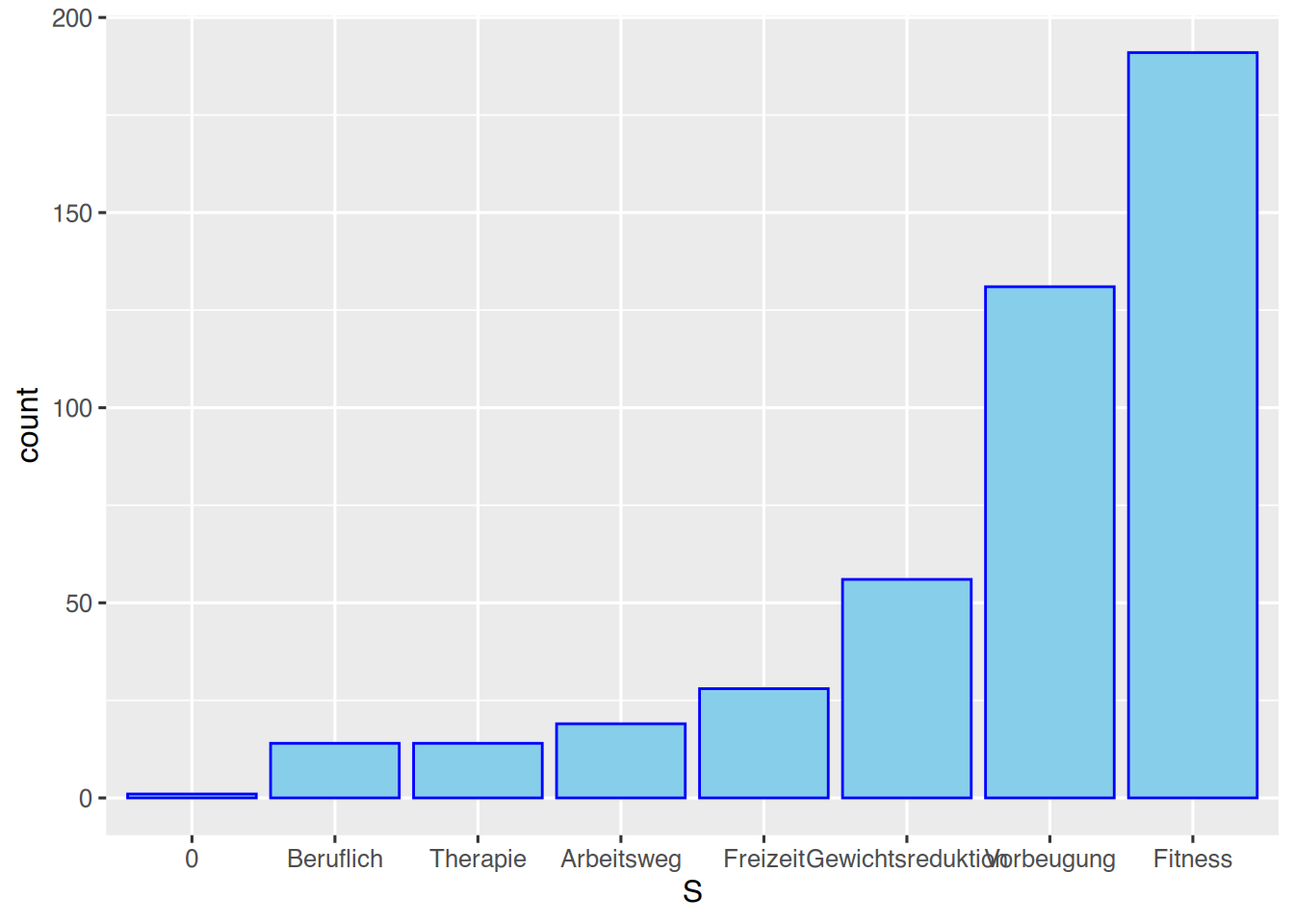
Über die Aesthetic fill können wir gruppieren, z.B. nach Geschlecht.
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum)) %>%
mutate(S = fct_rev(S)) %>%
ggplot() +
aes(x=S,
fill=Geschlecht) +
geom_bar(color="blue")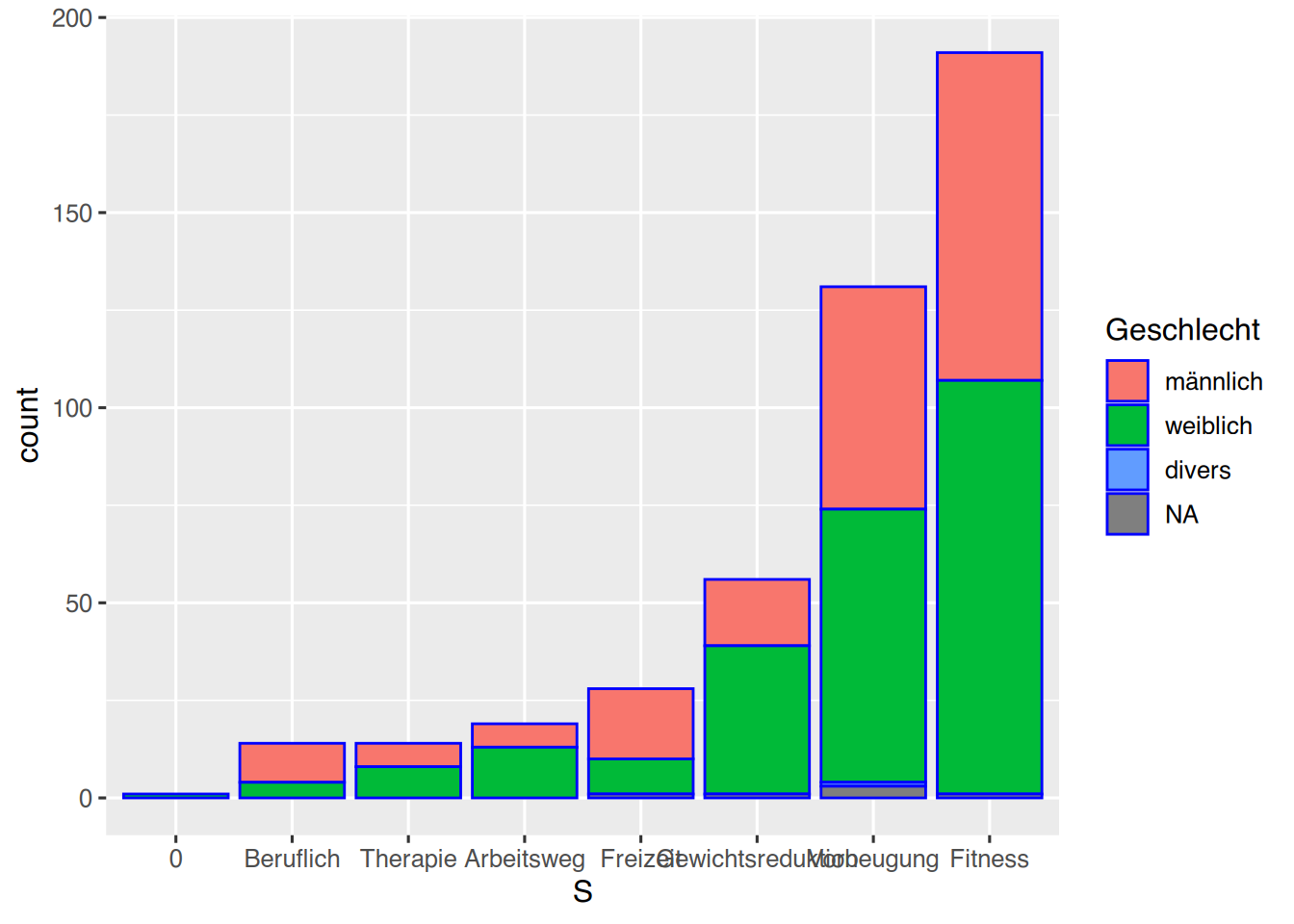
Mit dem Parameter position können wir die Säulenausgabe weiter beeinflussen. Die Angabe position="fill" erzeugt Prozentanteile.
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum)) %>%
mutate(S = fct_rev(S)) %>%
ggplot() +
aes(x=S,
fill=Geschlecht) +
geom_bar(color="blue",
position="fill")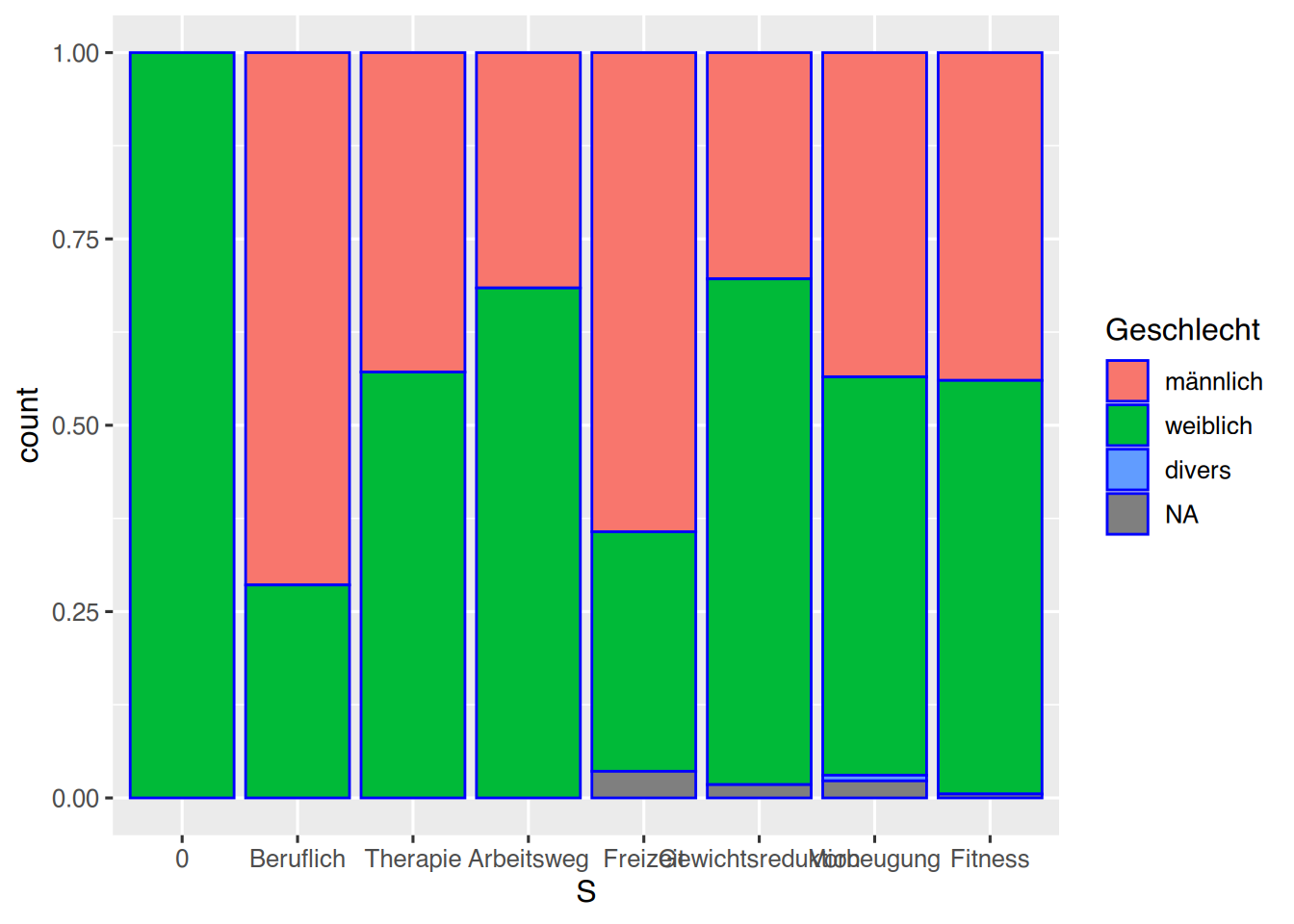
Mit position="dodge" werden die Säulen der Gruppen nebeneinander gestellt.
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum)) %>%
mutate(S = fct_rev(S)) %>%
ggplot() +
aes(x=S,
fill=Geschlecht) +
geom_bar(color="blue",
position="dodge")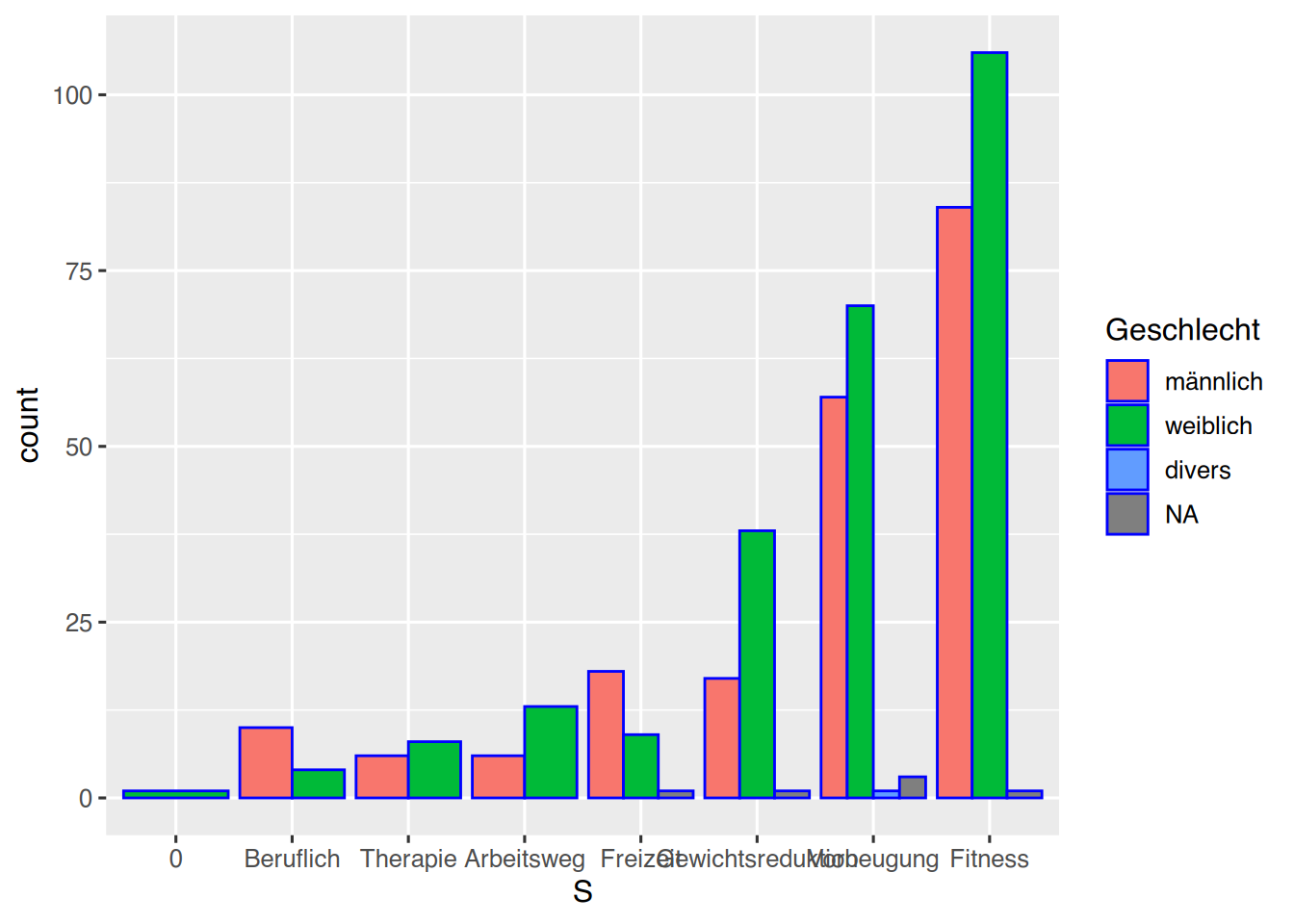
Mit position="jitter" wird etwas Rauschen bei der Positionierung verwendet. Das sieht nicht immer schön aus.
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum)) %>%
mutate(S = fct_rev(S)) %>%
ggplot() +
aes(x=S,
fill=Geschlecht) +
geom_bar(color="blue",
position="jitter")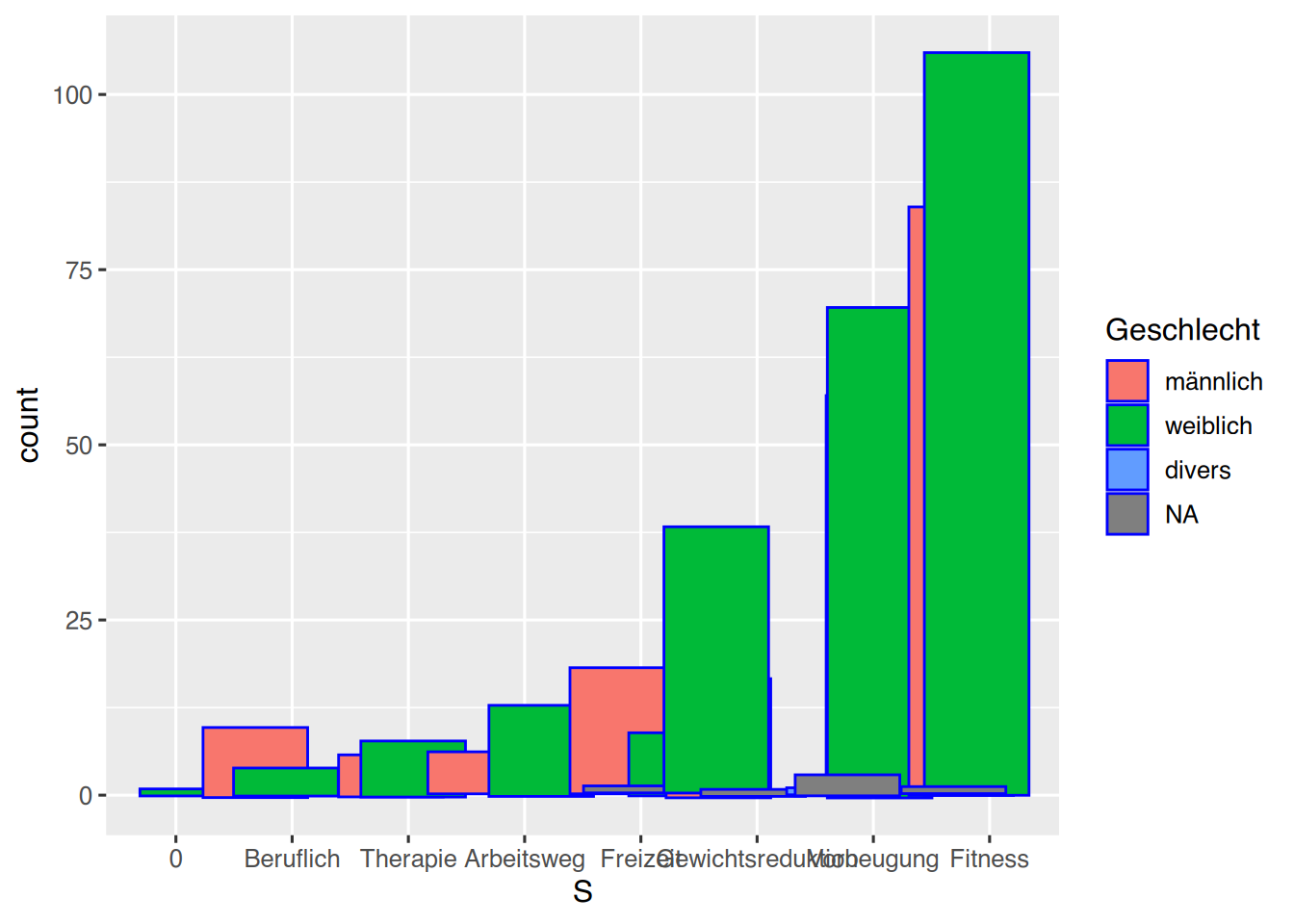
Die Default-Einstellung ist position="stack".
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum)) %>%
mutate(S = fct_rev(S)) %>%
ggplot() +
aes(x=S,
fill=Geschlecht) +
geom_bar(color="blue",
position="stack")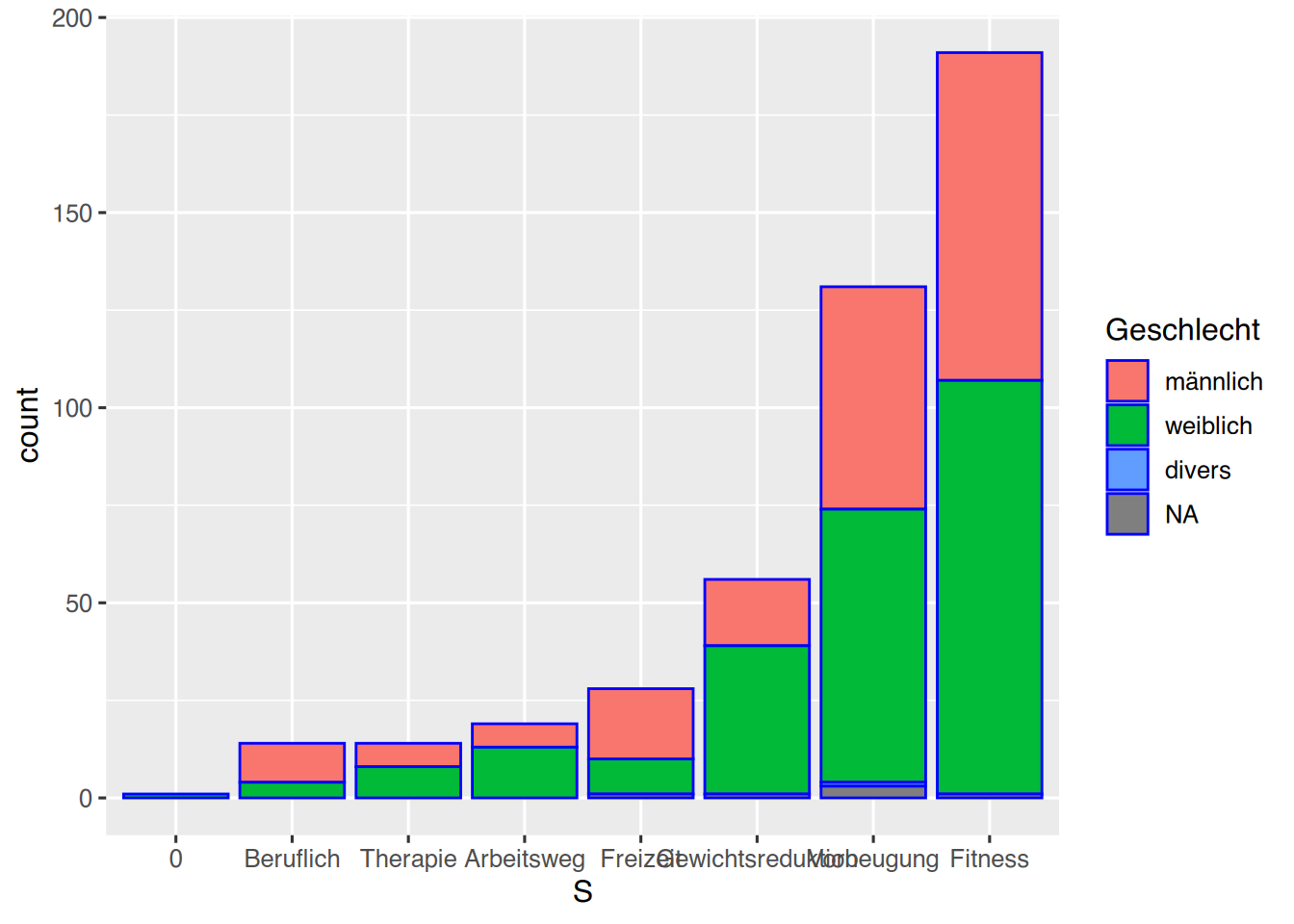
Mit der Funktion stat_count() können die jeweiligen Werte der Balken ausgeschrieben werden.
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum)) %>%
mutate(S = fct_rev(S)) %>%
ggplot() +
aes(x=S,
fill=Geschlecht) +
geom_bar(color="blue",
position="stack") +
stat_count(geom = "text",
aes(label = after_stat(count)),
position=position_stack(),
vjust=-0.5)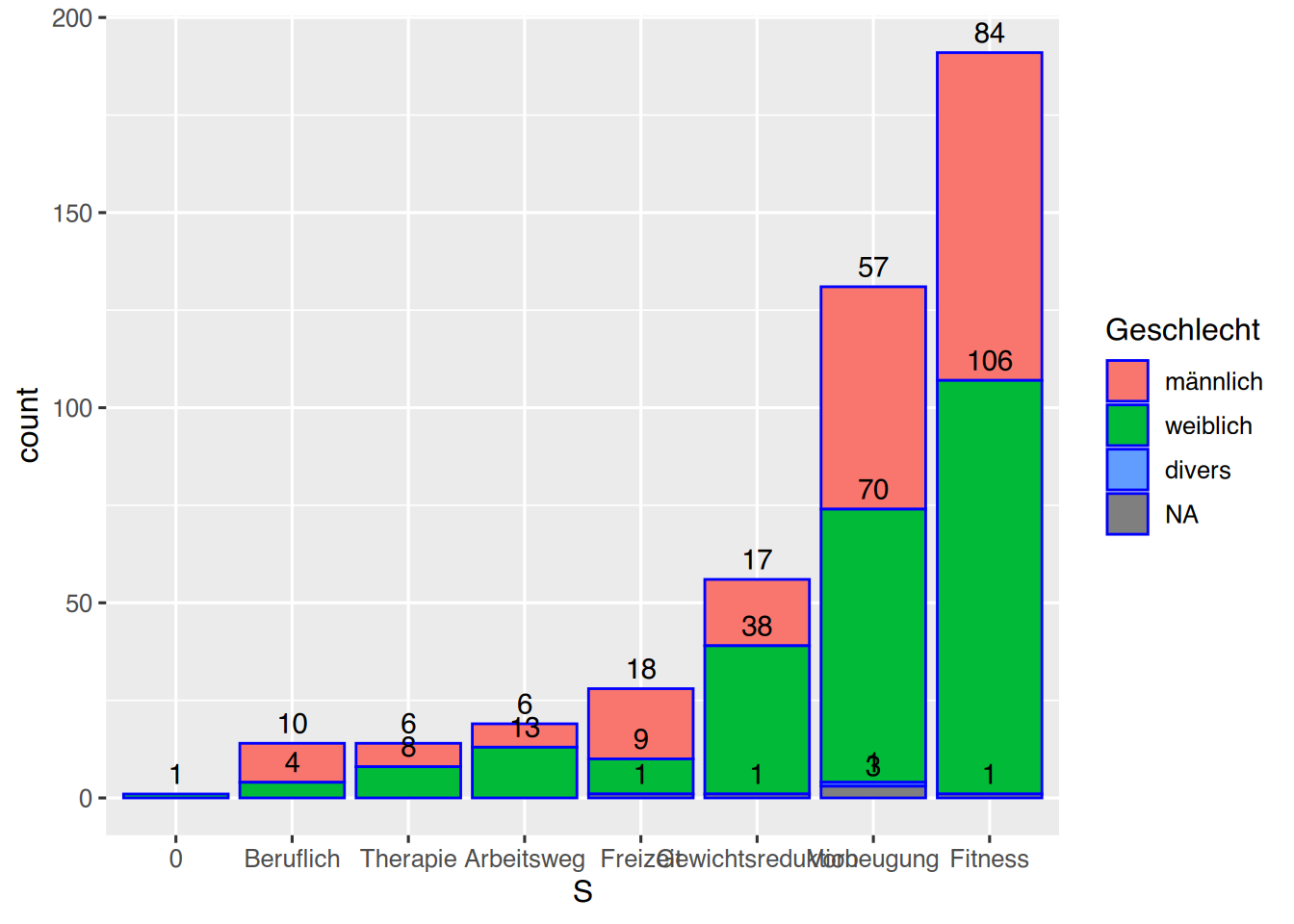
Mit dem Parameter vjust kann die Höhe der Beschriftungen verändert werden.
Beachten Sie, dass die Parameter position aus geom_bar() und stat_count() übereinstimmen müssen. Da in geom_bar(position="stack") gesetzt ist, muss innerhalb von stat_count() die Funktion position_stack() übergeben werden.
Ändern wir in geom_bar() den Parameter auf position="dodge", so muss innerhalb von stat_count() die Funktion position_dodge() übergeben werden.
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum)) %>%
mutate(S = fct_rev(S)) %>%
ggplot() +
aes(x=S,
fill=Geschlecht) +
geom_bar(color="blue",
position="dodge") +
stat_count(geom = "text",
aes(label = after_stat(count)),
position=position_dodge(width=1),
vjust=-0.5)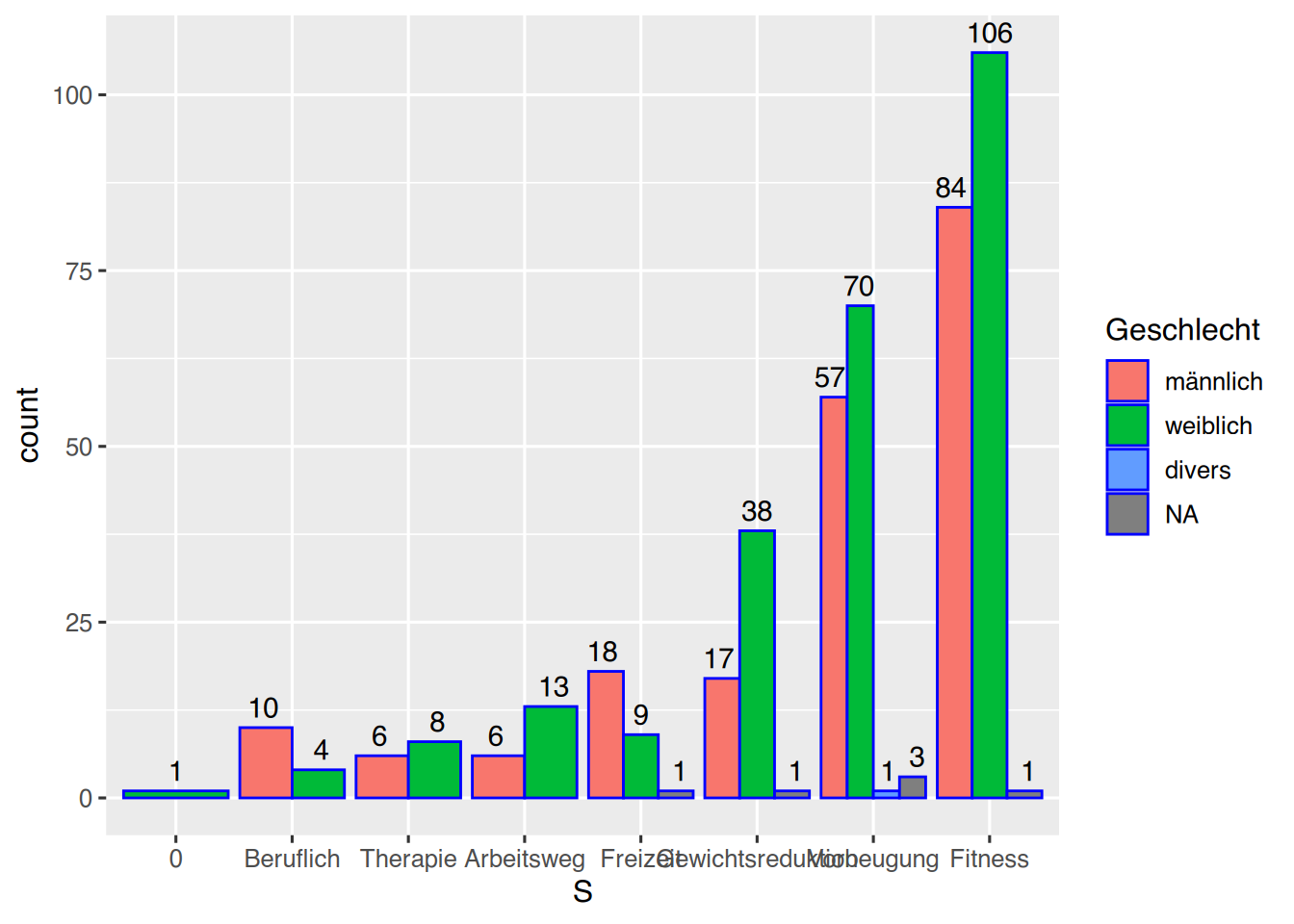
pf8 %>%
drop_na(SportWarum) %>%
mutate(S = fct_infreq(SportWarum)) %>%
mutate(S = fct_rev(S)) %>%
ggplot() +
aes(x=S,
fill=Geschlecht) +
geom_bar(color="blue",
position="fill") +
stat_count(geom = "text",
aes(label = after_stat(count)),
position=position_fill(vjust=0.5),
color="white")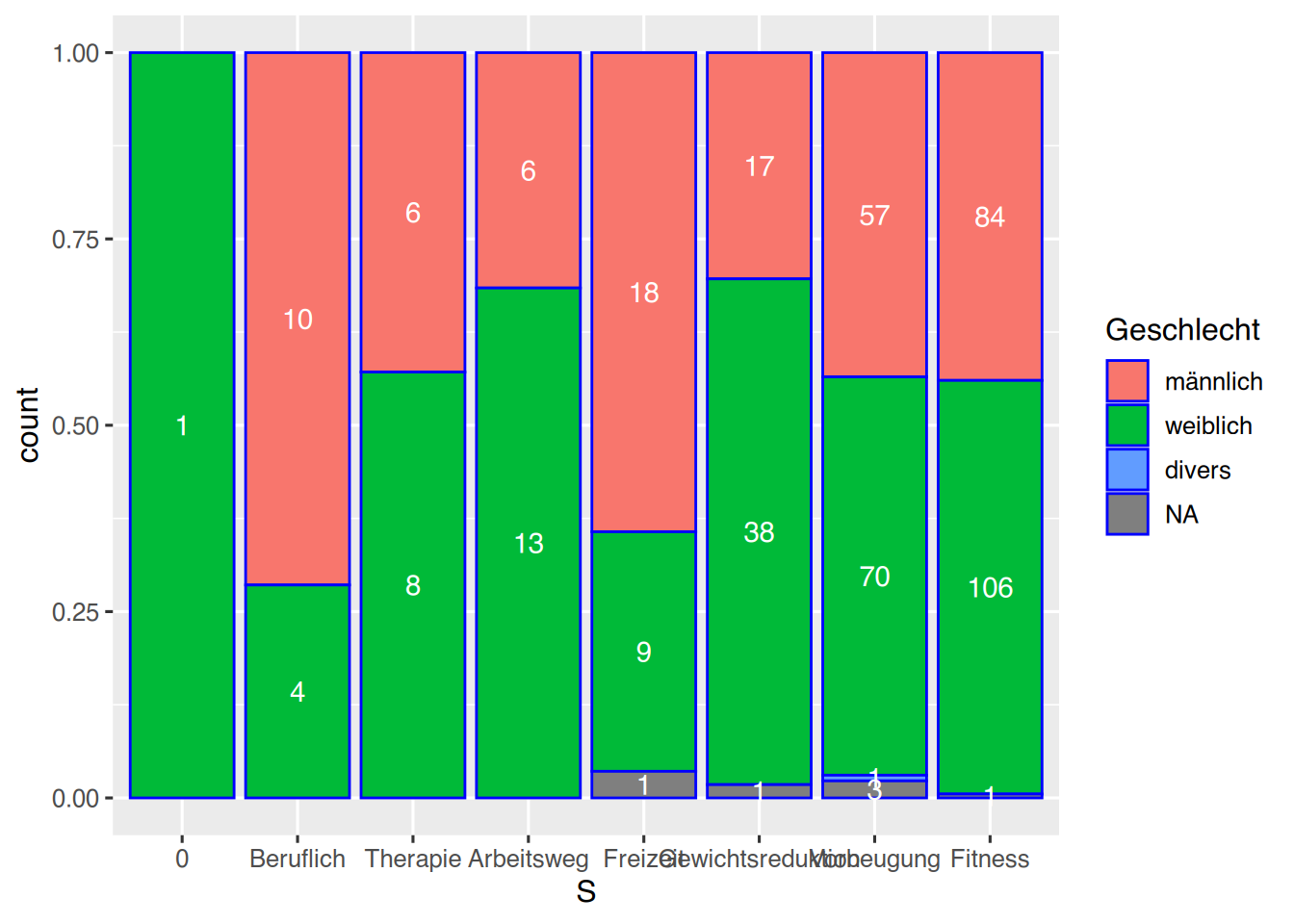
35.6.1 vorgegebene Werte
Mit dem Parameter stat="identity" können vorberechnete Werte (absolute Häufigkeiten) geplottet werden.
# vorgegebene Werte
df <- data.frame(Gruppe=c("Vanille","Schokolade","Erdbeer"),
Anteil=c(60, 25, 15))
# plotten
ggplot(df, aes(x=Gruppe, y=Anteil,
fill=Gruppe)) +
geom_bar(stat="identity",
position="dodge")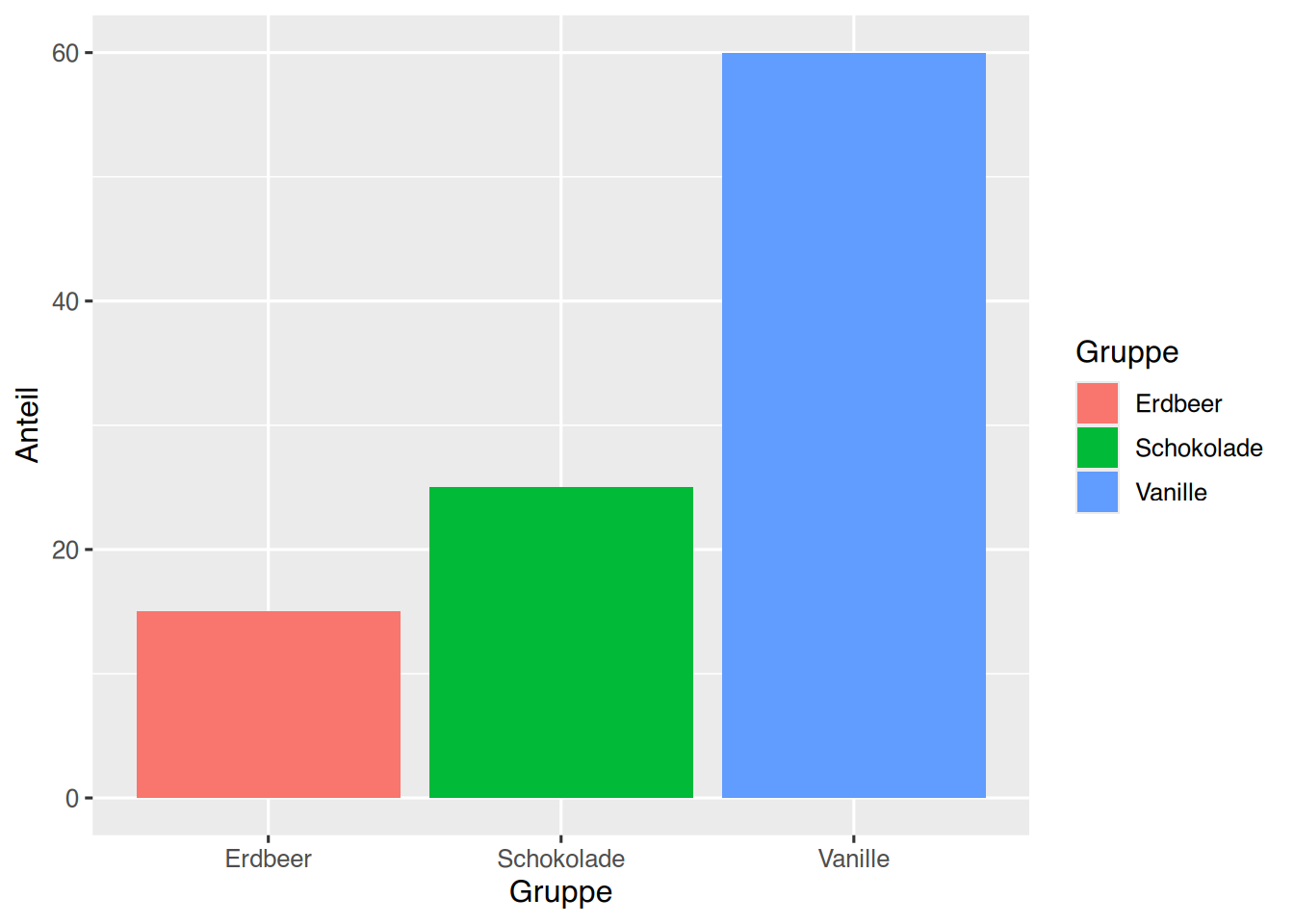
Alternativ kann das Plot mit der Funktion geom_col() erzeugt werden, die von Hause aus stat_identity() nutzt.
# vorgegebene Werte
df <- data.frame(Gruppe=c("Vanille","Schokolade","Erdbeer"),
Anteil=c(60, 25, 15))
# plotten mit geom_col()
ggplot(df, aes(x=Gruppe, y=Anteil,
fill=Gruppe)) +
geom_col(position="dodge")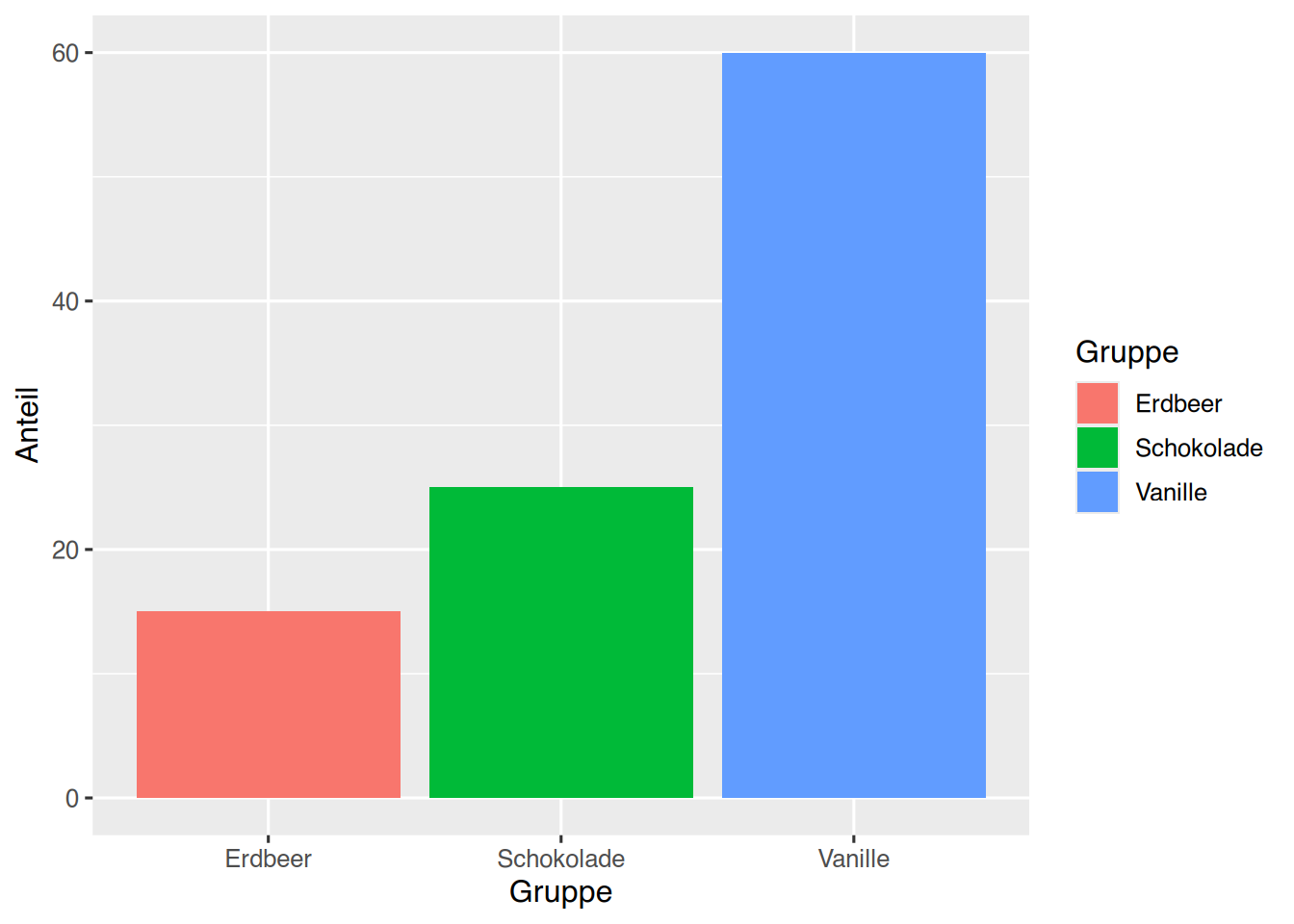
Mit der Funktion geom_text() können die Werte den Plot-Balken hinzugefügt werden. Über den Parameter vjust kann bestimmt werden, ob die Werte über oder in den Säulen stehen sollen.
ggplot(df, aes(x=Gruppe, y=Anteil,
fill=Gruppe)) +
geom_col(position="dodge")+
# Werte stehen über den Balken
geom_text(aes(label = Anteil), vjust = -0.2)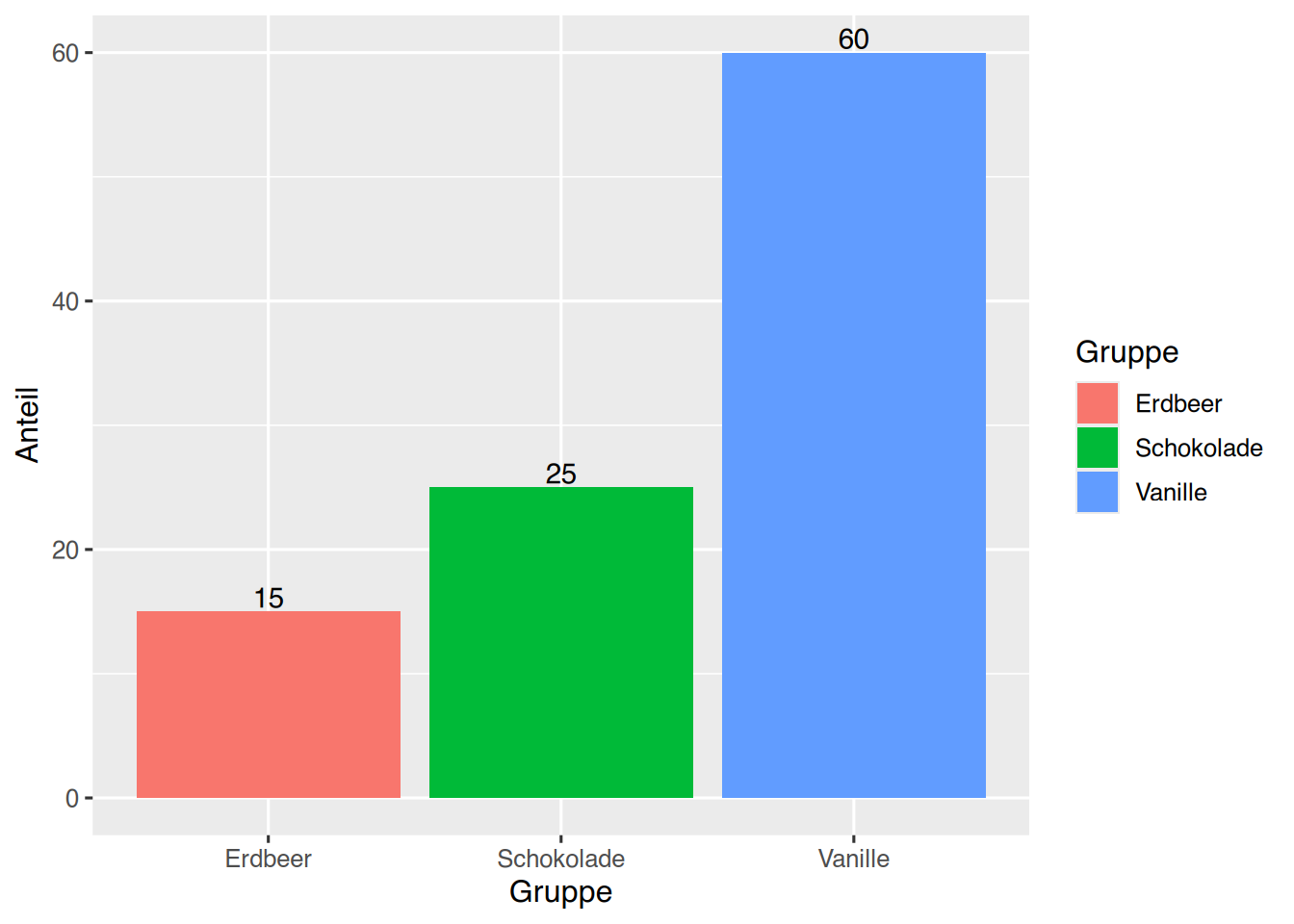
ggplot(df, aes(x=Gruppe, y=Anteil,
fill=Gruppe)) +
geom_col(position="dodge")+
# Werte stehen unter den Balken
geom_text(aes(label = Anteil), vjust = 2)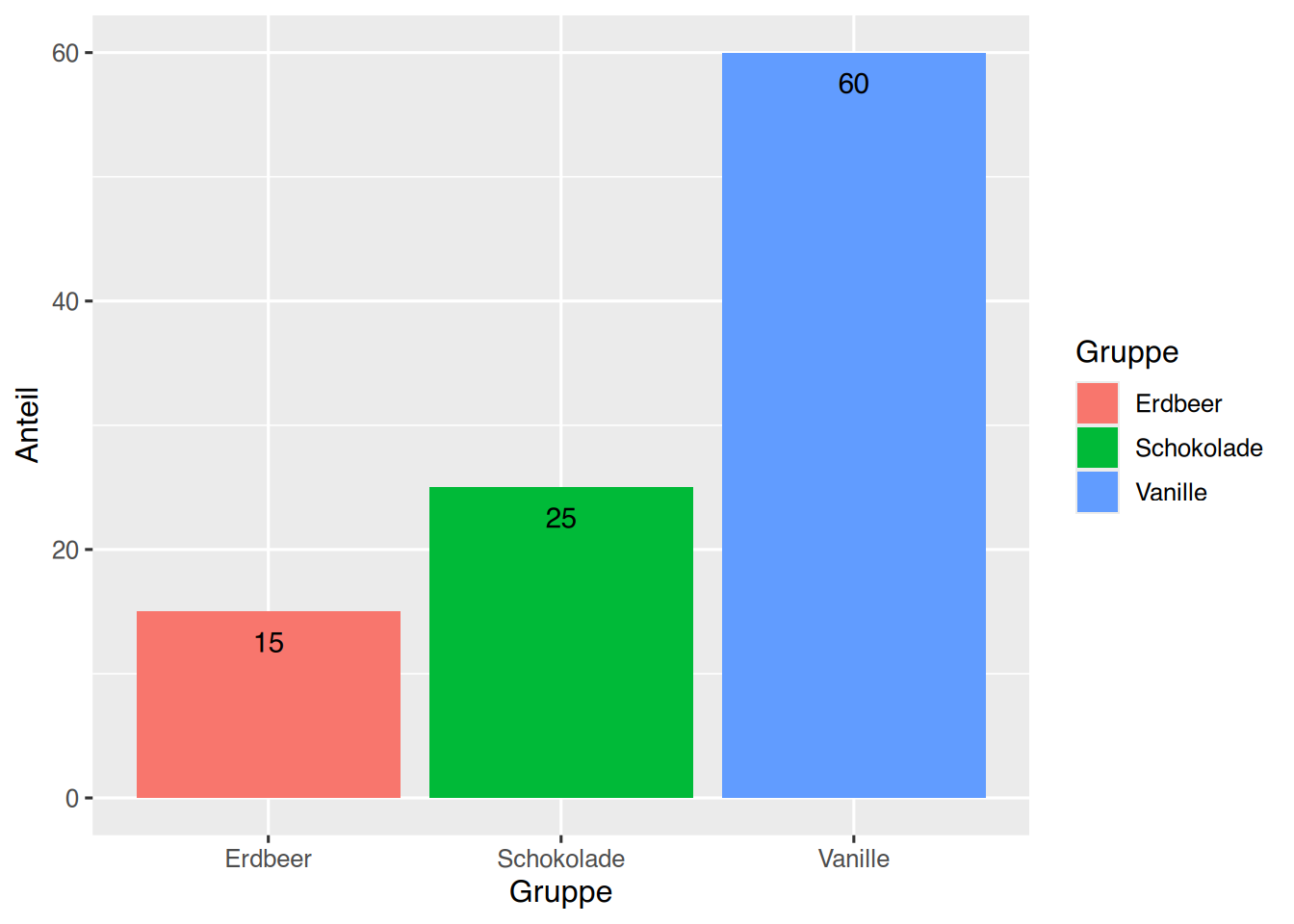
Das selbe Ergebis kann auch mit der Funktion stat_identity() erzielt werden. Achten Sie auch hier darauf, dass die Parameter position von geom_col() (bzw. geom_bar()) und von stat_identity() gleich sein müssen.
ggplot(df, aes(x=Gruppe, y=Anteil,
fill=Gruppe)) +
geom_col(position="dodge") +
stat_identity(geom = "text",
aes(label=Anteil),
position=position_dodge(width=1),
vjust=2, size=5, color="white")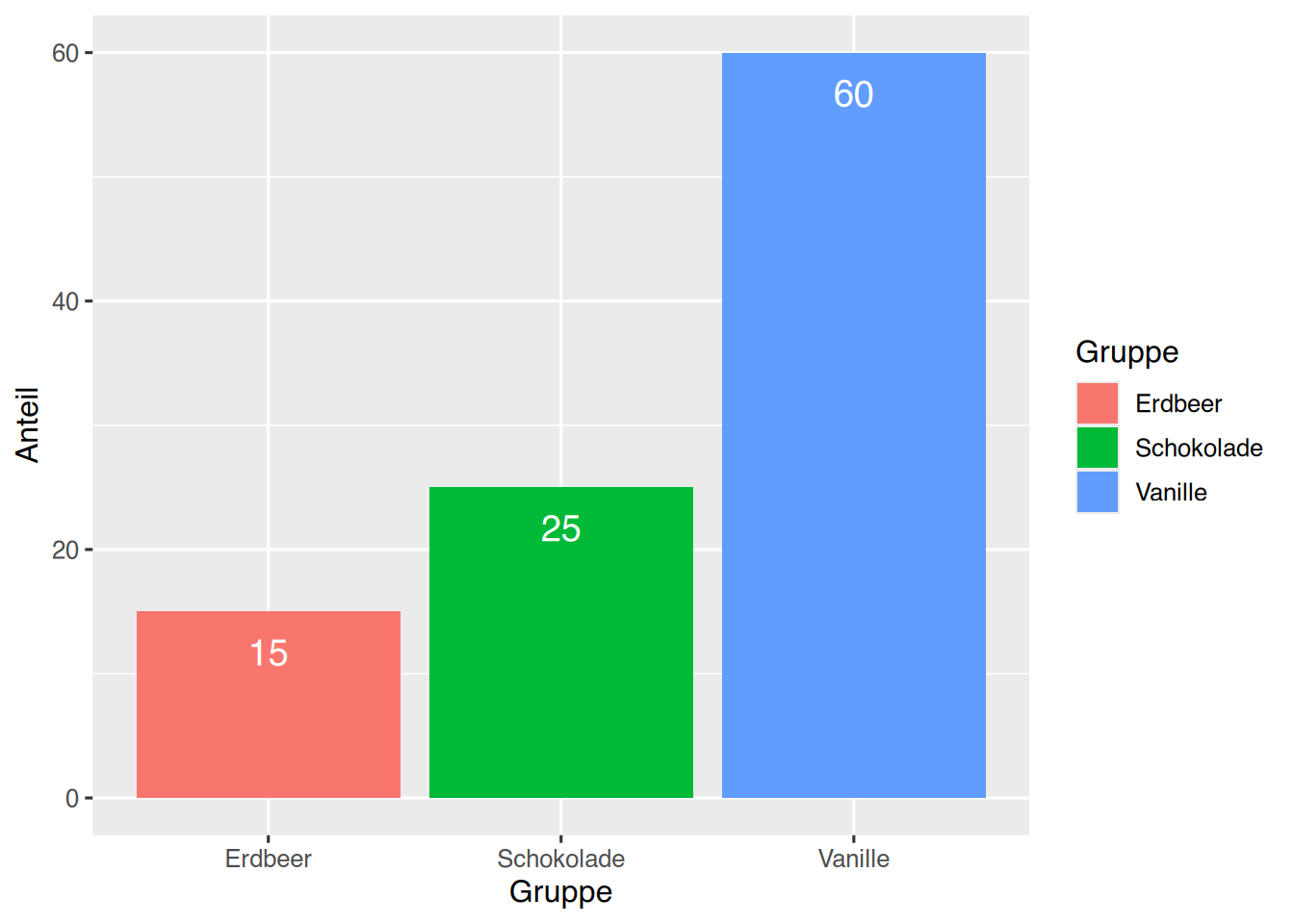
35.7 Balkendiagramme
Balkendiagramme können auf zwei Arten erstellt werden. In der aes()-Funktion tauschen wir einfach x durch y aus:
ggplot(pf8) +
aes(y=SportWarum ) +
geom_bar(color="blue",
fill="skyblue")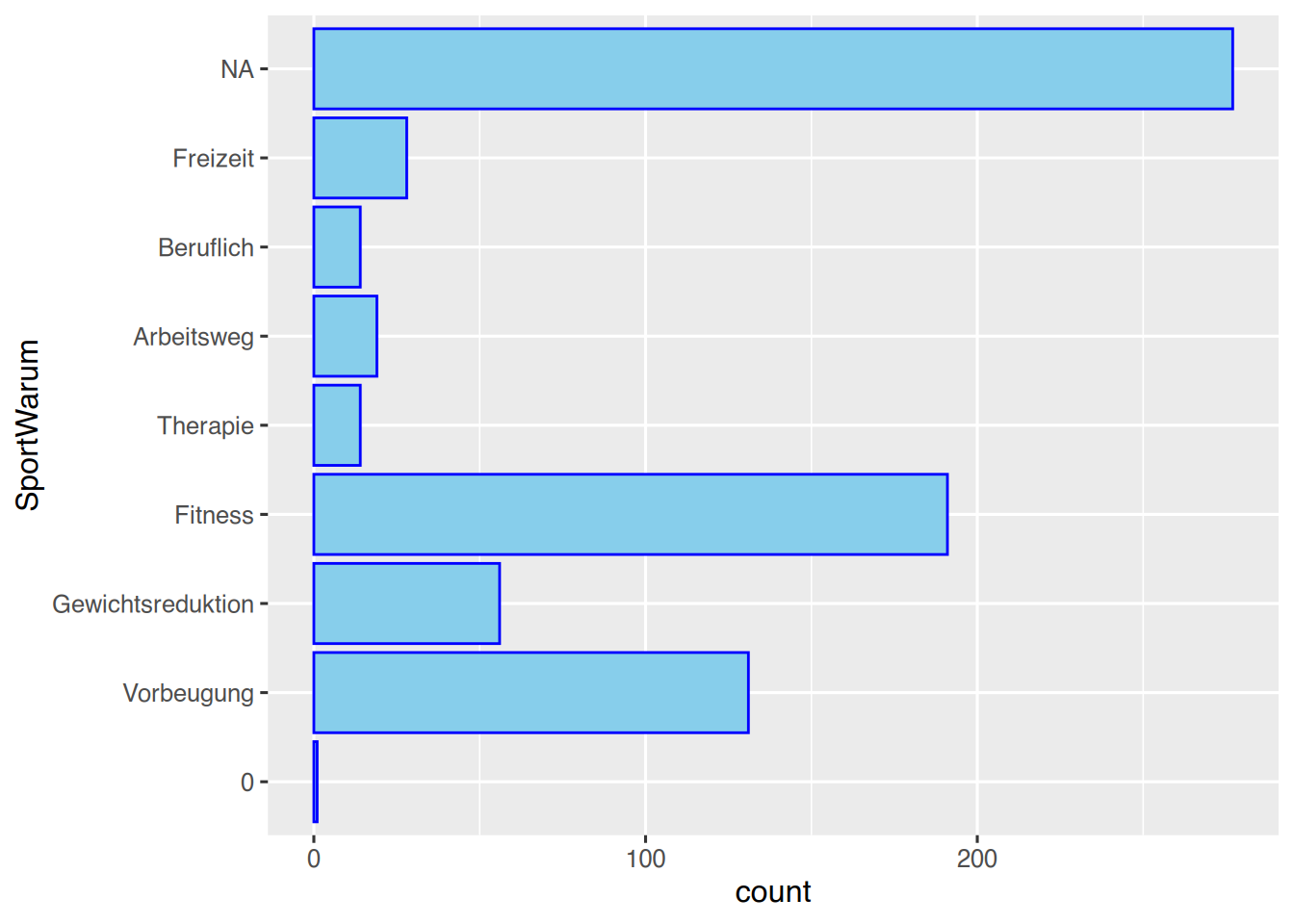
Alternativ kann dem Plotaufruf die Funktion coord_flip() angehängt werden.
ggplot(pf8) +
aes(x=SportWarum ) +
geom_bar(color="blue",
fill="skyblue") +
coord_flip()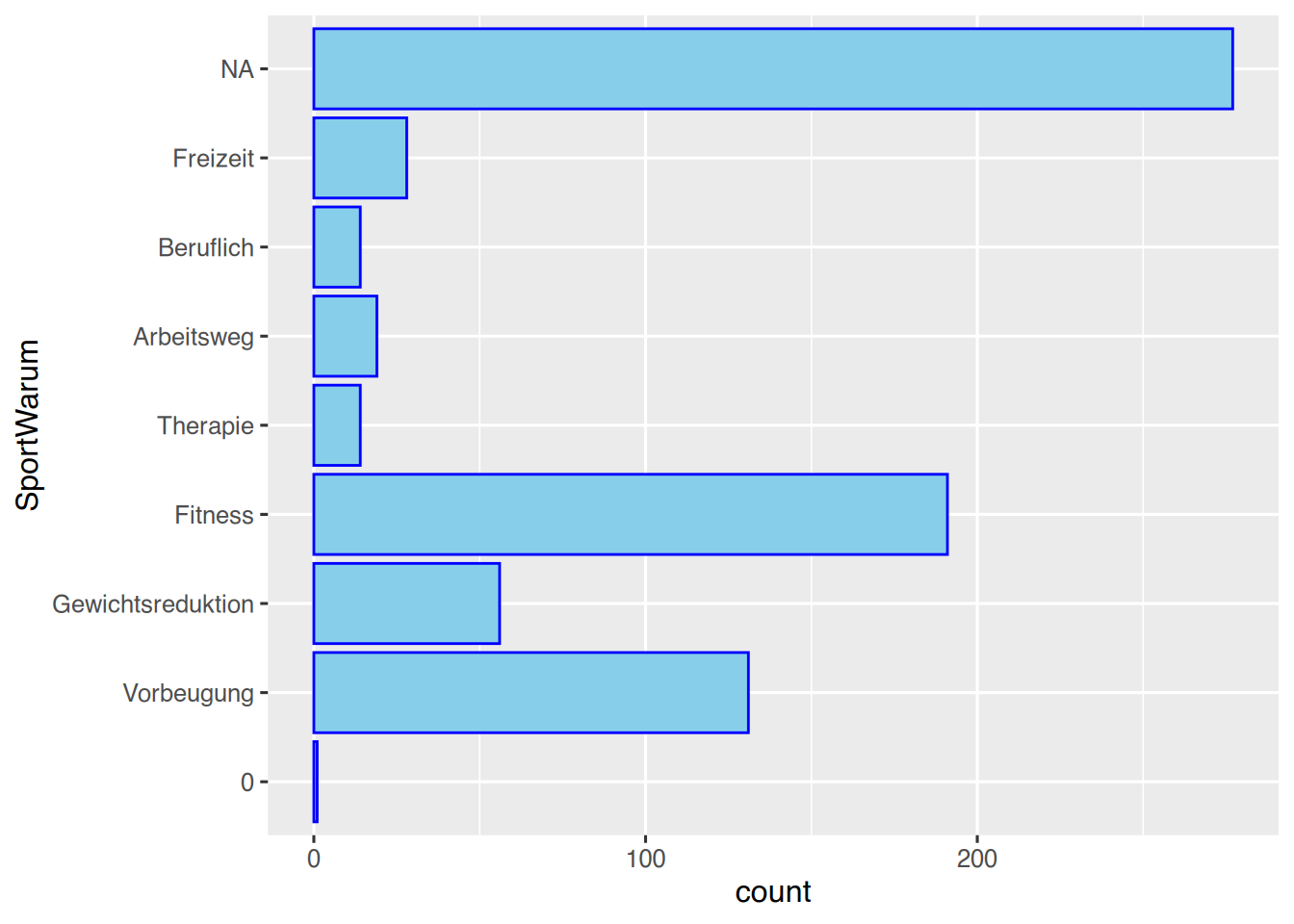
35.8 Boxplots
Boxplots werden mit der Funktion geom_boxplot() erzeugt.
pf8 %>%
ggplot() +
aes(x = Alter) +
geom_boxplot(fill="seagreen3")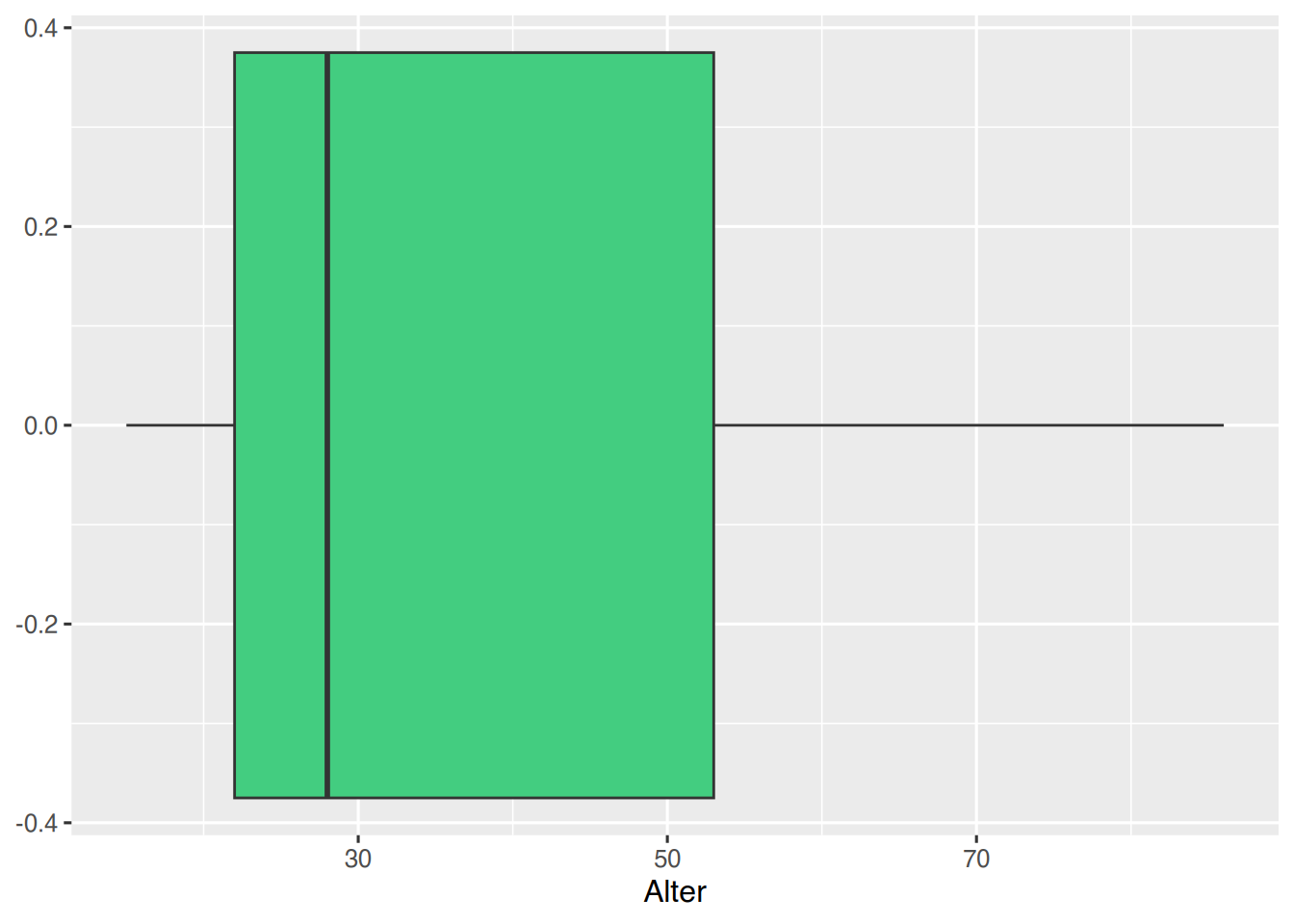
Das Boxplot liegt auf dem Bauch, das Alter auf der x-Achse abgebildet werden soll. Um das Boxplot zu drehen, muss aes() entsprechend angepasst werden.
pf8 %>%
ggplot() +
aes(y = Alter) +
geom_boxplot(fill="seagreen3")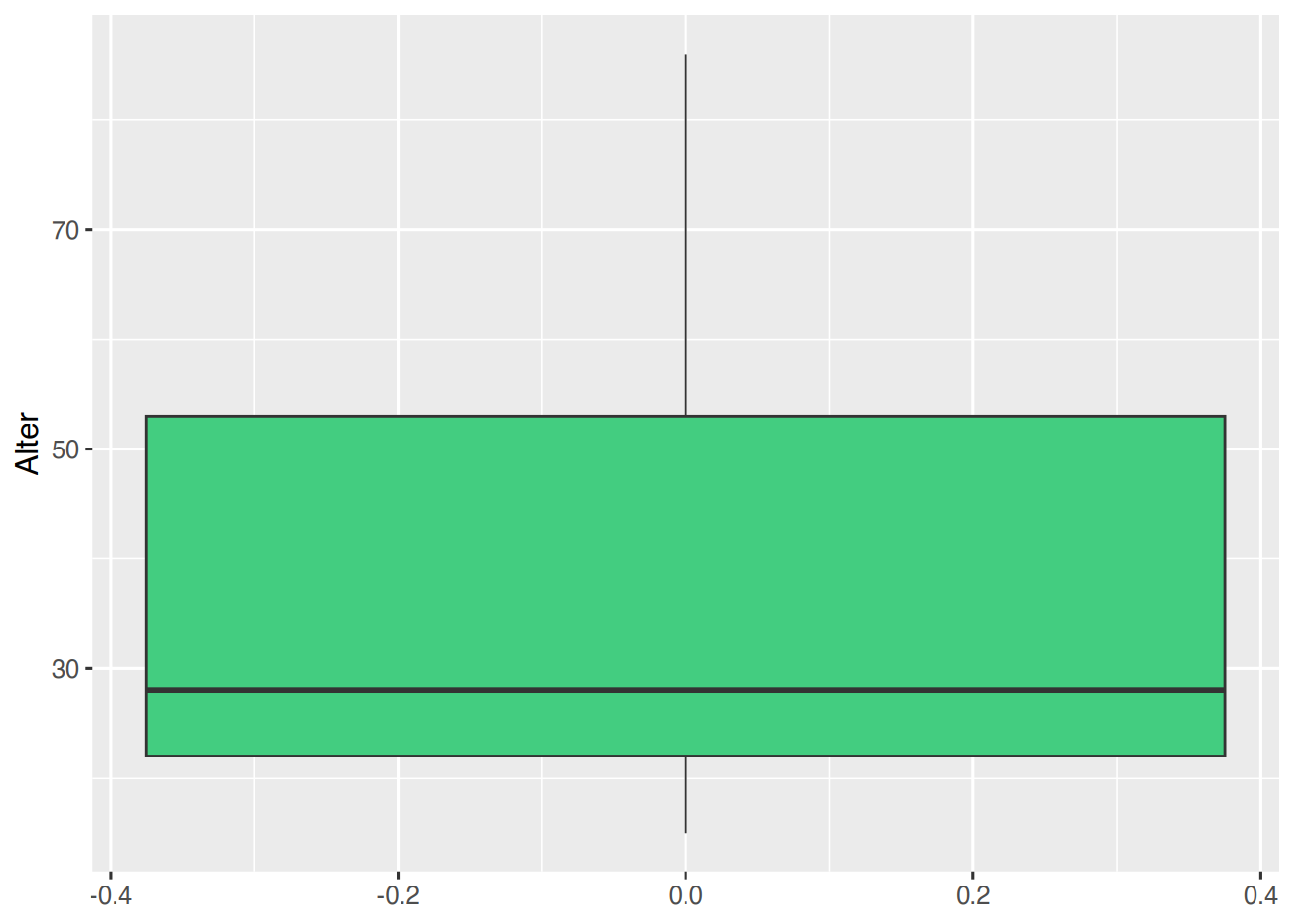
Mit der Funktion stat_boxplot() können wir die Whiskers hinzugefügt werden.
pf8 %>%
ggplot() +
aes(y = Alter) +
geom_boxplot(fill="seagreen3") +
stat_boxplot(geom="errorbar")
Über den color-Parameter können die Farben angepasst werden.
pf8 %>%
ggplot() +
aes(y = Alter) +
geom_boxplot(fill="seagreen3",
color="darkblue") +
stat_boxplot(geom="errorbar",
color="darkgreen")
Über die Aesthetics können wir nach Geschlecht gruppieren.
pf8 %>%
ggplot() +
aes(y = Alter,
x = Geschlecht) +
geom_boxplot(fill="seagreen3",
color="darkblue") +
stat_boxplot(geom="errorbar",
color="darkgreen")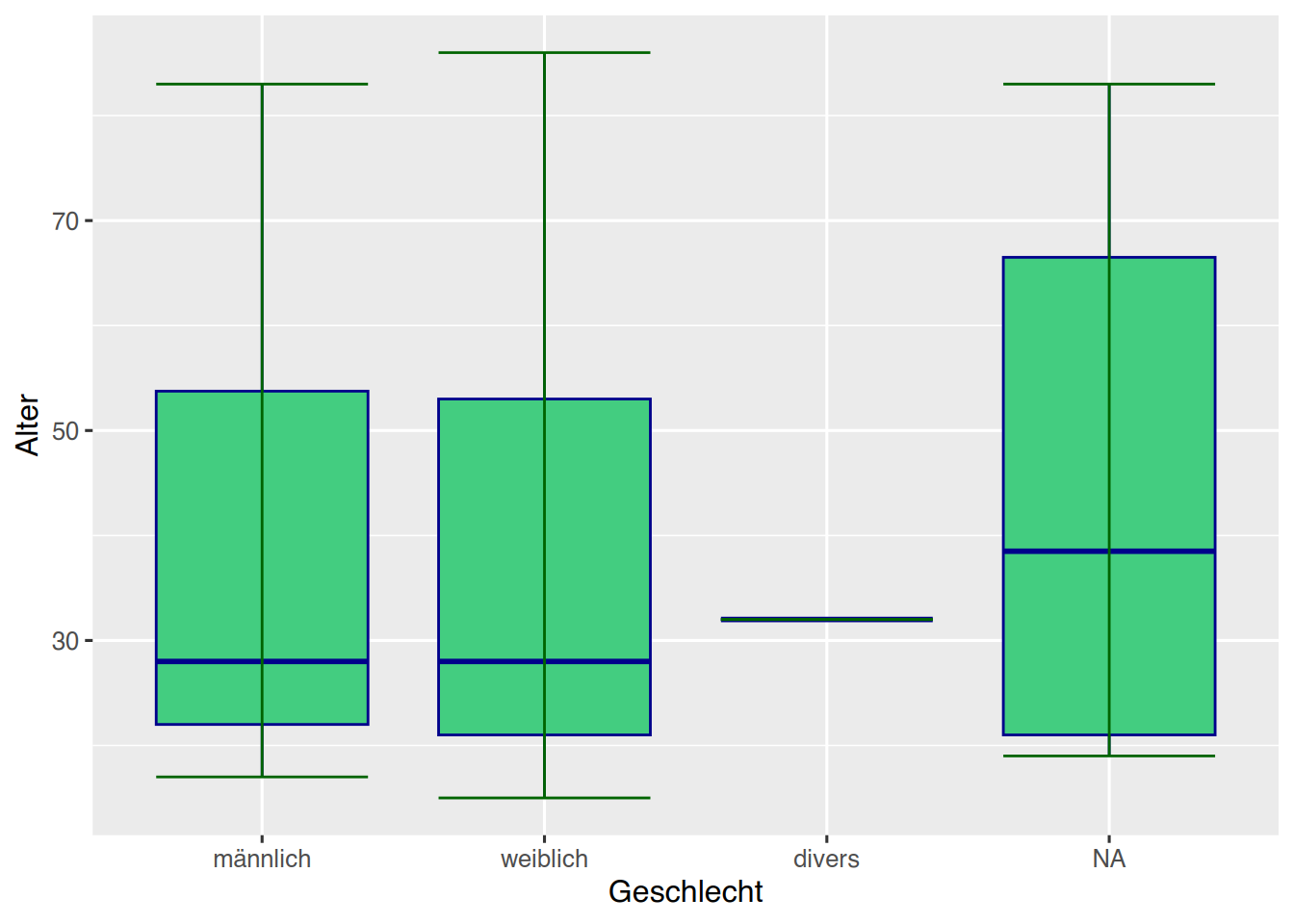
Bzw. ohne NA:
pf8 %>%
drop_na(Geschlecht) %>%
ggplot() +
aes(y = Alter,
x = Geschlecht) +
geom_boxplot(fill="seagreen3",
color="darkblue") +
stat_boxplot(geom="errorbar",
color="darkgreen")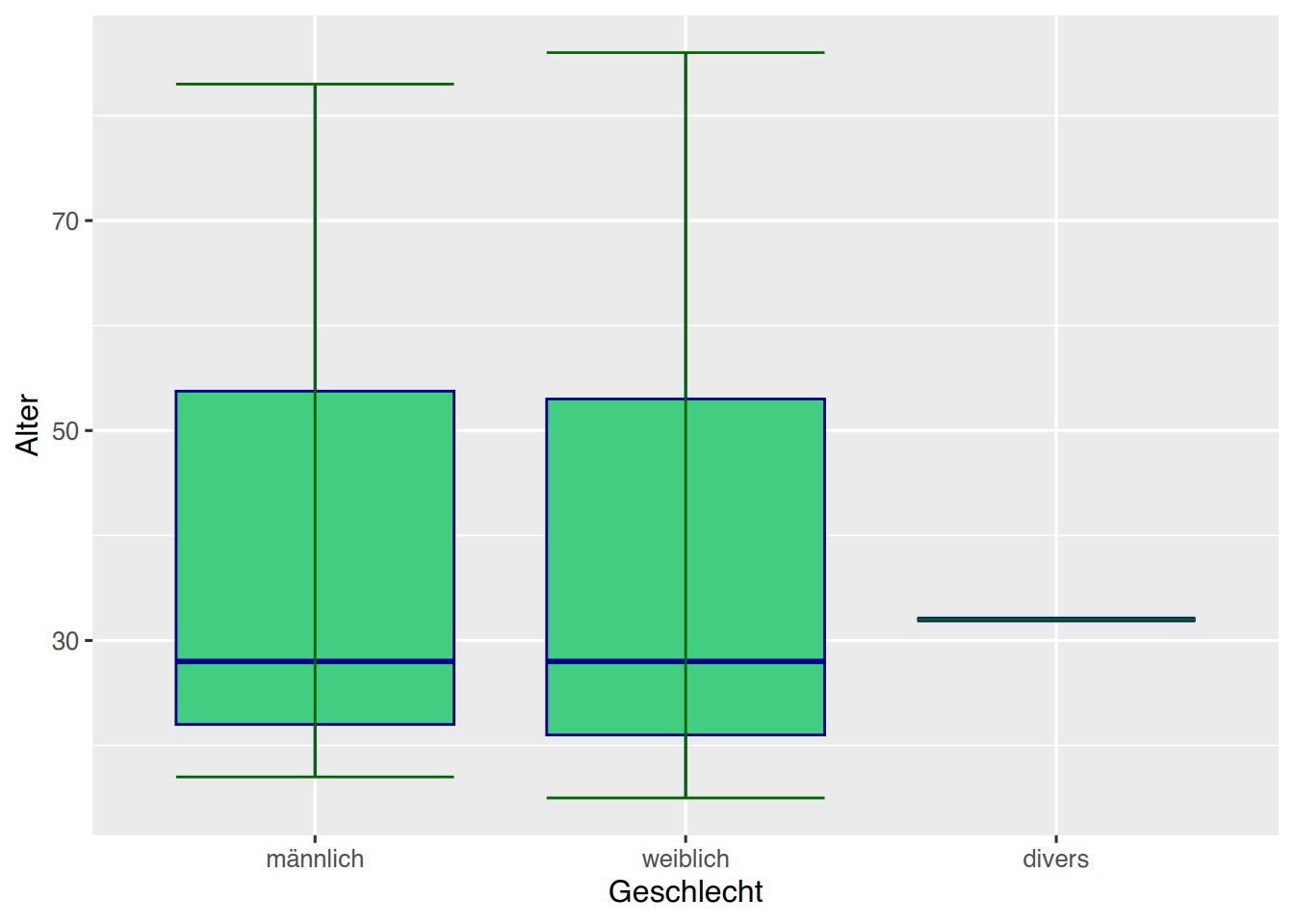
Über die Aesthetics können wir die Boxplots auch nach Geschlecht färben. Dazu muss der Parameter aus geom_boxplot() entfernt werden.
pf8 %>%
drop_na(Geschlecht) %>%
ggplot() +
aes(y = Alter,
x = Geschlecht,
fill = Geschlecht) +
geom_boxplot(color="darkblue") +
stat_boxplot(geom="errorbar",
color="darkgreen")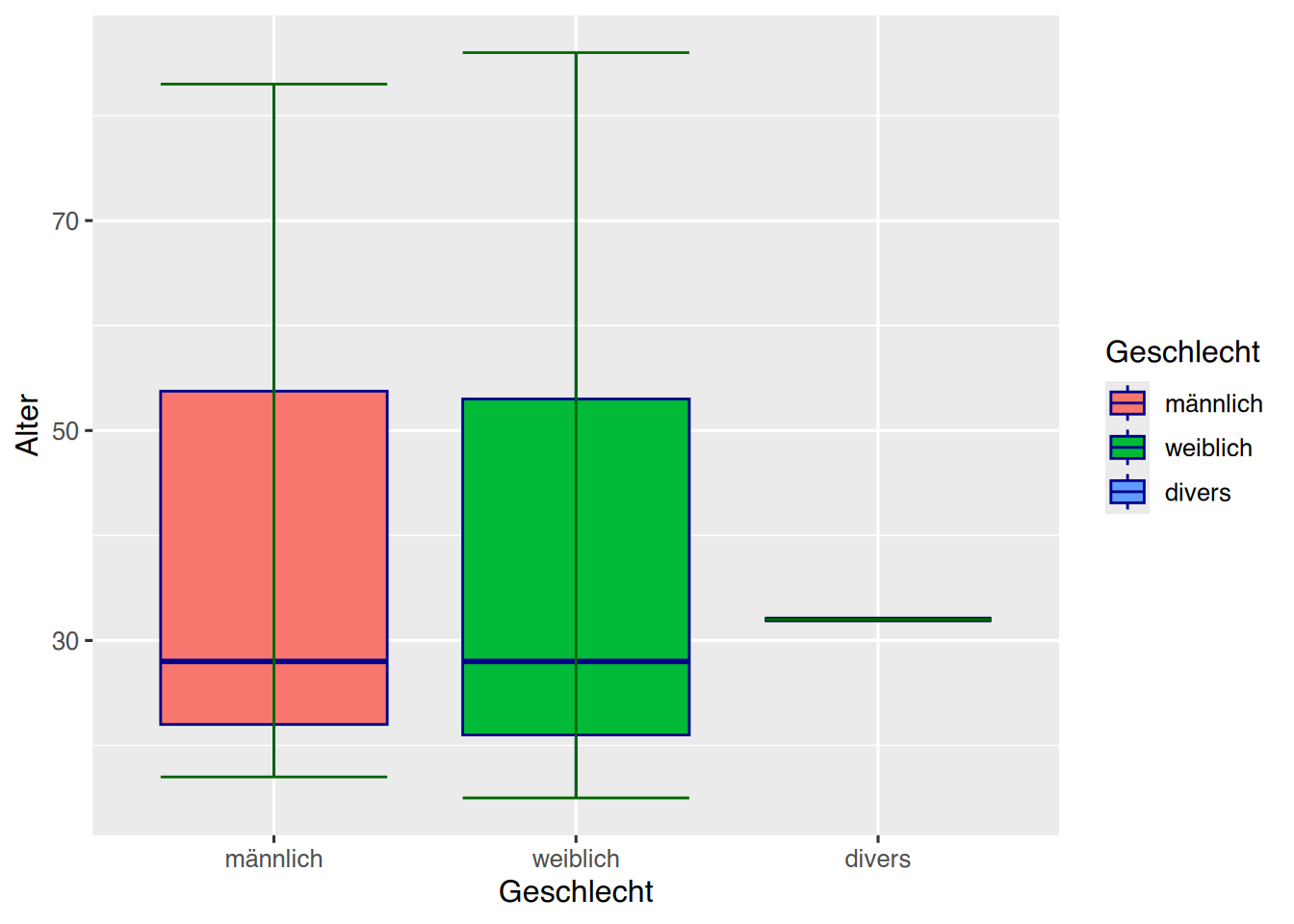
In manchen Journals ist es üblich, ebenfalls das arithmetische Mittel per Raute den Boxplots hinzuzufügen. Dies kann mit der Funktion stat_summary() umgesetzt werden.
pf8 %>%
drop_na(Geschlecht) %>%
ggplot() +
aes(y = Alter,
x = Geschlecht,
fill = Geschlecht) +
geom_boxplot(color="darkblue") +
stat_boxplot(geom="errorbar",
color="darkgreen")+
stat_summary(fun=mean,
colour="darkred",
geom="point",
shape=18,
size=5,
show.legend = F)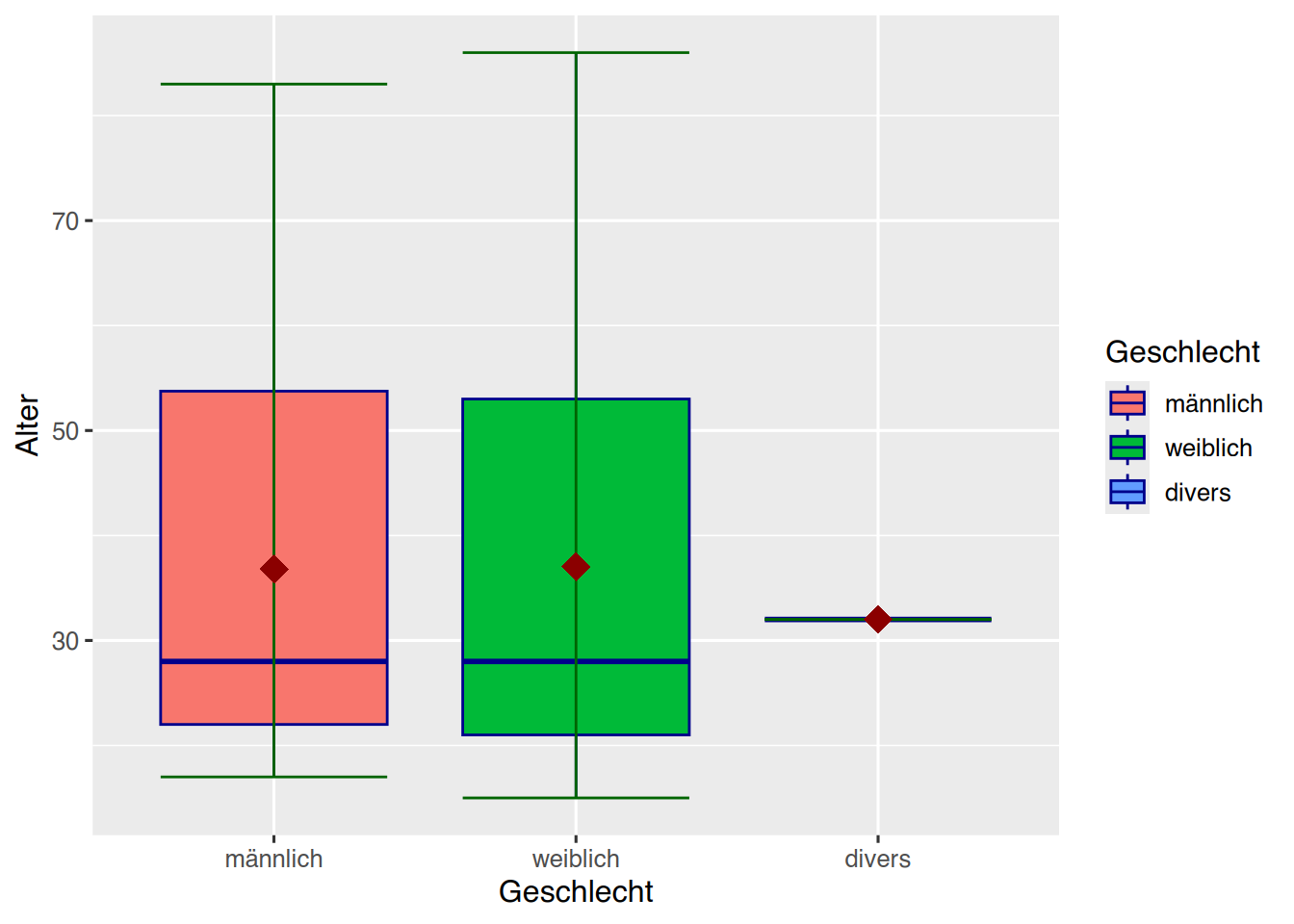
Mit facet_wrap() können wir beispielweise nach Standort gruppieren.
pf8 %>%
drop_na(Geschlecht) %>%
ggplot() +
aes(y = Alter,
x = Geschlecht,
fill = Geschlecht) +
geom_boxplot(color="darkblue") +
stat_boxplot(geom="errorbar",
color="darkgreen")+
stat_summary(fun=mean,
colour="darkred",
geom="point",
shape=18,
size=5,
show.legend = F)+
facet_wrap(~ Standort)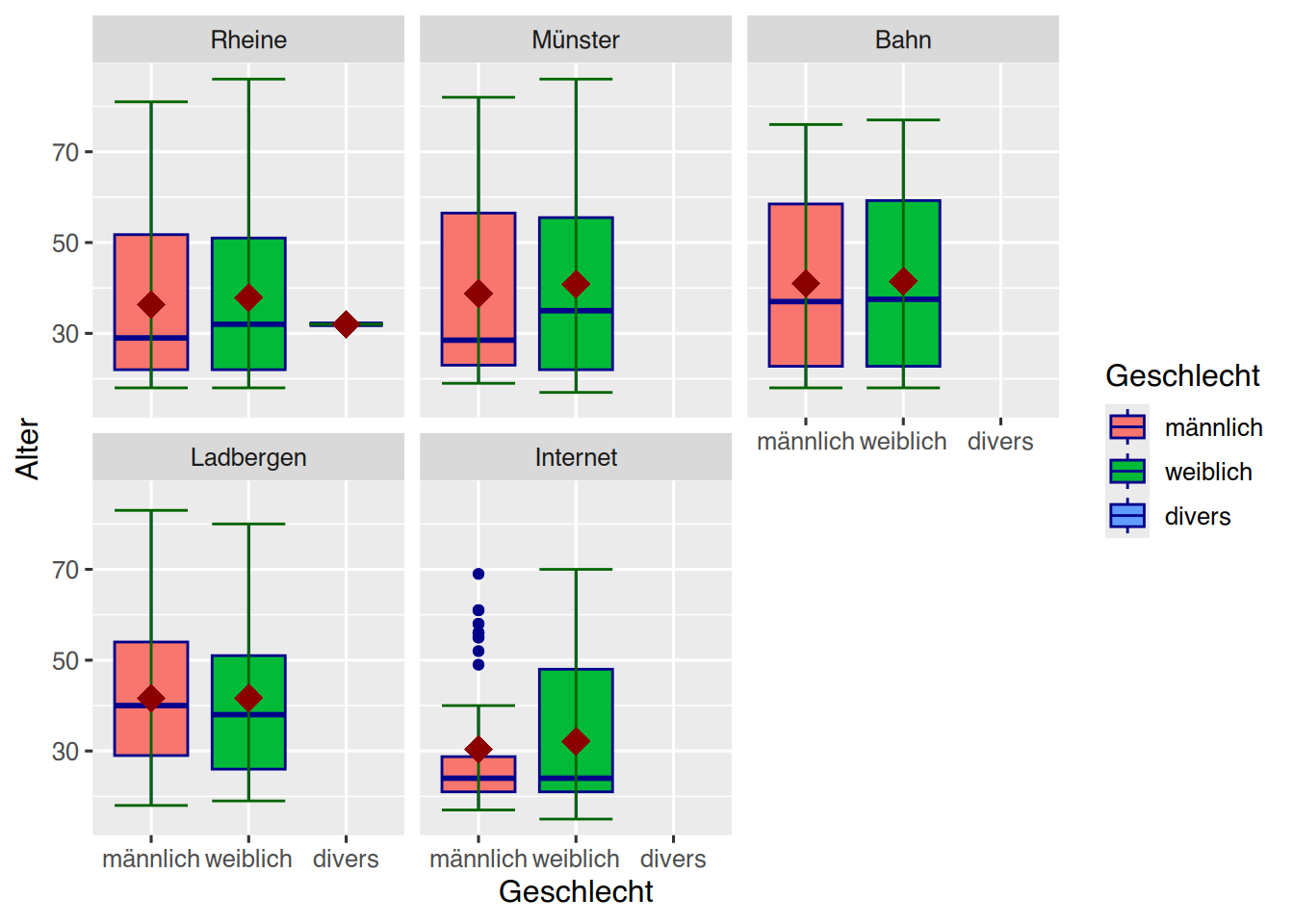
35.9 Kreisdiagramme
Kreisdiagramme werden von ggplot() nicht direkt unterstützt, können jedoch mit einem Trick erzeugt werden. Hierbei wird das Koordinatensystem eines Balkendiagramms mittels coord_polar() so verbogen, dass ein Kreis dabei herauskommt.
# pfusche Pie-Chart zurecht
pf8 %>%
ggplot() +
aes(x="", fill=Geschlecht) +
geom_bar(color="white") +
# verbiege Koordinatensystem
coord_polar("y") +
# entferne Achsen und Ticks
theme_void()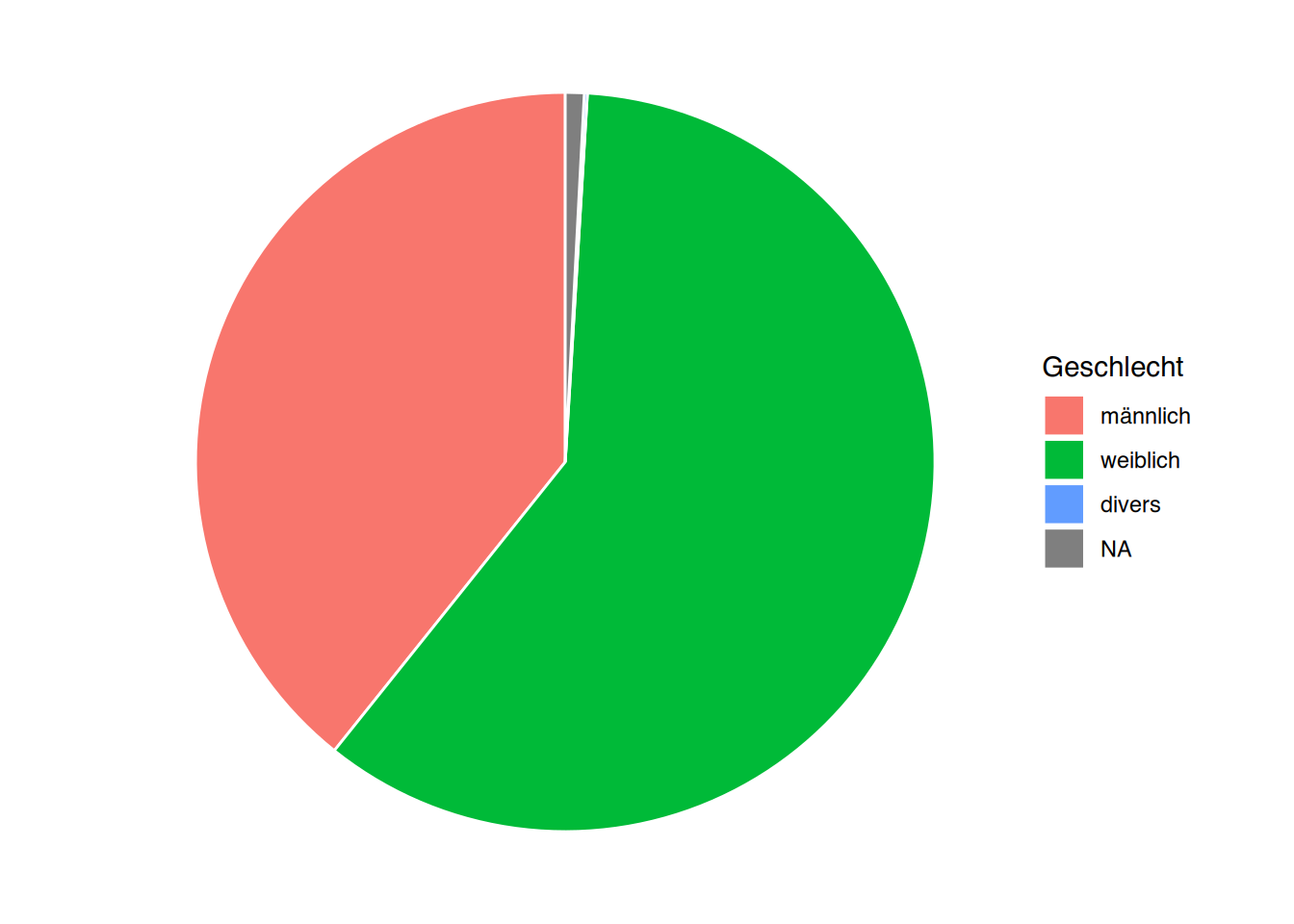
Das funktioniert auch mit geom_col() und absoluten Häufigkeiten.
# vorgegebene Werte
df <- data.frame(Gruppe=c("Vanille","Schokolade","Erdbeer"),
Anteil=c(60, 25, 15))
# pfusche Pie-Chart zurecht
ggplot(df, aes(x="",
y=Anteil,
fill=Gruppe)) +
geom_col(color="white") +
# verbiege Koordinatensystem
coord_polar("y", start=0) +
# entferne Achsen und Ticks
theme_void()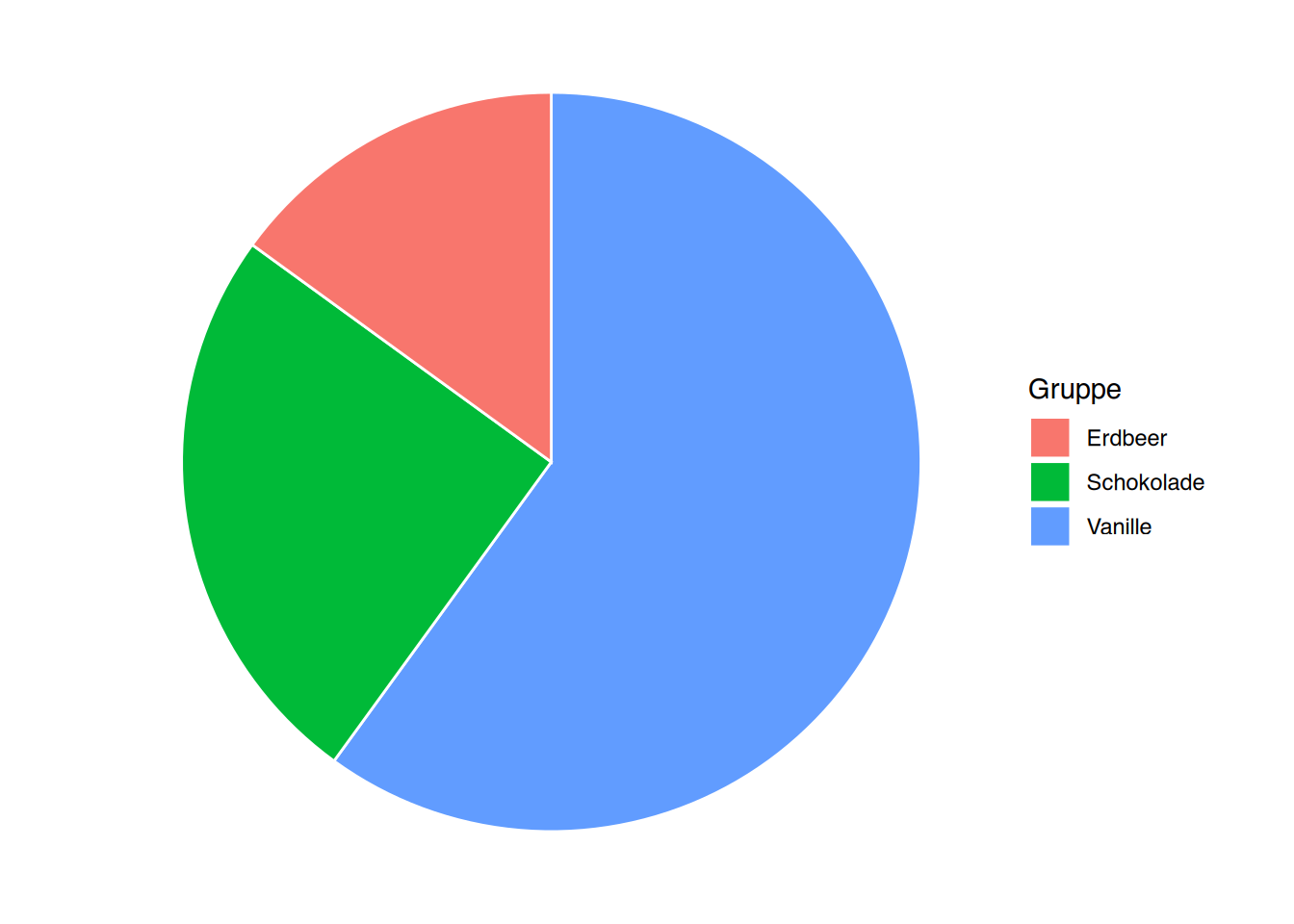
Mittels geom_text() können die Werte in den Kreis geschrieben werden.
ggplot(df, aes(x="",
y=Anteil,
fill=Gruppe)) +
geom_col(color="white") +
# füge Werte hinzu
geom_text(aes(label = Anteil),
position = position_stack(vjust = 0.5)) +
# verbiege Koordinatensystem
coord_polar("y", start=0) +
# entferne Achsen und Ticks
theme_void()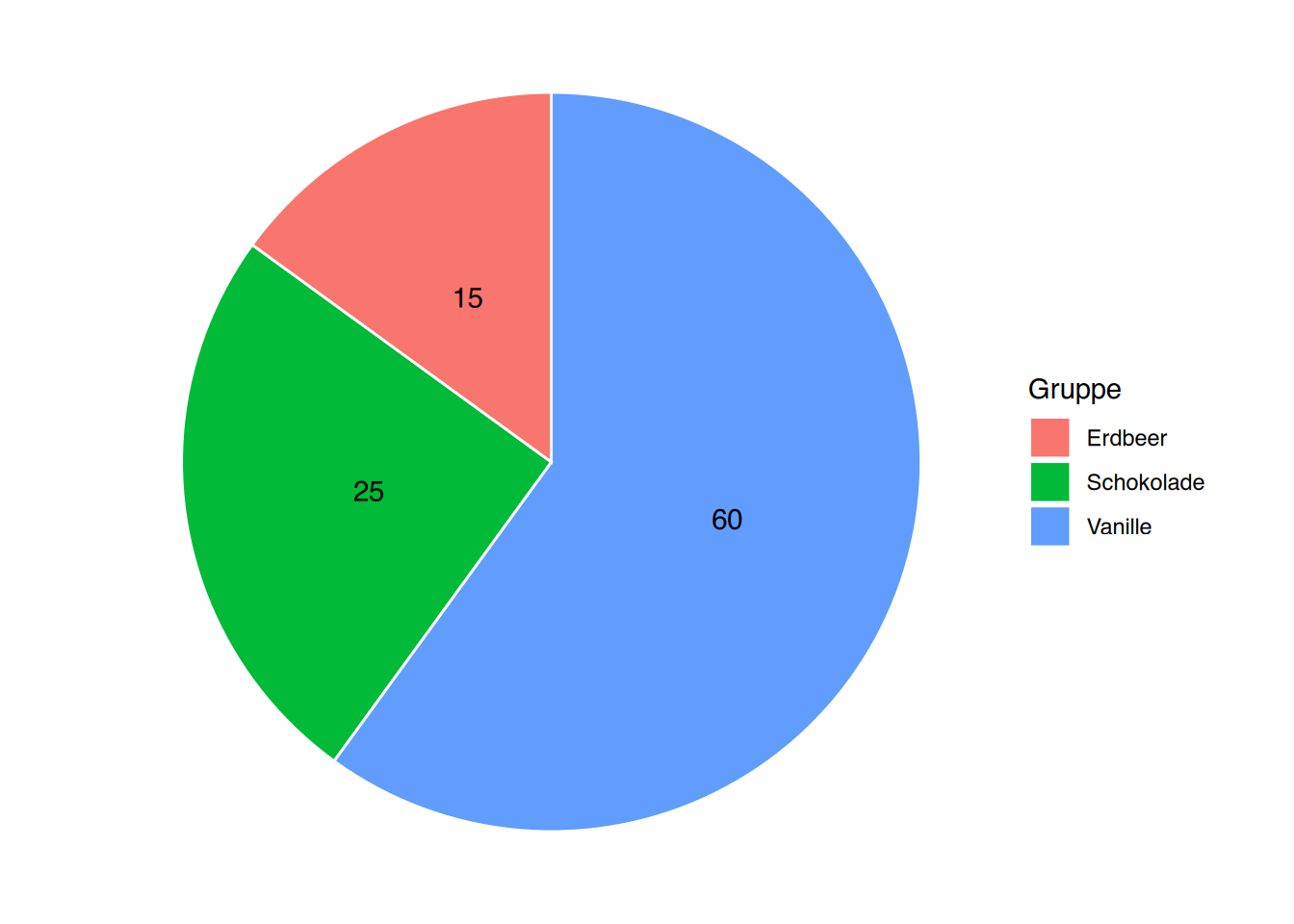
35.10 QQ-Plots
Zur Überprüfung, ob eine Variable normalverteilt ist, werden häufig Quantil-Quantil-Diagramme (QQ-Plots) erzeugt. Für ggplot() steht hierfür die Funktion stat_qq() zur Verfügung.
# erstelle QQ-Plot für Variable "age"
# gruppiert nach "sex"
ggplot(epa, aes(sample=age)) +
stat_qq(aes(color=sex))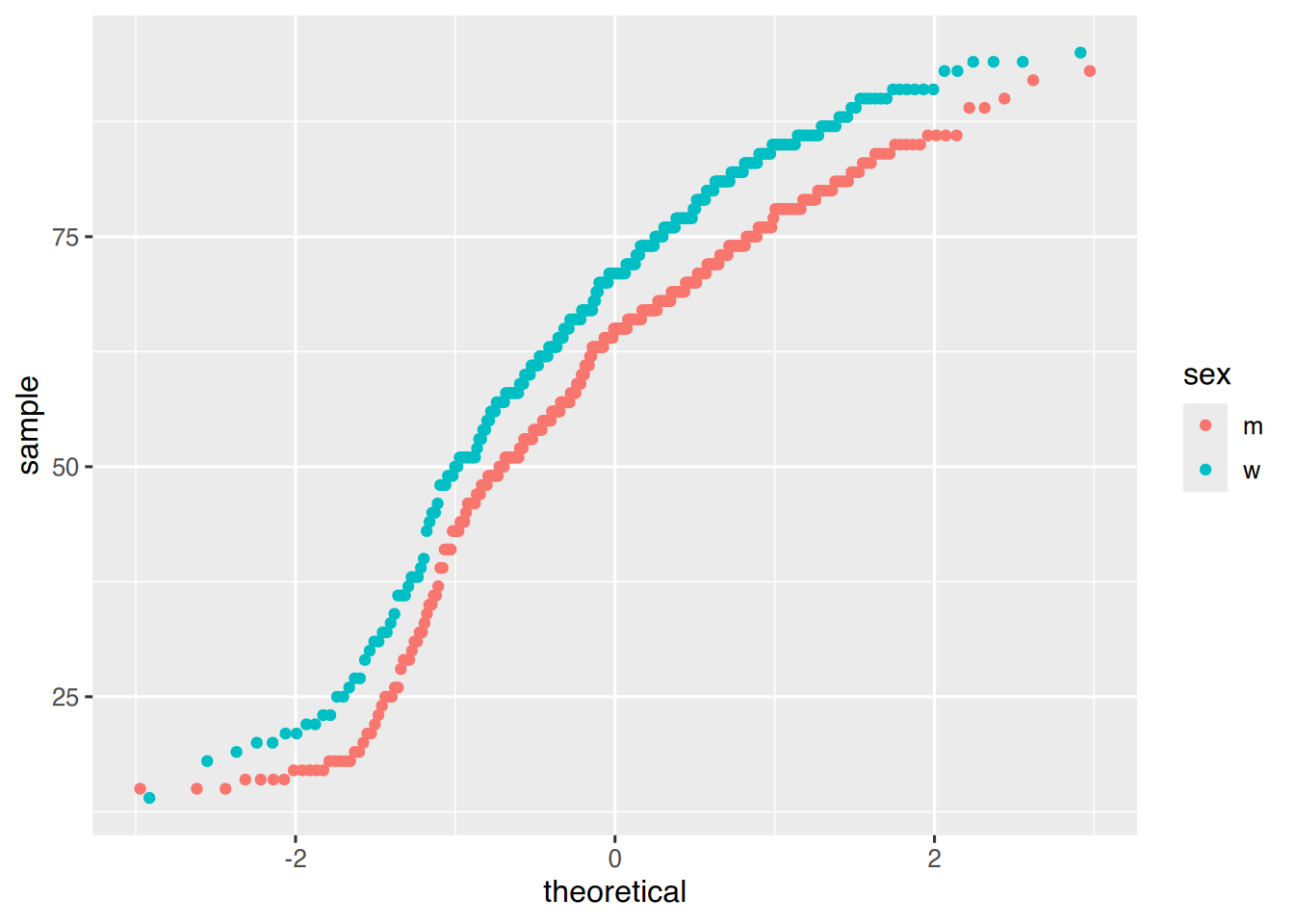
Mit Zusatzpaketen kann auch hier die Funktionalität erhöht werden. Beispielsweise ist im Paket ggpubr die Funktion ggqqplot() enthalten, die automatisch das Konfidenzintervall hinzufügt. Der Aufruf ist etwas anders als bei ggplot():
ggpubr::ggqqplot(epa, x="age", color="sex") 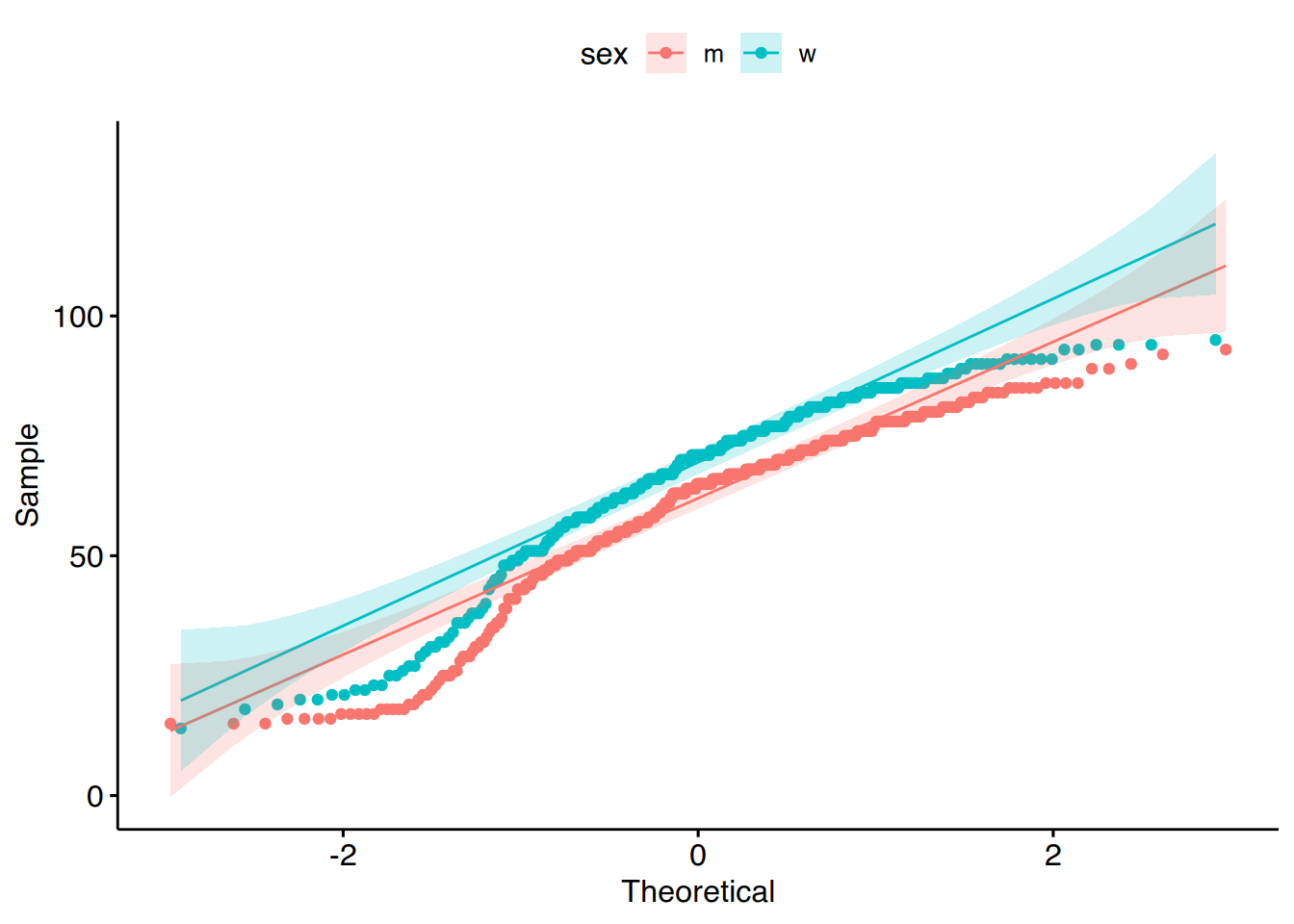
Weitere Möglichkeiten bietet das Zusatzpaket qqplotr. Es fügt weitere Geome und Funktionen für ggplot hinzu.
# aktiviere die Zusatzfunktionen
library(qqplotr)## Error in library(qqplotr): es gibt kein Paket namens 'qqplotr'ggplot(epa, aes(sample=age)) +
# zeichne Punkte wie im "Original"
stat_qq_point(aes(color=sex)) +
# füge Linie hinzu
stat_qq_line(aes(color=sex)) +
# füge Konfidenzgrenzen hinzu
stat_qq_band(aes(fill=sex))## Error in stat_qq_point(aes(color = sex)): konnte Funktion "stat_qq_point" nicht findenDieses Plot ist aber nicht ganz so “schön” wie die vorangegangenen. Es wird besser, wenn wir die Layerreihenfolge vertauschen und den Alpha-Wert der Farbe herbasetzen.
ggplot(epa, aes(sample=age)) +
# fange mit Konfidenzgrenzen an
# setzte Alpha-Wert für Farbe
stat_qq_band(aes(fill=sex), alpha=2/10) +
# füge Linie hinzu
stat_qq_line(aes(color=sex)) +
# zeichne Punkte wie im "Original"
stat_qq_point(aes(color=sex)) ## Error in stat_qq_band(aes(fill = sex), alpha = 2/10): konnte Funktion "stat_qq_band" nicht finden35.11 Diagramme speichern
35.11.1 R-Objekt
ggplot()-Diagramme können in R-Objekte (zwischen)gespeichert werden.
# Plot in Obkjekt "p" speichern
p <- ggplot(pf8) +
aes(x="", fill=Geschlecht) +
geom_bar(color="white")
# Plot anzeigen
p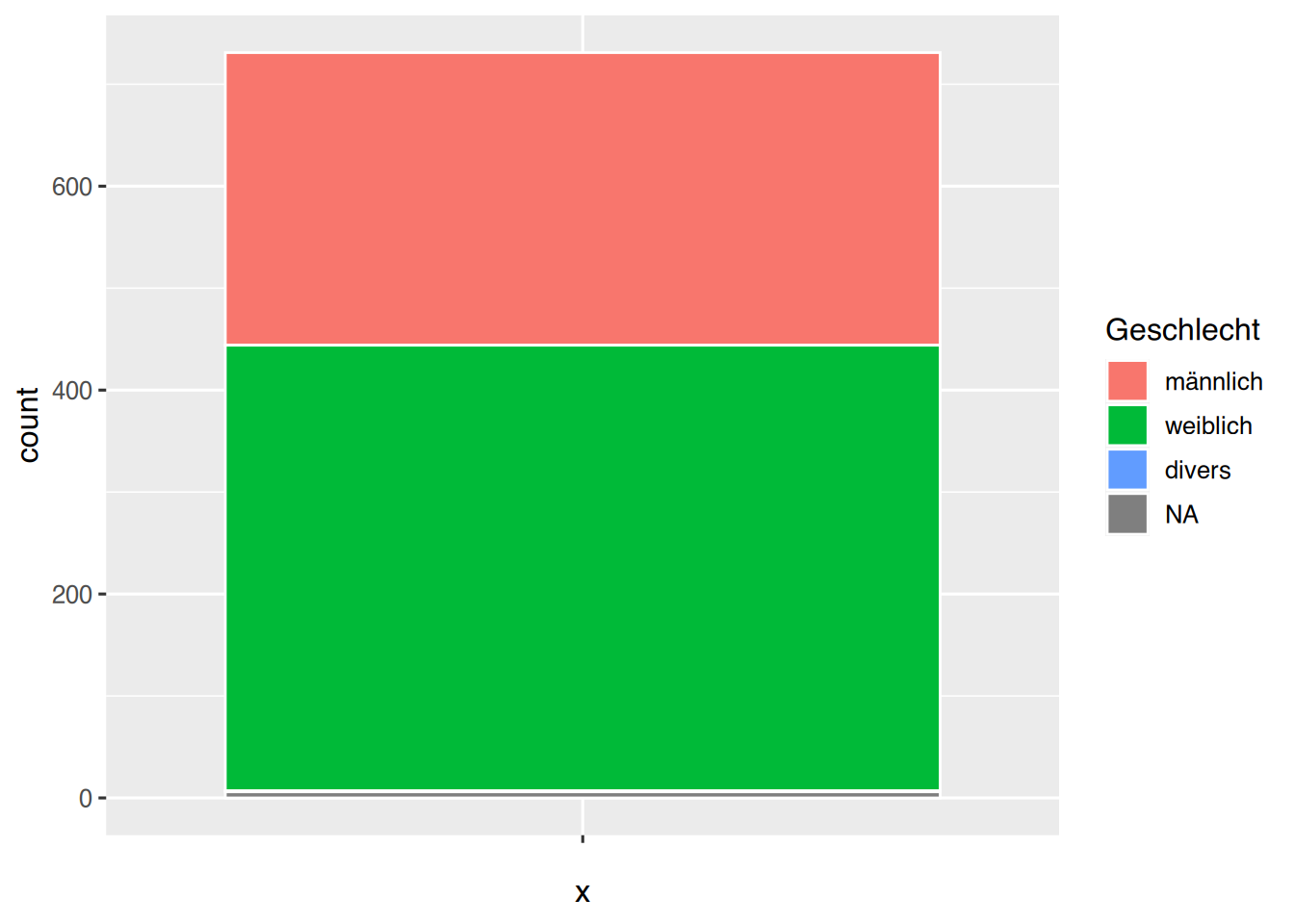
Das Objekt kann nun weitere Funktionen verarbeiten.
# Plot in Obkjekt "p" speichern
p <- ggplot(pf8) +
aes(x="", fill=Geschlecht) +
geom_bar(color="white")
# Plot weiter bearbeiten
p + coord_polar("y", start=0) +
theme_void()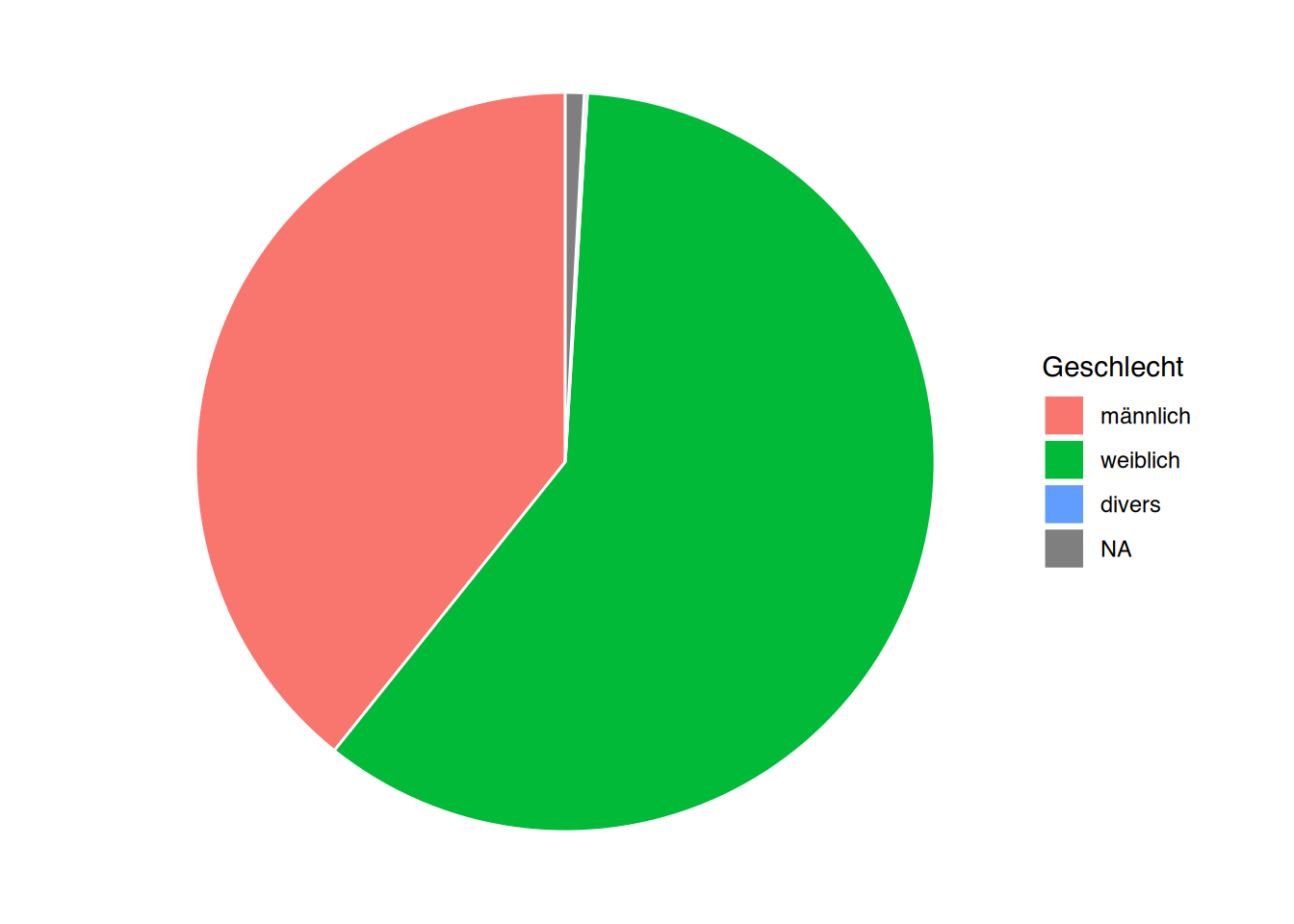
35.11.2 in Datei speichern
Mit der Funktion ggsave() können ggplot()-Diagramme als Datei gespeichert werden.
# Plot erzeugen
ggplot(pf8) +
aes(x="", fill=Geschlecht) +
geom_bar(color="white") +
coord_polar("y", start=0) +
theme_void()
# Plot in Datei speichern
ggsave("MeinPlot.png",
units="mm",
width=400,
height=200,
dpi=300,
pointsize=7)Liegt der Plot in einem R-Objekt, kann dies mit dem Parameter plot angegeben werden.
# Plot ist in Obkjekt "p" gespeichert
ggsave("MeinPlot.png",
plot= p,
units="mm",
width=400,
height=200,
dpi=300,
pointsize=7)35.12 Aussehen ändern
35.12.1 Titel und Überschriften
Die Plottitel können mit der Funktion labs() verändert werden.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue",
fill="skyblue") +
labs(title="Dies ist Überschrift",
subtitle="Dies ist die Unter-Überschrift",
caption="Dies ist die Bildunterschrift")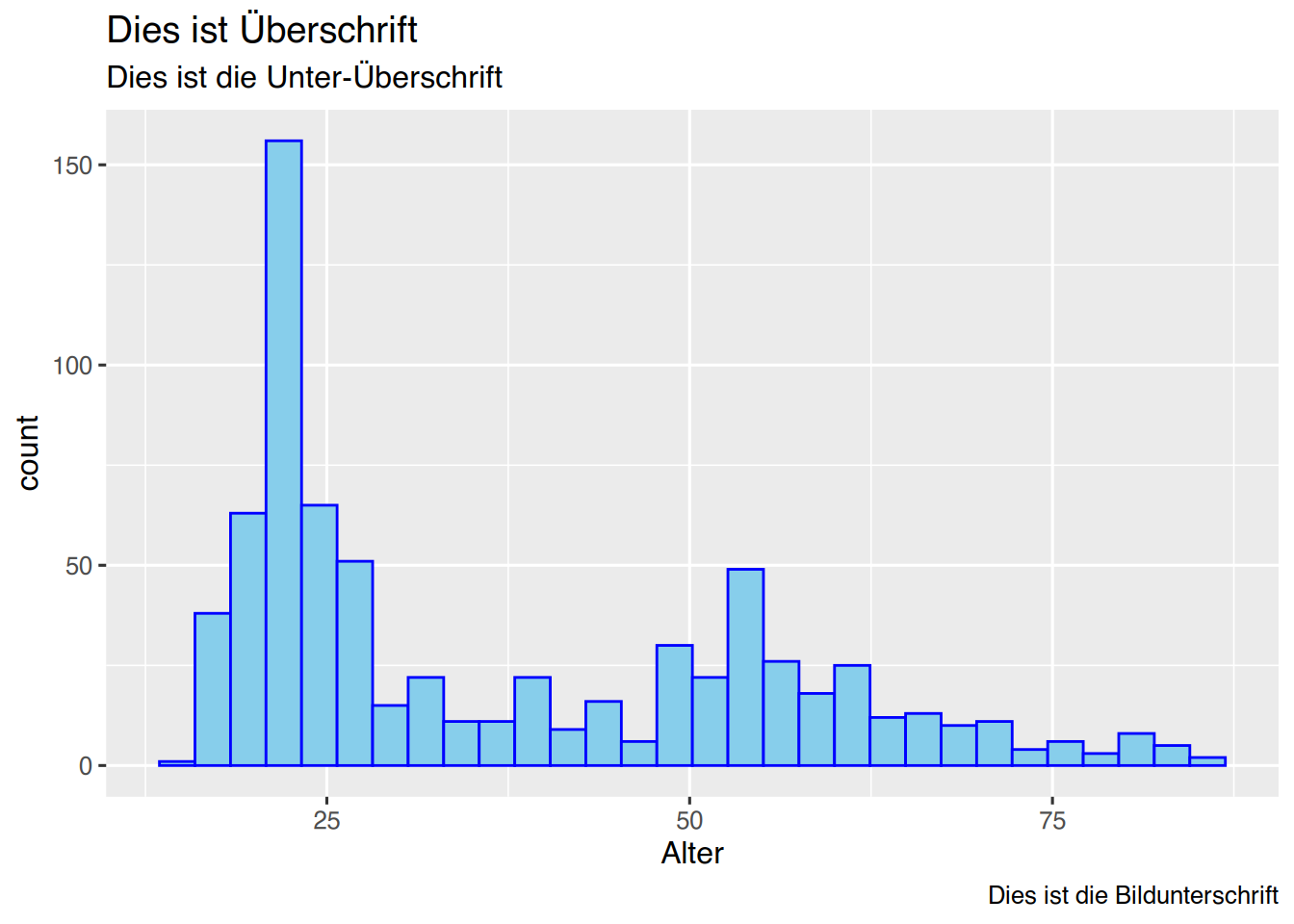
Die Schriftart kann mittels theme()-Funktion geändert werden.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue",
fill="skyblue") +
labs(title="Dies ist Überschrift",
subtitle="Dies ist die Unter-Überschrift in fett",
caption="Dies ist die Bildunterschrift in kursiv") +
theme(plot.title = element_text(size=18)) +
theme(plot.subtitle = element_text(size=10, face="bold")) +
theme(plot.caption = element_text(size=8, face="italic"))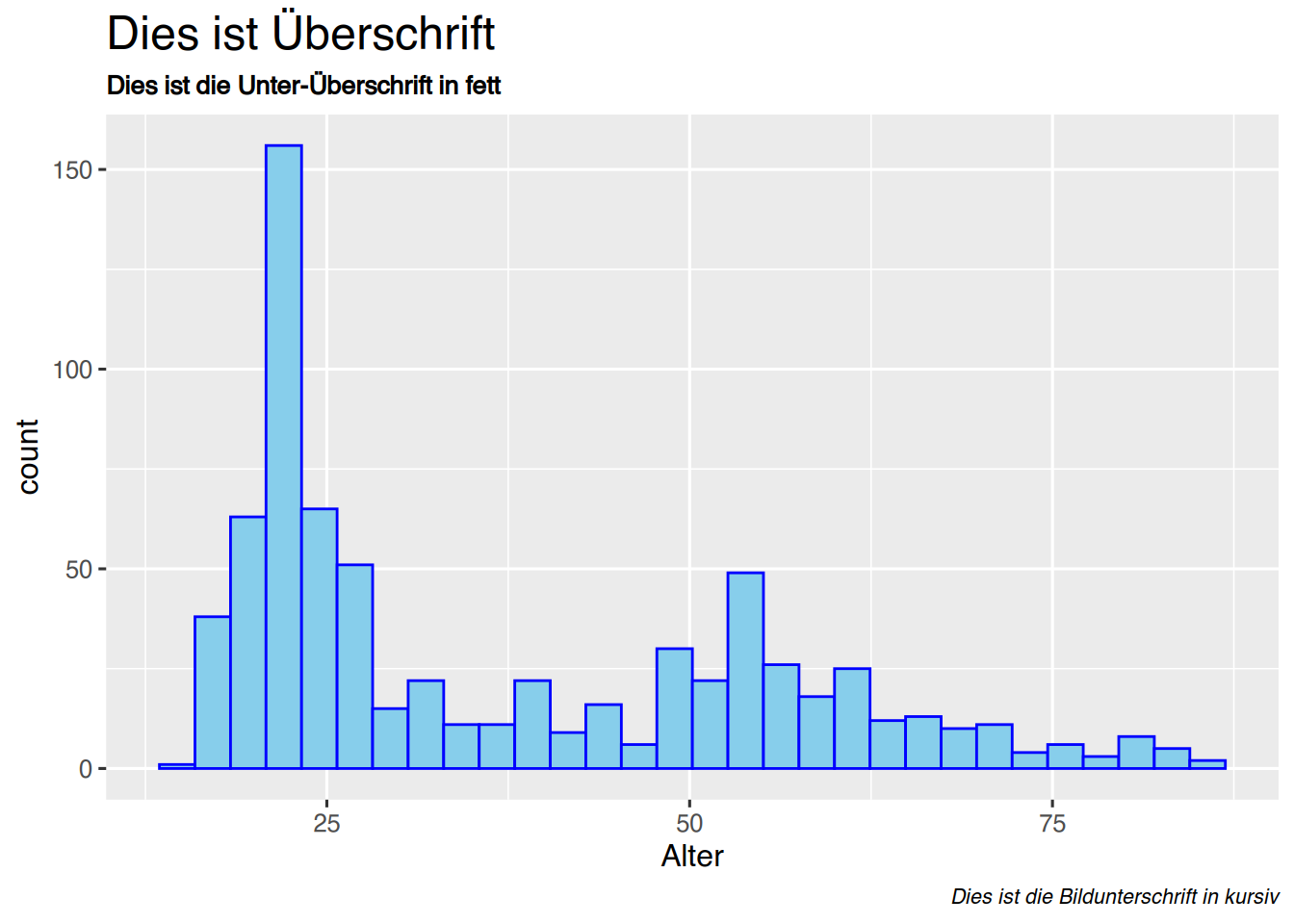
35.12.2 Achsen
Die Achsenbeschriftung kann mittels xlab() und ylab() verändert werden.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue",
fill="skyblue") +
xlab("Ich bin die x-Achse") +
ylab("Ich bin die y-Achse")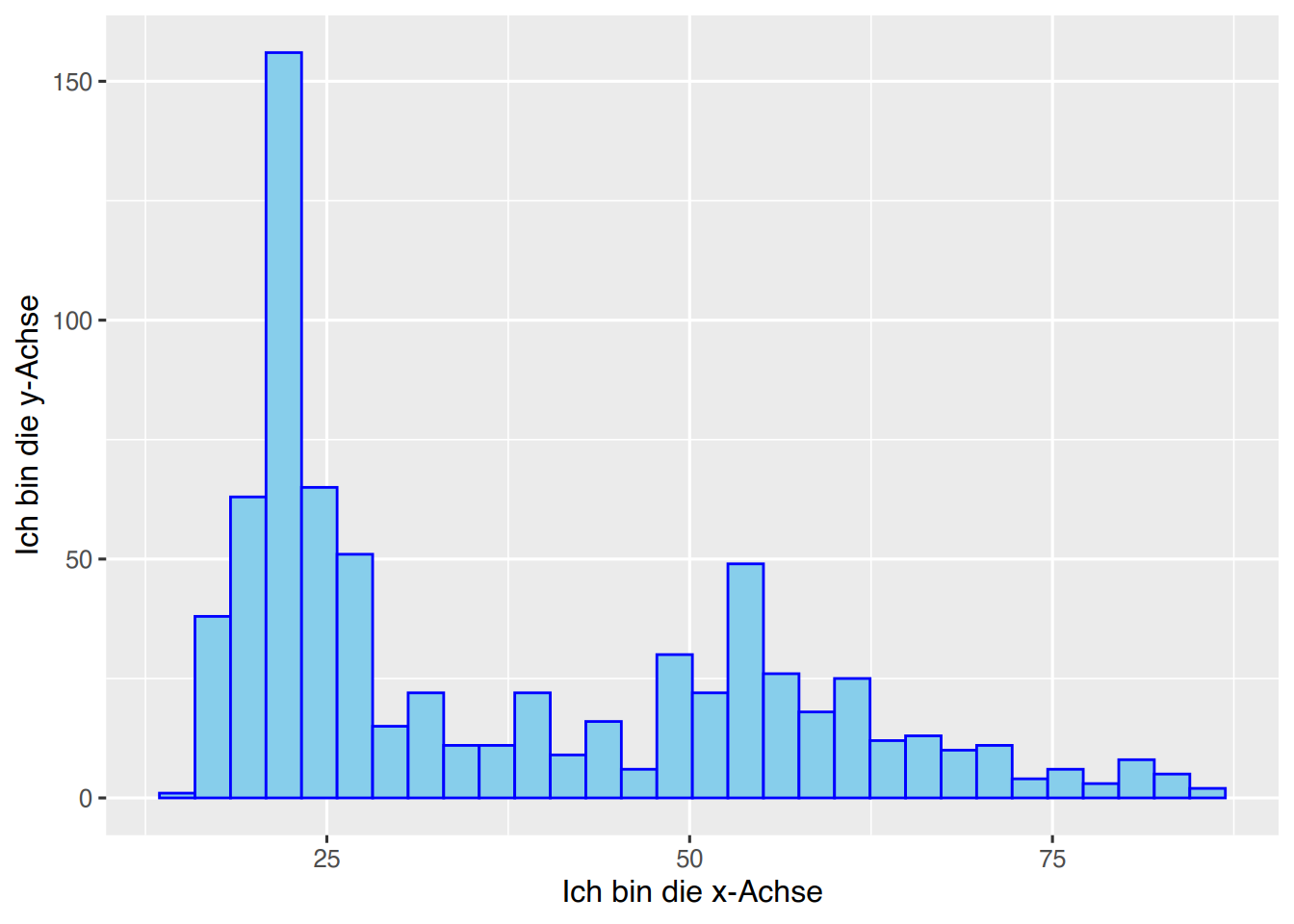
Die Längen der Achsenausschnitte können mit xlim() und ylim() angegeben werden.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue",
fill="skyblue") +
xlab("Ich bin die x-Achse") +
ylab("Ich bin die y-Achse")+
xlim(20, 50) +
ylim(-20, 75)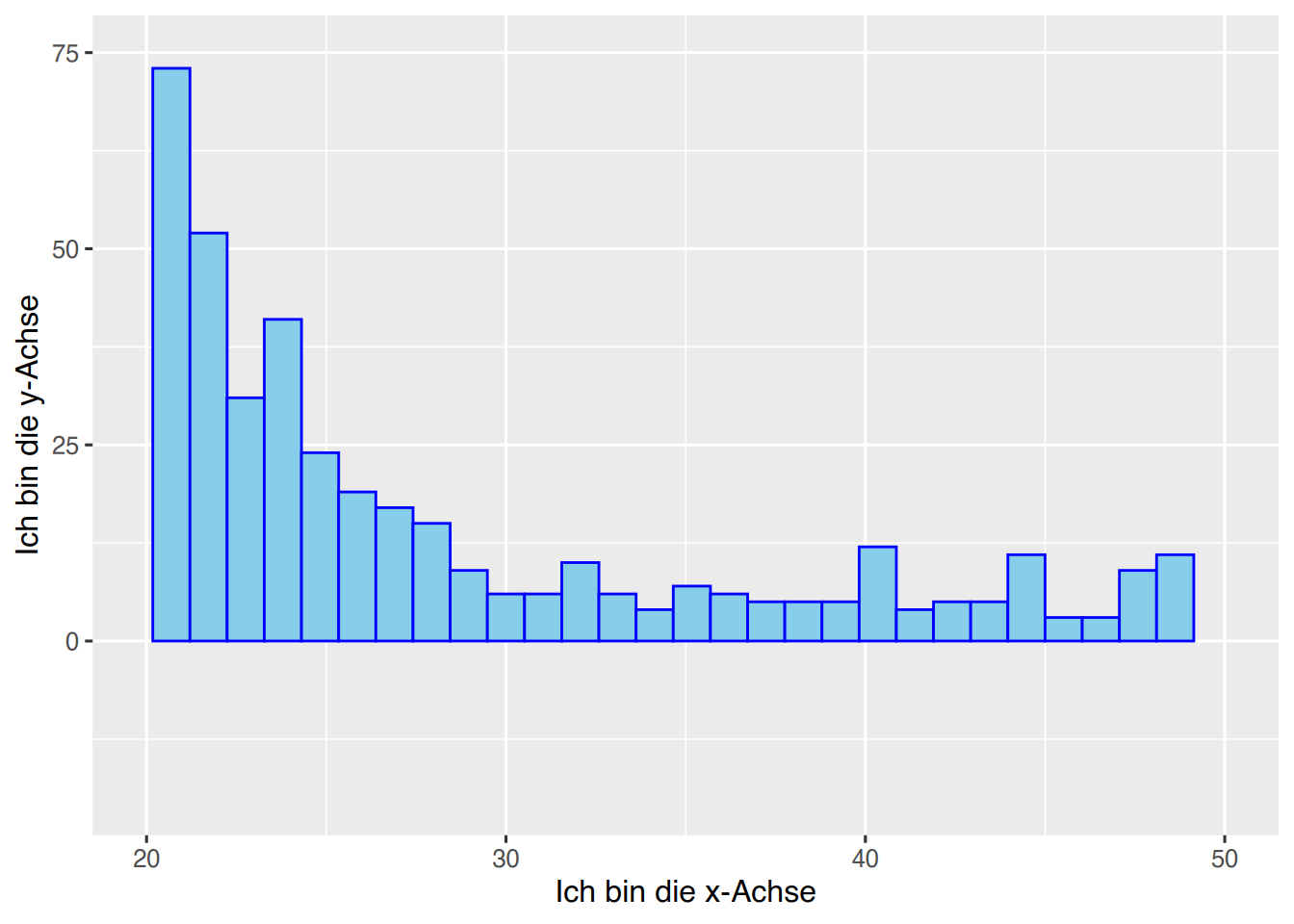
Die Anzahl und Position der Achsenmarkierungen können mit scale_x_continuous() und scale_y_continuous() angepasst werden.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue",
fill="skyblue") +
xlab("Ich bin die x-Achse") +
ylab("Ich bin die y-Achse")+
xlim(20, 50) +
ylim(-20, 75) +
scale_x_continuous(breaks = c(20, 25, 30, 35, 40)) +
scale_y_continuous(breaks = seq(-20, 100, 10) )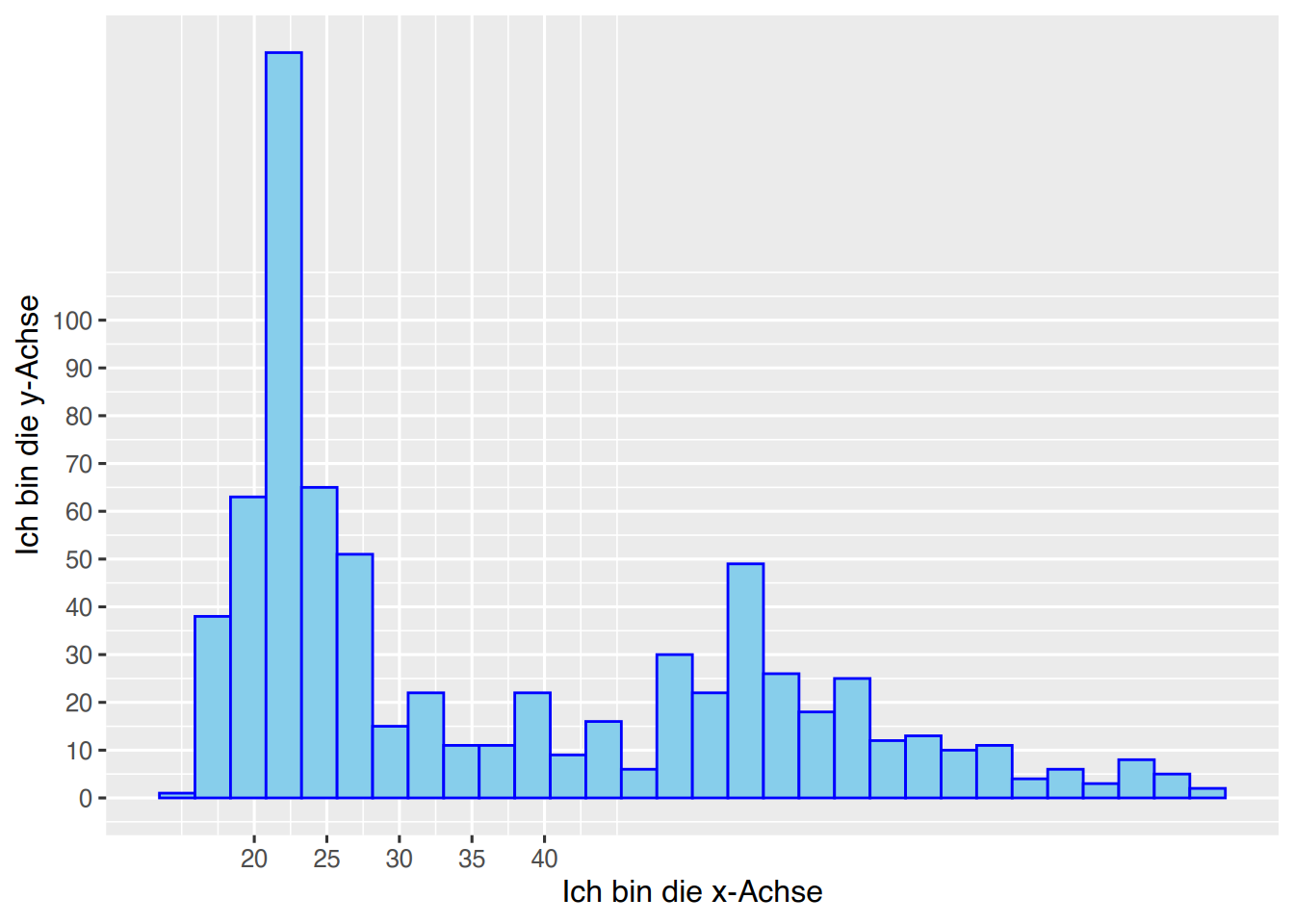
Die Schriftarten werden ebenfalls mit der theme()-Funktion gesteuert.
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue",
fill="skyblue") +
xlab("Ich bin die x-Achse") +
ylab("Ich bin die y-Achse")+
xlim(20, 50) +
ylim(-20, 75) +
scale_x_continuous(breaks = c(20, 25, 30, 35, 40)) +
scale_y_continuous(breaks = seq(-20, 100, 10) ) +
theme(axis.title.x = element_text(size=18, face="bold")) +
theme(axis.title.y = element_text(size=14, face="italic")) +
theme(axis.text.x = element_text(size=8, color="red")) +
theme(axis.text.y = element_text(size=4, color="blue")) +
theme(axis.ticks.x = element_line(size=8, color="darkgreen")) +
theme(axis.ticks.y = element_line(size=8, color="orange"))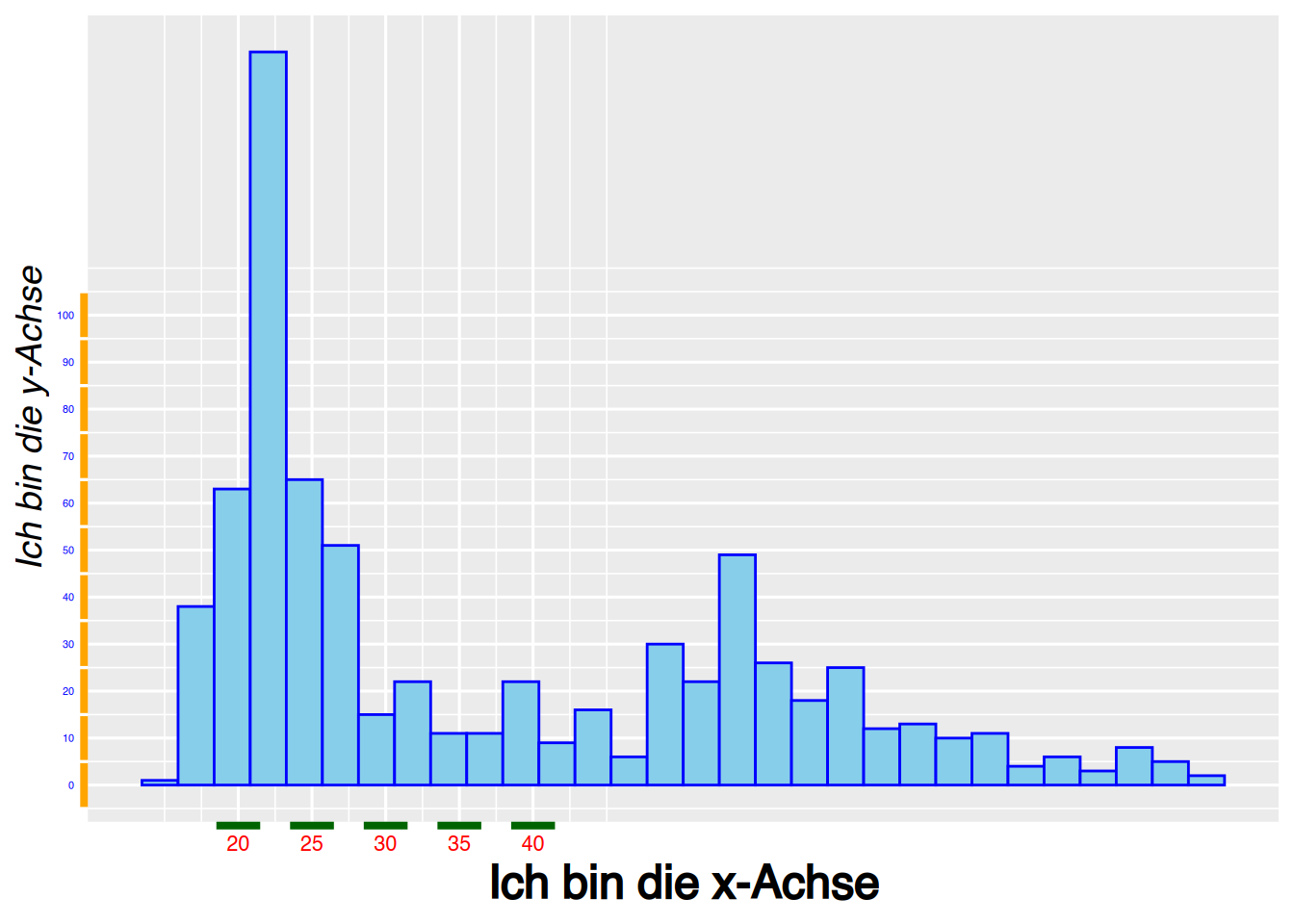
So können die Achsenbeschriftungen auch über den Parameter angle gedreht werden.
ggplot(pf8) +
aes(x=SportWarum ) +
geom_bar(color="blue",
fill="skyblue") +
theme(axis.text.x = element_text(angle = 45,
hjust = 1,
size=10))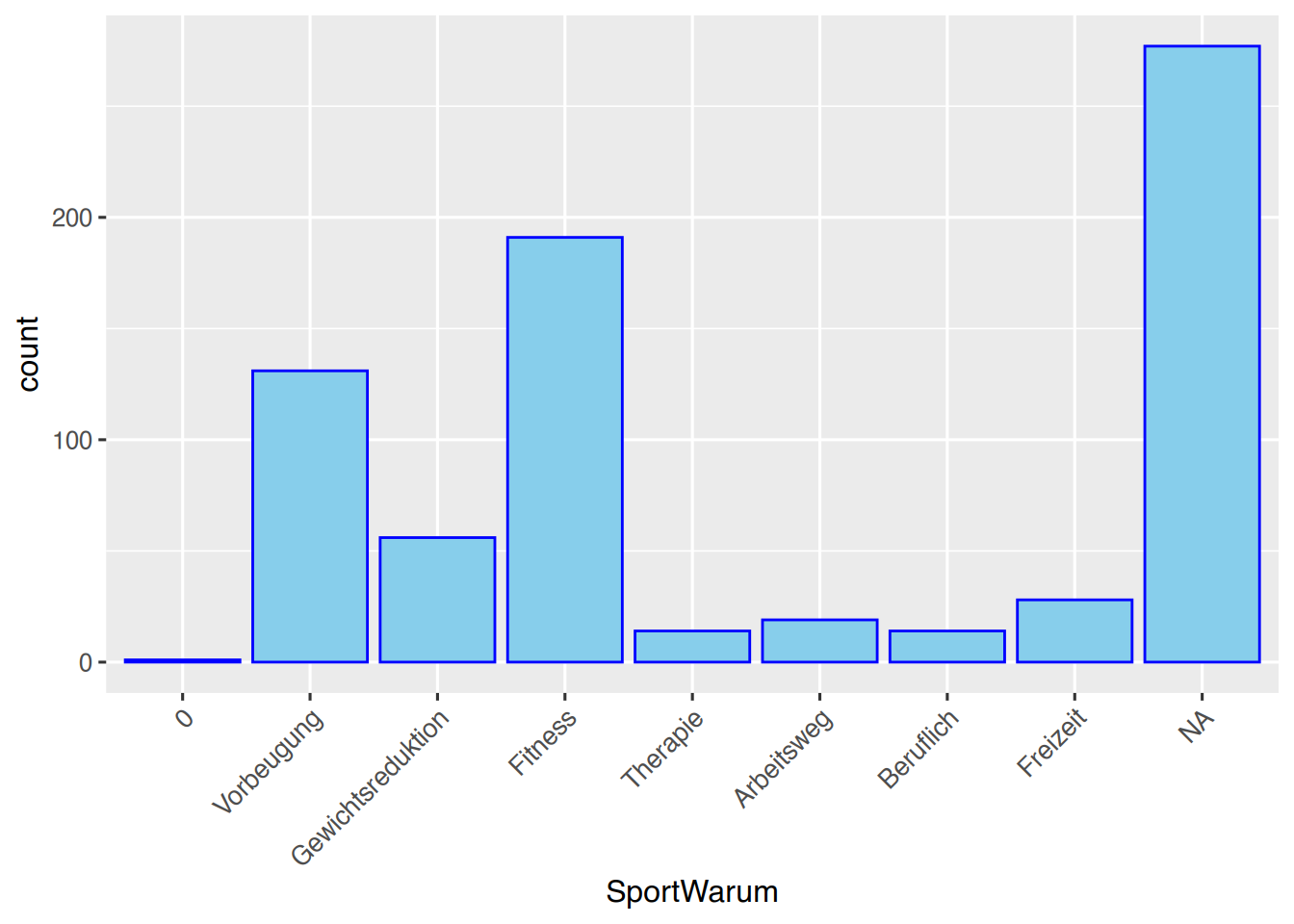
Sollen die Ticks und ihre Striche vollständig entfernt werden, so geht dies mit den Funktionen theme() und element_blank().
ggplot(pf8) +
aes(x=Alter) +
geom_histogram(color="blue",
fill="skyblue") +
theme(axis.text.x=element_blank(),
axis.ticks.x=element_blank(),
axis.text.y=element_blank(),
axis.ticks.y=element_blank())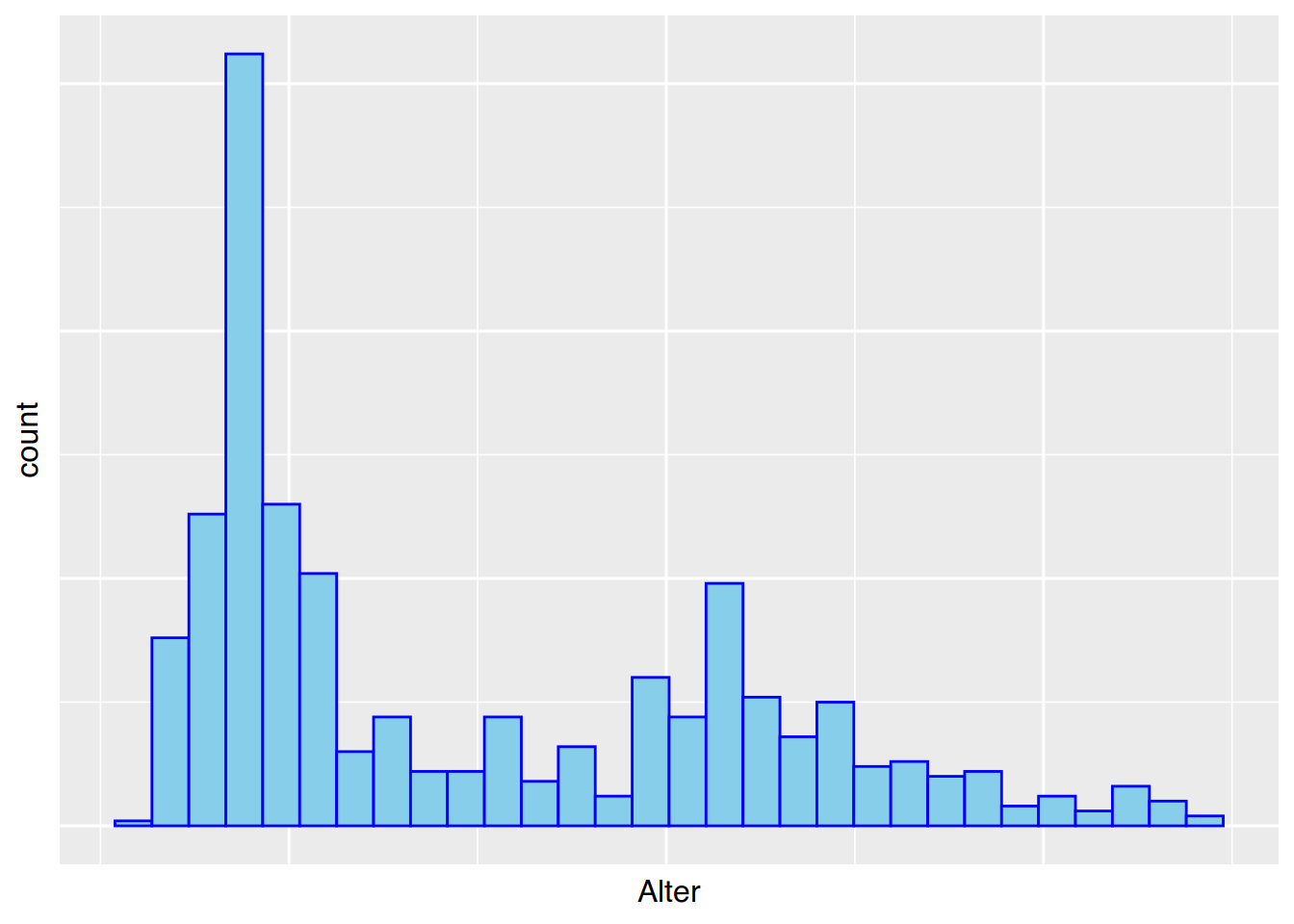
35.12.3 Legendenbox
Der Title der Legendbox kann prinzipiell geändert werden, indem die ursprüngliche Variable umbenannt wird. Dann ändert sich der Legendenboxtitel ebenfalls entsprechend. Sollte dies keine “gute” Lösung sein, z.B. weil mehrere Wörter als Titel verwendet werden sollen, kann der Title über die labs()-Funktion geändert werden. Hierbei besteht eine Abhängigkeit zur Gruppierung über die aes()-Funktion. Im folgenden Beispiel wird die Gruppierung über den Parameter fill=Geschlecht erzeugt. Daher muss der Legendentitel mittels labs(fill="NEU") geändert werden.
pf8 %>%
ggplot() +
aes(y = Alter,
fill= Geschlecht) +
geom_boxplot() +
labs(fill="Neuer Titel")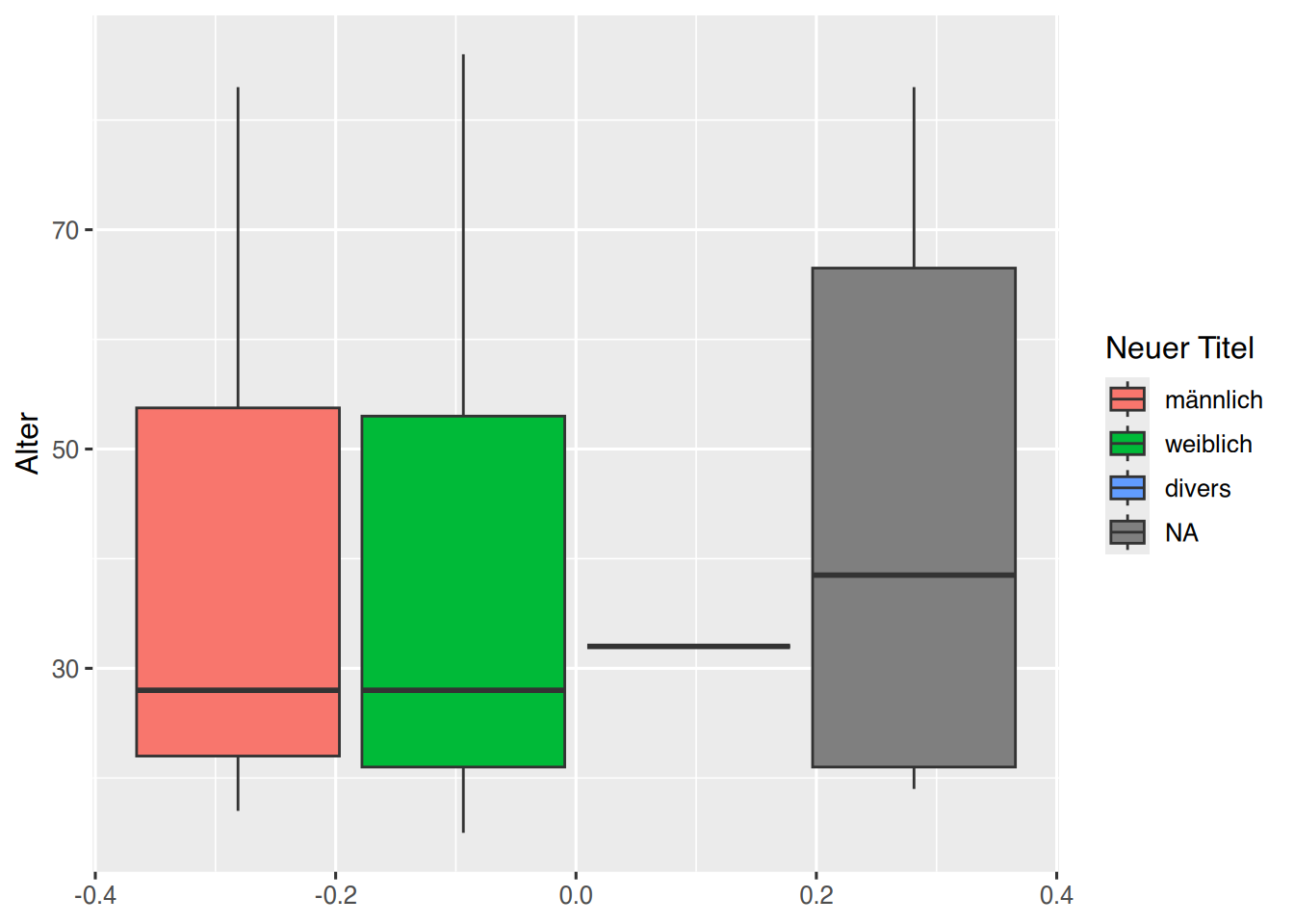
Sollen die Einträge der Legenbox geändert werden, kann dies zum einen erfolgen, indem die Levels des Originalfactor entsprechend umgeschrieben werden. Dann ändern sich auch die Einträge in der Legendenbox. Sollte dies keine “gute” Lösung sein, können die Einträge von Hand über die Funktion scale_fill_discrete() überschrieben werden.
pf8 %>%
ggplot() +
aes(y = Alter,
fill= Geschlecht) +
geom_boxplot() +
labs(fill="Neuer Titel") +
scale_fill_discrete(labels=c('men', 'woman', 'divers', 'no answer'))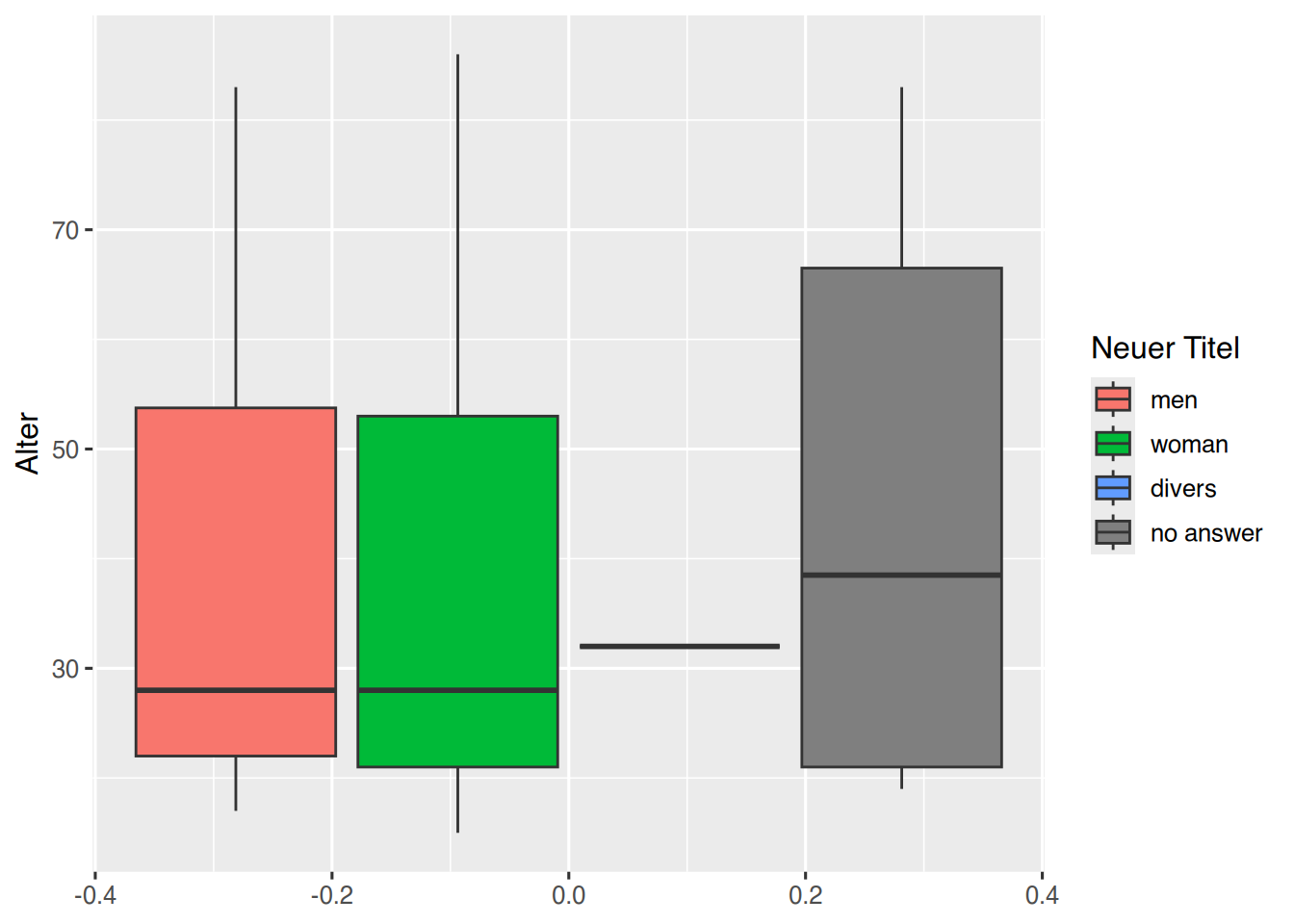
Soll die Reihenfolge der Levelboxelemte geändert werden, kann ebenfalls auf die Funktion scale_fill_discrete() zurückgegriffen werden.
pf8 %>%
ggplot() +
aes(y = Alter,
fill= Geschlecht) +
geom_boxplot() +
labs(fill="Neuer Titel") +
scale_fill_discrete(breaks=c(NA, "weiblich", "divers", "männlich"))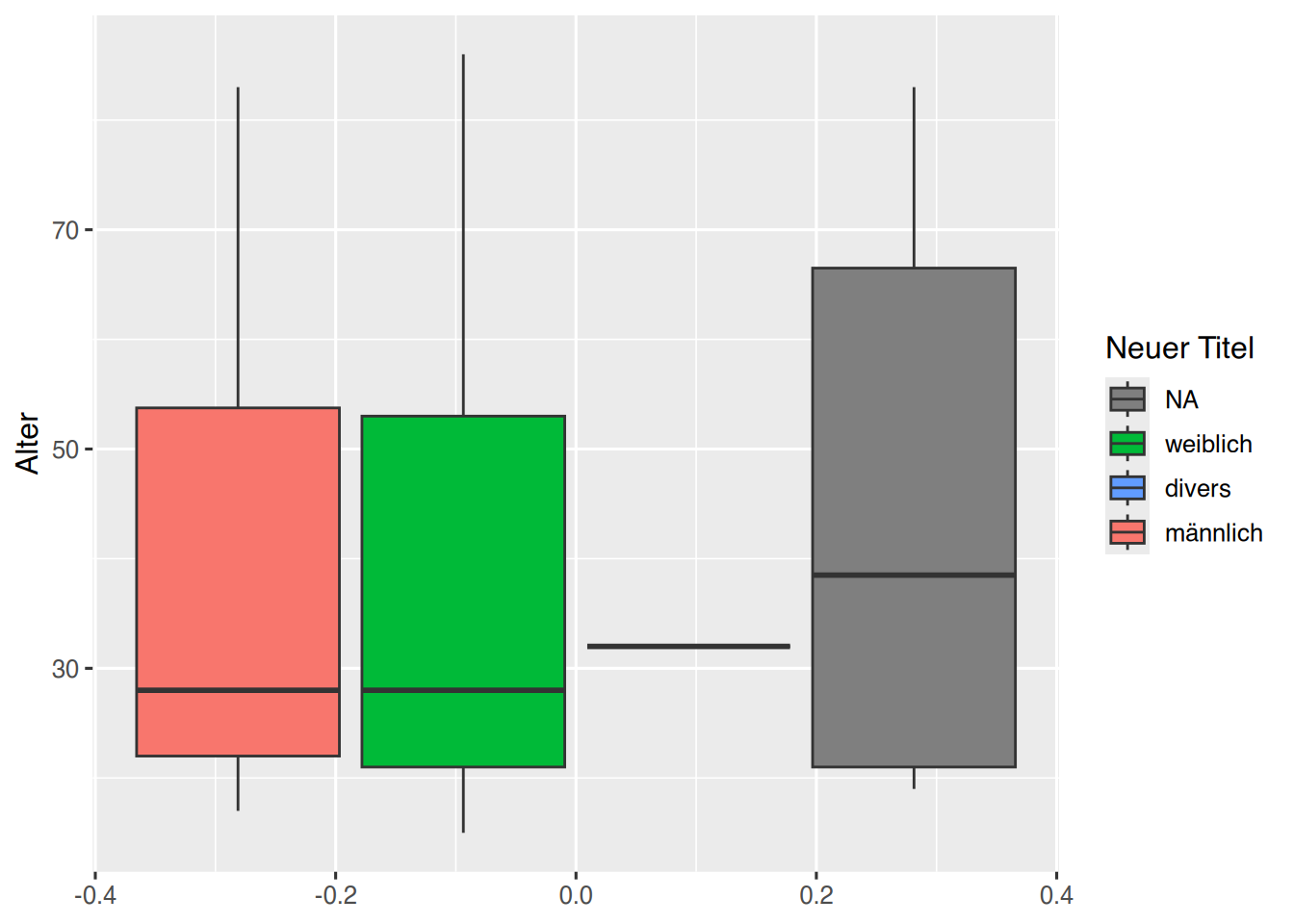
Die Schriftarten der Legendenbox können ebenfalls mit der theme()-Funktion verändert werden.
pf8 %>%
ggplot() +
aes(y = Alter,
fill= Geschlecht) +
geom_boxplot() +
theme(legend.title = element_text(color="red", face="bold")) +
theme(legend.text = element_text(color="blue", face="italic"))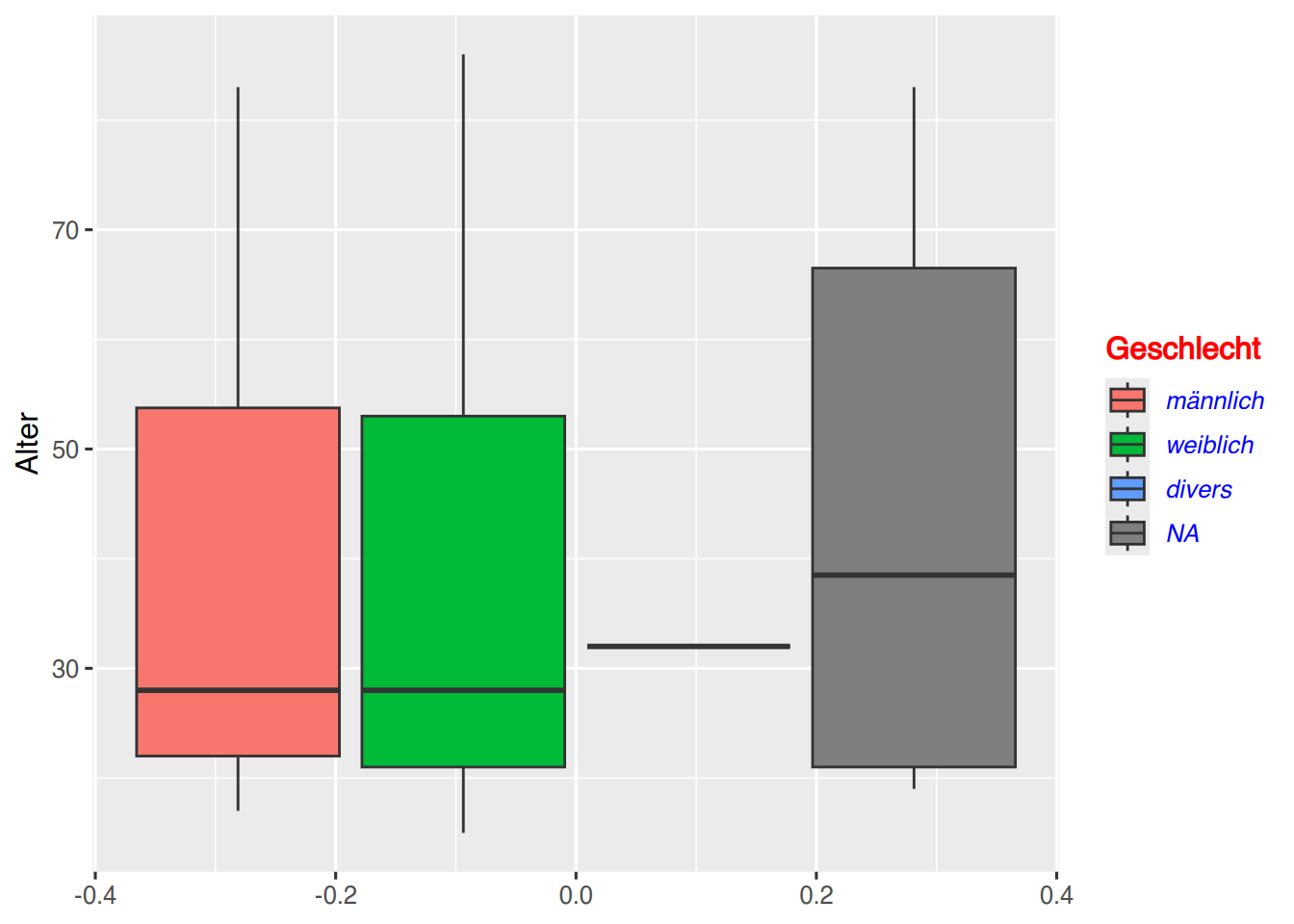
Soll die Legendenbox vollständig entfernt werden, so geht dies über die Funktion theme(legend.position = "none")
pf8 %>%
ggplot() +
aes(y = Alter,
fill= Geschlecht) +
geom_boxplot() +
theme(legend.position = "none")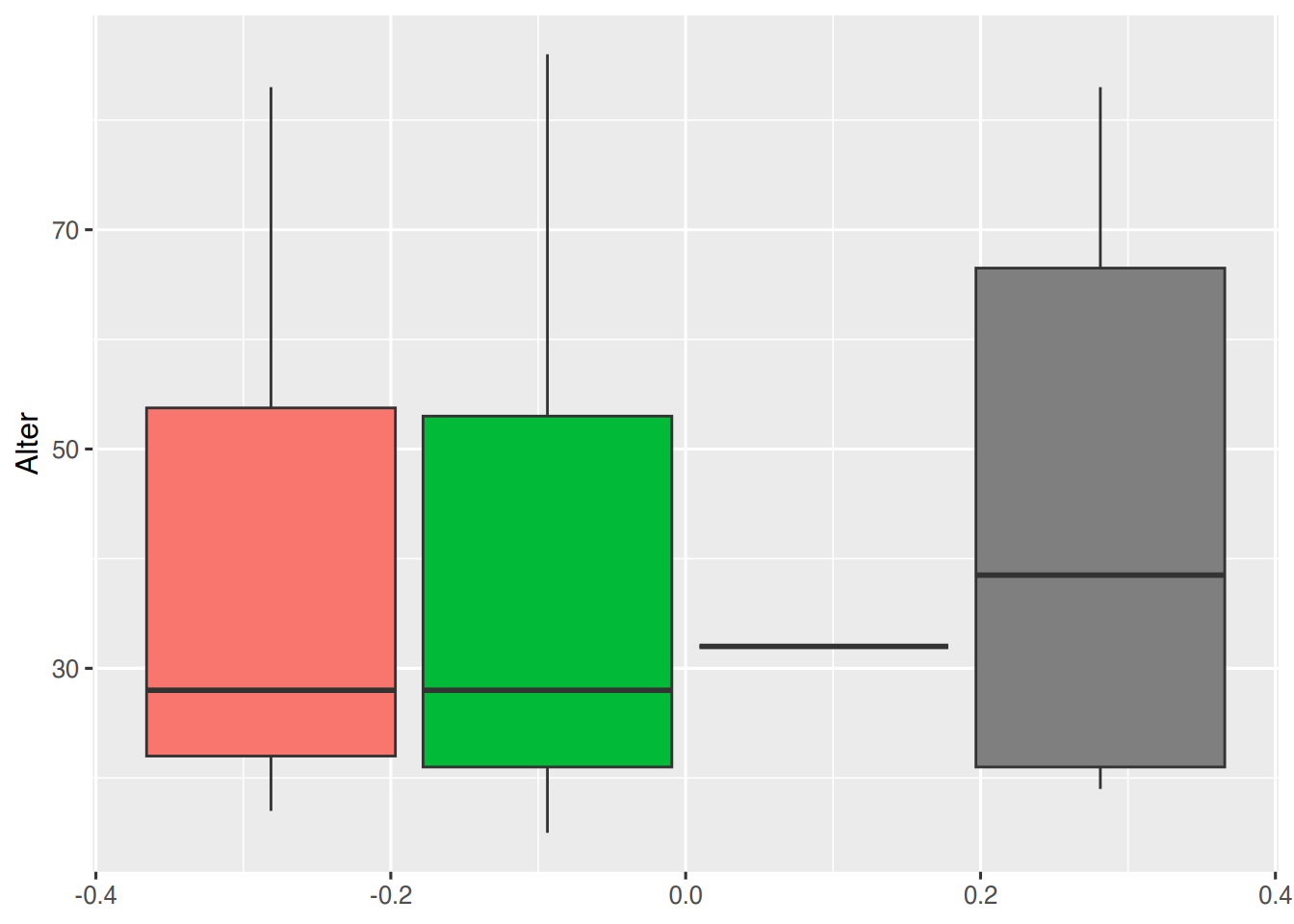
35.12.4 Farben
Ohne weitere Angaben verwendet ggplot() seine “eingebauten” Standardfarben.
Wenn wir das Alter der Probanden nach Standort gruppieren, wählt ggplot() diese Farben aus:
pf8 %>%
ggplot() +
aes(y = Alter,
color= Standort,
fill= Standort) +
geom_boxplot()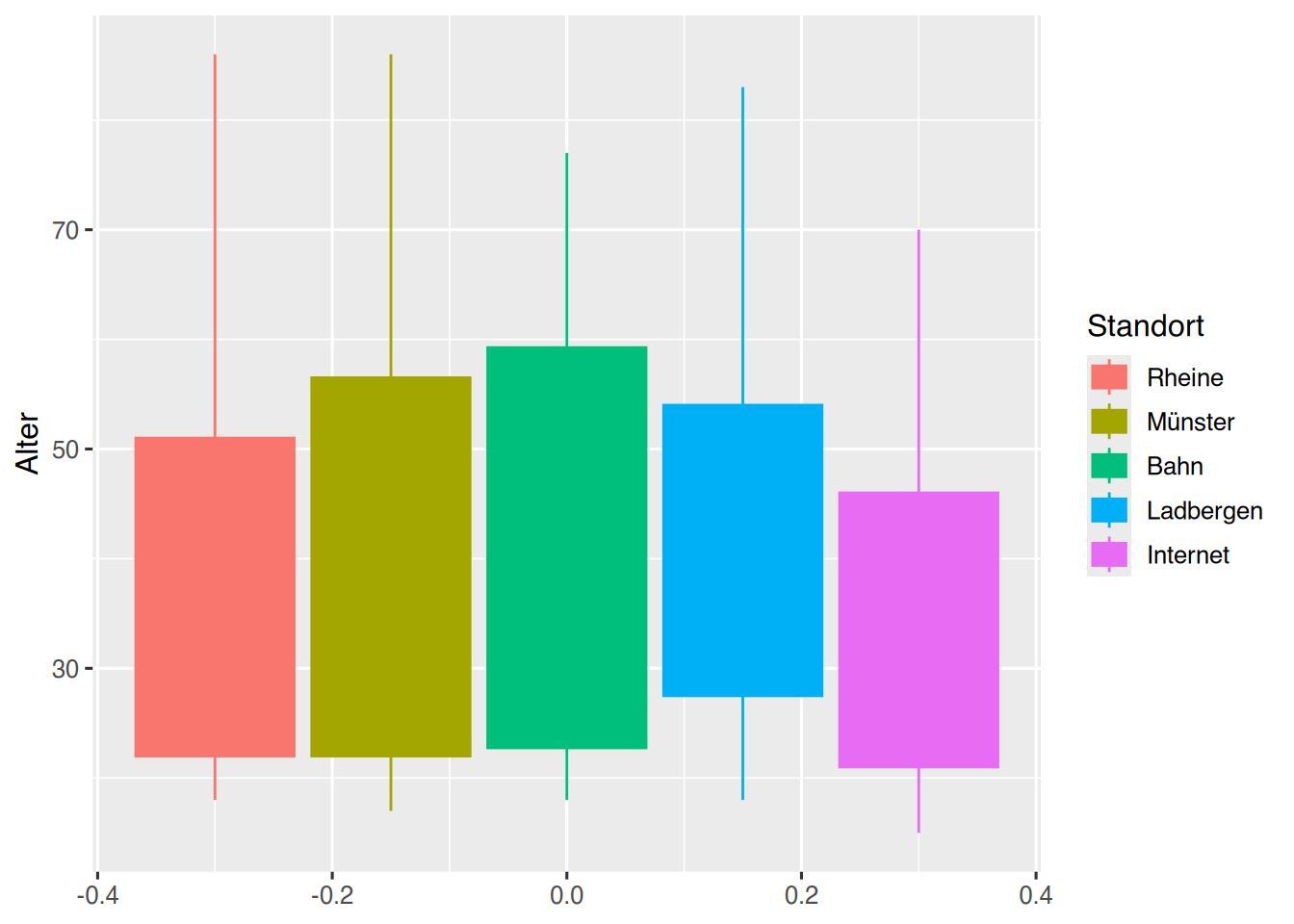
Die Farbintensität und -helligkeit der Standardfarben können über die Funktionen scale_color_hue() und scale_fill_hue() angepasst werden. Hierbei steht der Parameter c für die Farbintensität, und l für die Helligkeit.
pf8 %>%
ggplot() +
aes(y = Alter,
color= Standort,
fill= Standort) +
geom_boxplot() +
scale_color_hue(l=40, c=35) +
scale_fill_hue(l=40, c=35) 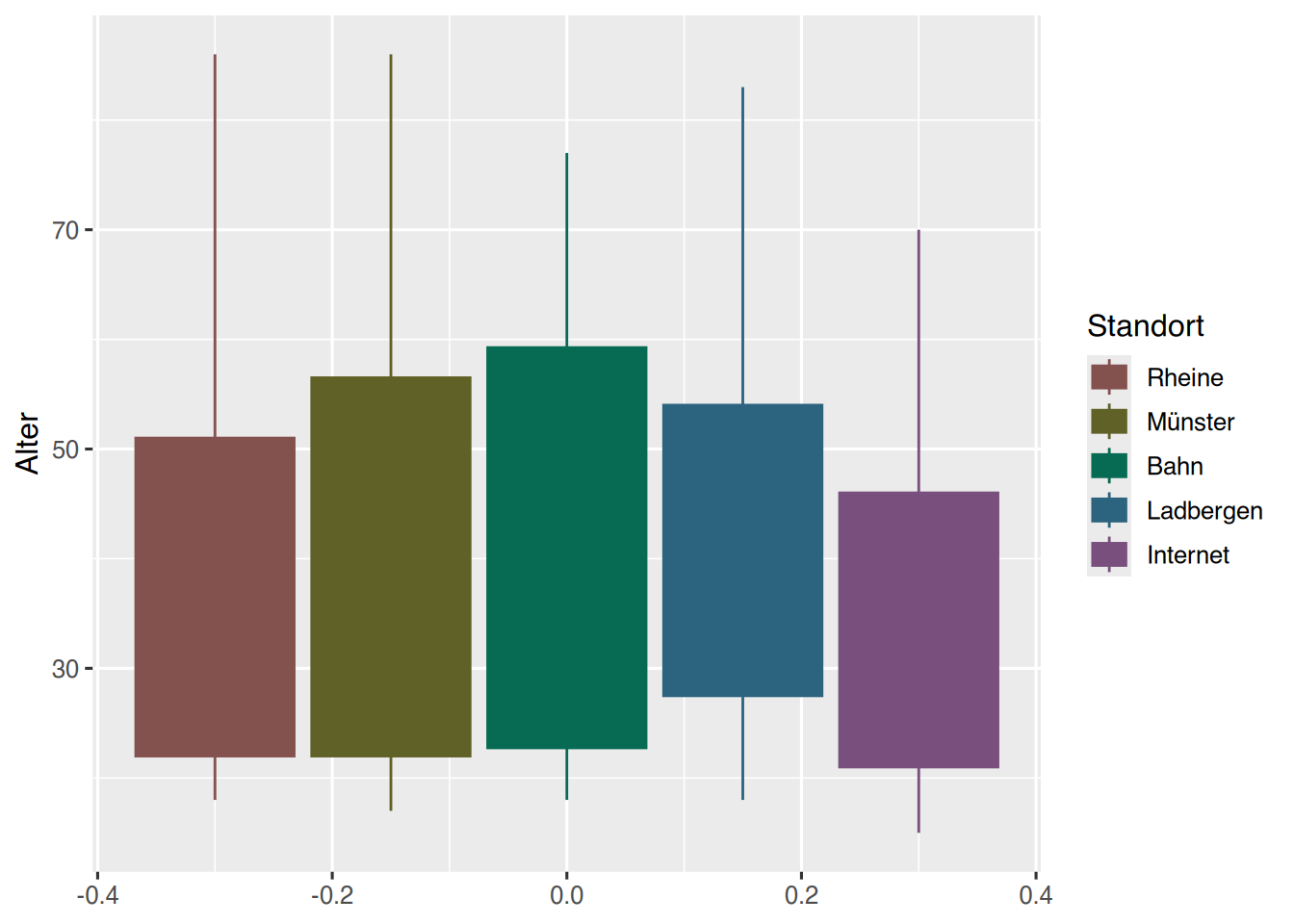
Über die Funktionen scale_color_manual() und scale_fill_manual() können die Farben manuell ausgewählt werden (eine Liste aller Farbnamen liefert die Funktion colors()):
pf8 %>%
ggplot() +
aes(y = Alter,
color= Standort,
fill= Standort) +
geom_boxplot() +
scale_color_manual(values=c("darkblue", "navy", "blue",
"skyblue2", "skyblue3")) +
scale_fill_manual(values=c("hotpink", "pink", "pink3",
"red3", "red4"))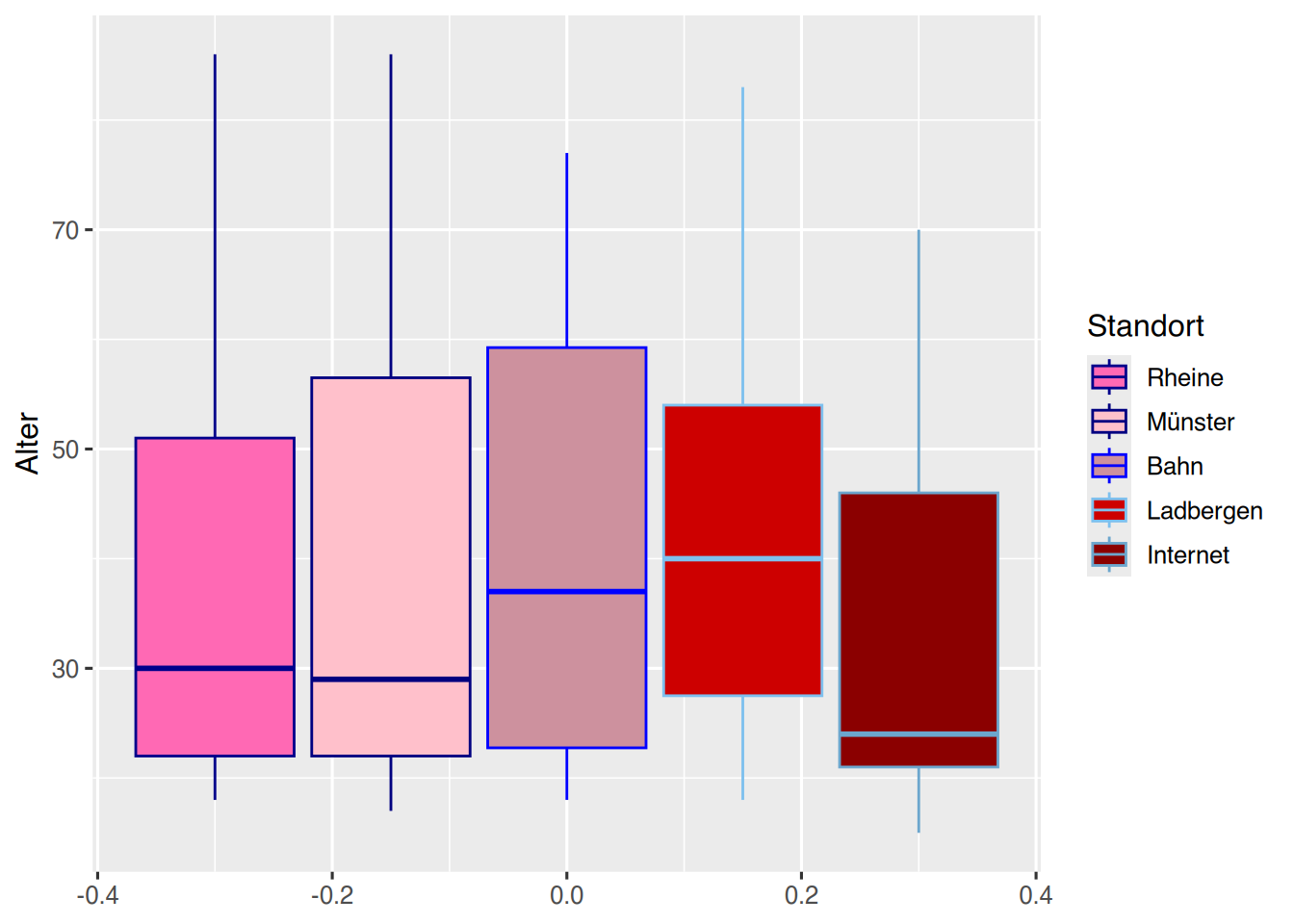
Die Farben können über den breaks-Parameter auch direkt den Levelausprägungen zugewiesen werden.
pf8 %>%
ggplot() +
aes(y = Alter,
color= Standort,
fill= Standort) +
geom_boxplot() +
scale_color_manual(values=c("darkblue", "navy", "blue",
"skyblue2", "skyblue3"),
breaks=c("Internet", "Ladbergen", "Münster",
"Rheine", "Bahn")) +
scale_fill_manual(values=c("hotpink", "pink", "pink3",
"red3", "red4"),
breaks=c("Internet", "Ladbergen", "Münster",
"Rheine", "Bahn"))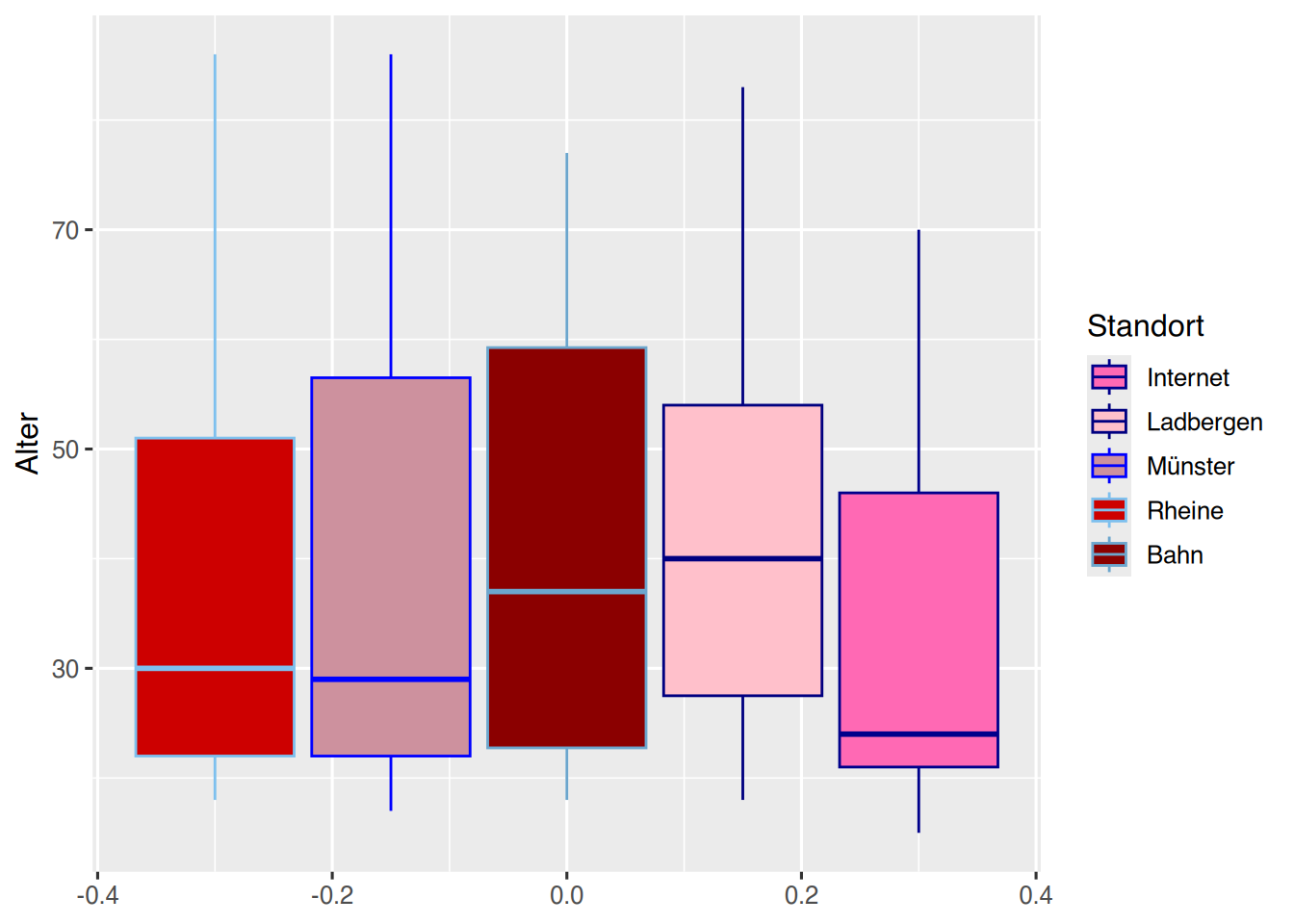
Hierdurch hat sich auch die Reihenfolge der Levelboxelemte geändert.
Soll die Reihenfolge der Levelboxelemte geändert werden, ohne dass eigene Farben definiert werden, kann auf die Funktion scale_fill_discrete() zurückgegriffen werden.
pf8 %>%
ggplot() +
aes(y = Alter,
fill= Standort) +
geom_boxplot() +
scale_fill_discrete(breaks=c("Internet", "Ladbergen", "Bahn",
"Rheine", "Münster"))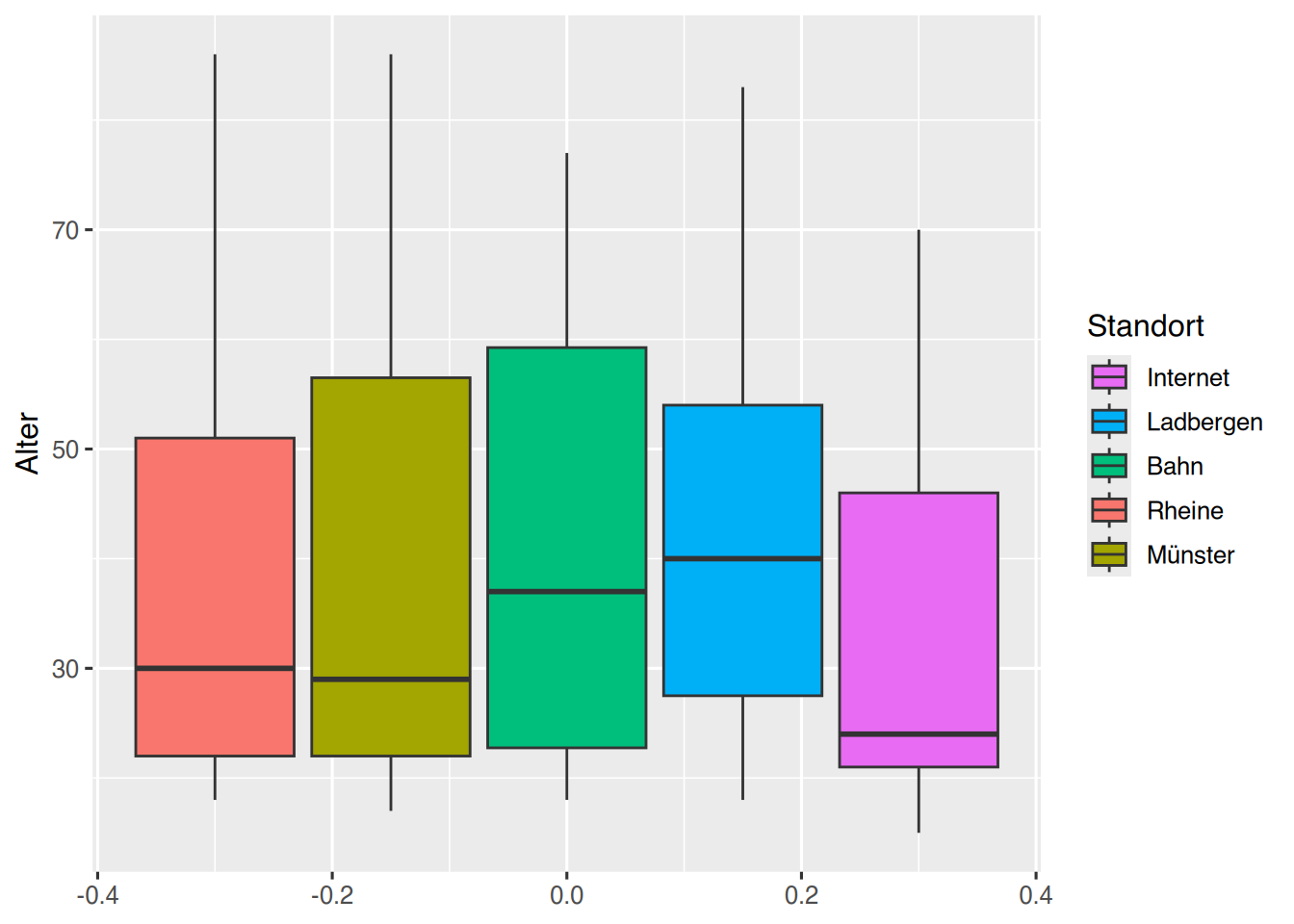
35.12.5 Linien
Mit den Funktionen geom_vline() und geom_hline() können vertikale und horizontale Linien hinzugefügt werden. Mit den Parametern xintercept und yintercept wird der Schnitt durch die jeweilige Achse festegelegt.
ggplot(pf8) +
aes(x = Alter,
y = Gewicht) +
geom_point() +
geom_hline(yintercept = 60,
linetype = "solid",
color = "blue",
size = 3) +
geom_vline(xintercept=40,
linetype="dashed",
color="red")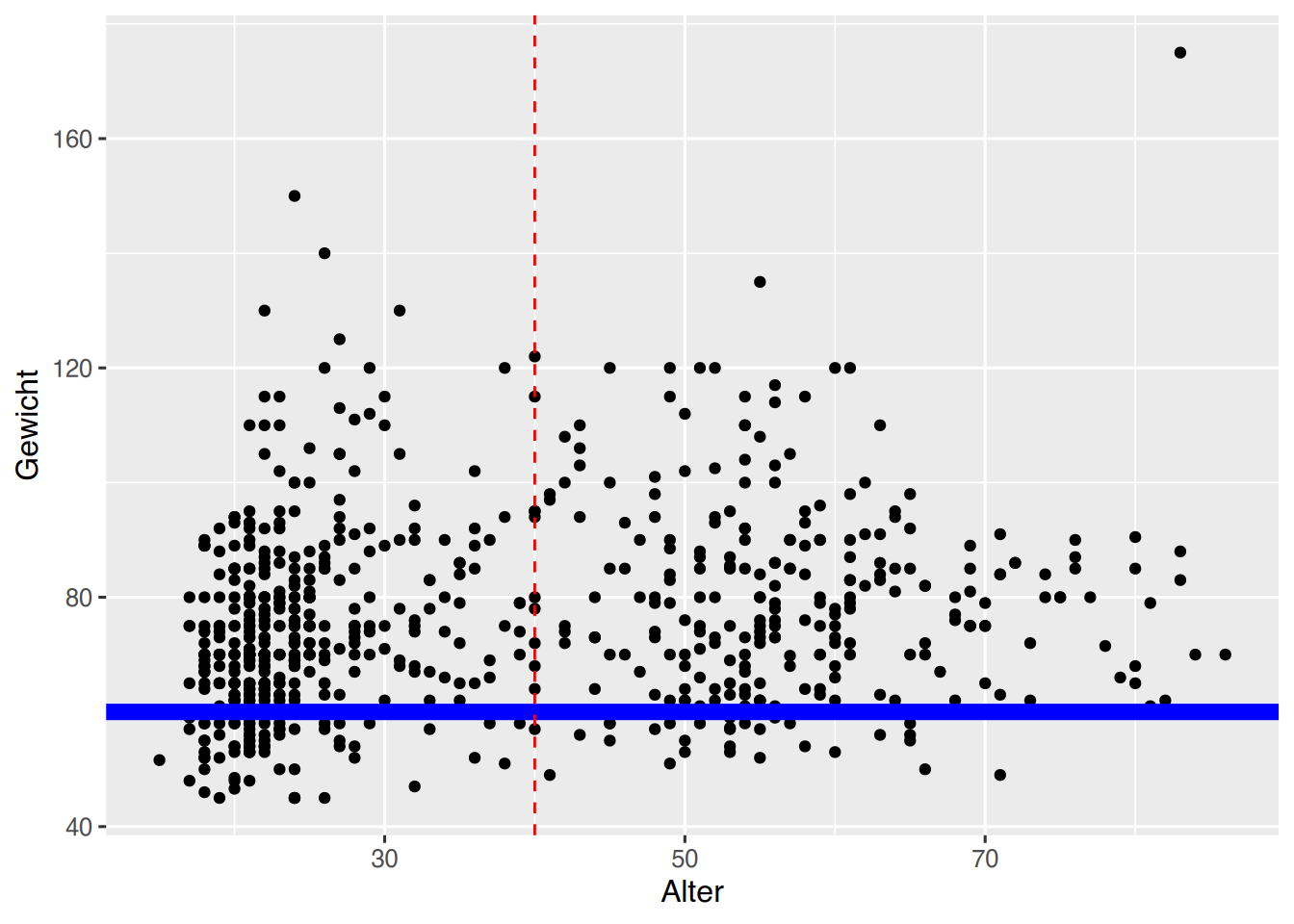
Um zwei beliebige Punkte mit einer Linie zu verbinden, muss zunächst ein Hilfsdatenframe mit den Koordinaten der Punkte erstellt werden. Dieses neue Datenframe wird dann der Funktion geom_line() übergeben.
# Hilfsdatenframe
strich <- data.frame(x = c(30,50),
y = c(10,90))
ggplot(pf8) +
aes(x = Alter,
y = Gewicht) +
geom_point() +
geom_line(data = strich,
aes(x=x, y=y),
color="darkgreen",
size=1)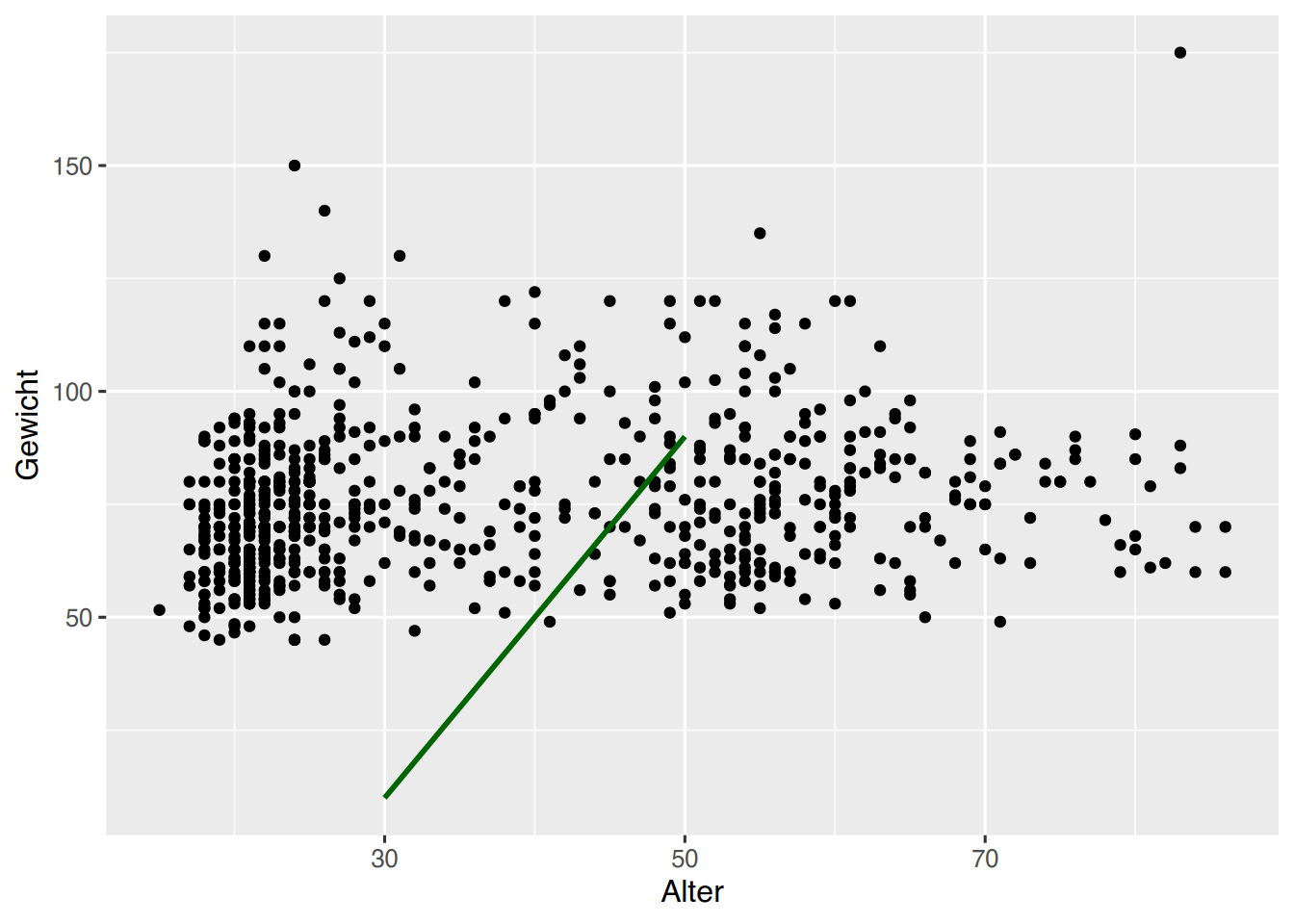
35.12.6 Text
Texte können mit der annotate()-Funktion hinzugfügt werden.
ggplot(pf8) +
aes(x = Alter,
y = Gewicht) +
geom_point() +
annotate(geom="text",
x=30, y=120,
label="Huhu",
color="darkgreen",
size=14)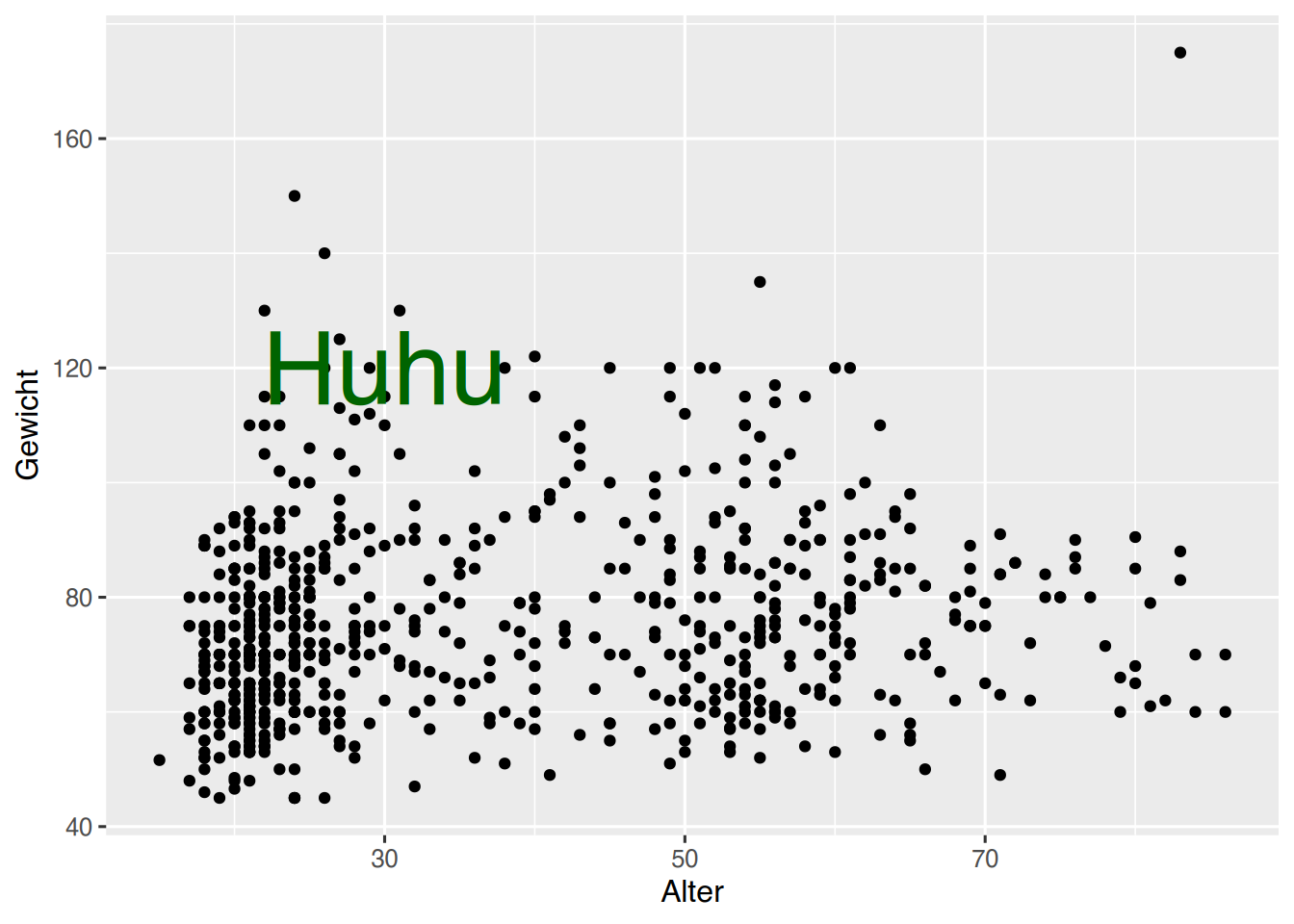
35.12.7 Sonderzeichen
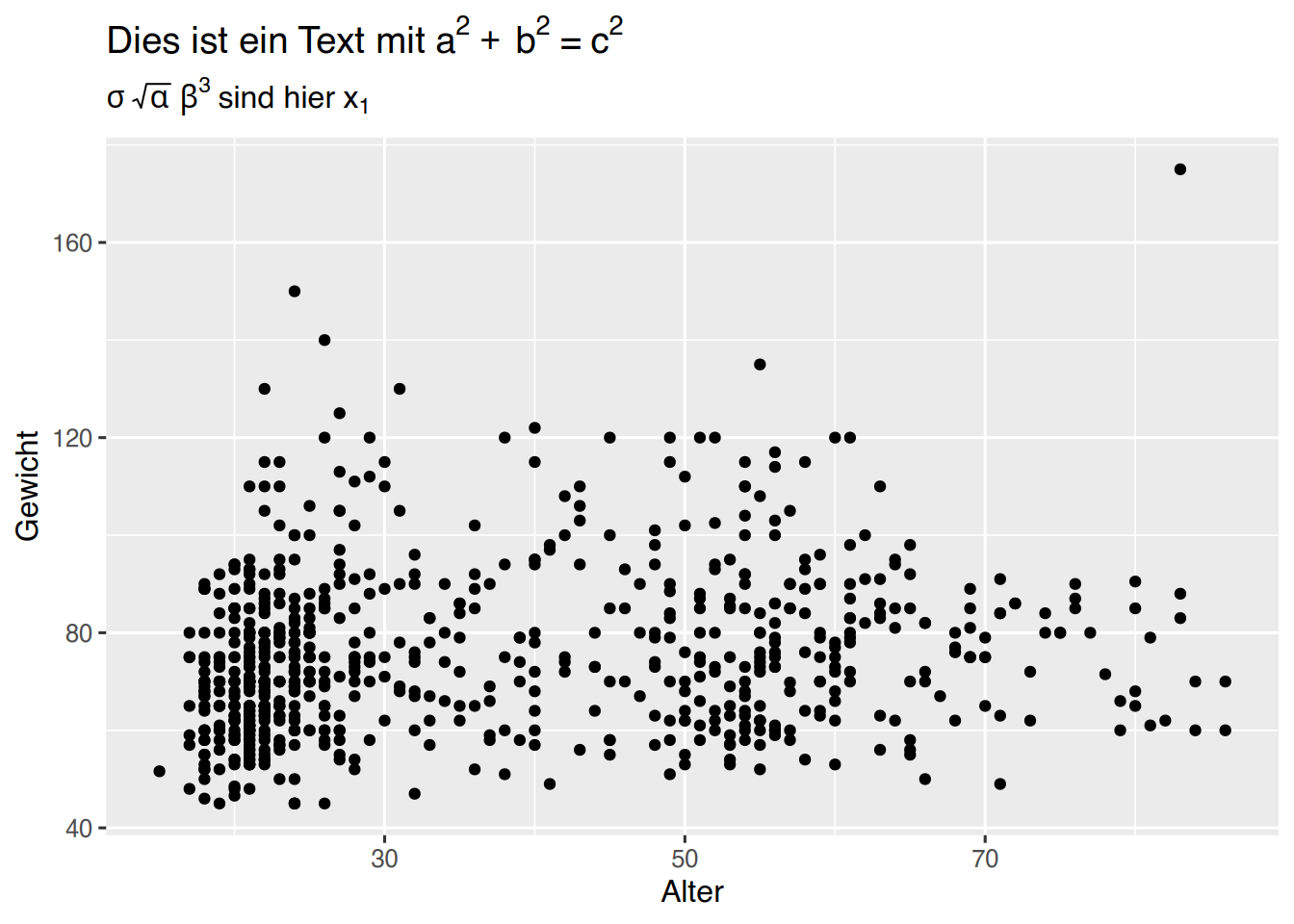
Sonderzeichen und Mathematiksymbole können mittels der expression()-Funktion hinzugefügt werden.
ggplot(pf8) +
aes(x = Alter,
y = Gewicht) +
geom_point() +
labs(title = expression("Dies ist ein Text mit"~a^2~+~b^2~"="~c^2),
subtitle = expression(sigma~sqrt(alpha)~beta^3~"sind hier"~x[1])
)
35.12.8 Themes
In ggplot() sind 8 verschiedene “Themes” implementiert, die das Aussehen der nicht-datenbezogenen Anteile des Plots festlegen. Das Standardtheme ist theme_grey(), die anderen sind:
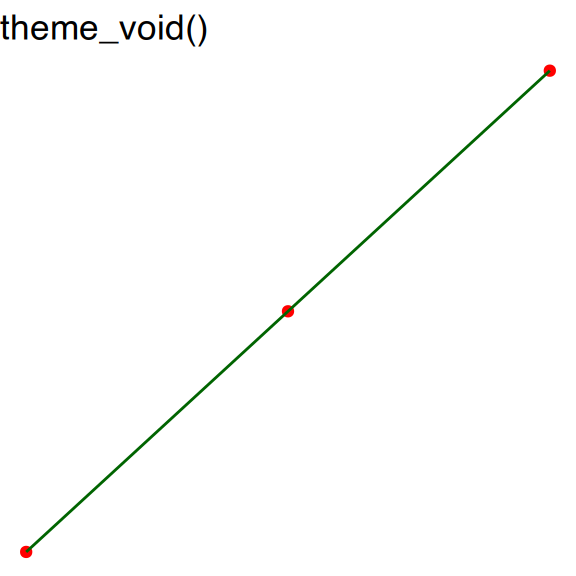
theme_bw(): Eine Variante vontheme_grey()mit weißem Hintergrund und dünnen grauen Gitterlinien.theme_linedraw(): Ein Theme mit nur schwarzen Linien unterschiedlicher Stärke auf weißem Hintergrund, das an eine Zeichnung erinnert.theme_light(): Ähnlich wietheme_linedraw(), aber mit hellgrauen Linien und Achsen, um die Aufmerksamkeit stärker auf die Daten zu lenken.theme_dark(): Das dunkle Pendant vontheme_light(), mit ähnlichen Linienstärken, aber dunklem Hintergrund. Nützlich, um dünne farbige Linien hervorzuheben.theme_minimal(): Ein minimalistisches Theme ohne Hintergrundanmerkungen.theme_classic(): Ein klassisch aussehendes Theme mit x- und y-Achsen-Linien und ohne Gitterlinien.theme_void(): Ein vollständig leeres Theme.
# dummy Daten
df <- data.frame(x = 1:3, y = 1:3)
# Basisplot
base <- ggplot(df, aes(x, y)) +
geom_point(color="red") +
geom_line(color="darkgreen")
# Themepark
base + ggtitle("theme_grey()")
base + theme_grey() + ggtitle("theme_grey()")
base + theme_bw() + ggtitle("theme_bw()")
base + theme_linedraw() + ggtitle("theme_linedraw()")
base + theme_light() + ggtitle("theme_light()")
base + theme_dark() + ggtitle("theme_dark()")
base + theme_minimal() + ggtitle("theme_minimal()")
base + theme_classic() + ggtitle("theme_classic()")
base + theme_void() + ggtitle("theme_void()")