A <- c(48.6, 50.8, 49.4, 47.1, 50.1, 52.5, 49.8, 49, 50.6, 46.7)
B <- c(68, 71.9, 67, 71.5, 70.1, 69.9, 64.5, 68.9, 68, 67.8, 68.3, 68.9)
C <- c(67.5, 61.4, 62.5, 67.4, 64.2, 65.4, 62.5, 63.2, 63.9, 61.2, 64.8, 60.5, 62.3)
df <- data.frame(Therapie = c(rep("A", length(A)),
rep("B", length(B)),
rep("C", length(C))),
Aknereduktion = c(A,B,C)
)55 Lösungen ANOVA
Hier finden Sie die Lösungen zu den Übungsaufgaben von Abschnitt 44.11.
Die hier vorgestellten Lösungen stellen immer nur eine mögliche Vorgehensweisen dar und sind sicherlich nicht der Weisheit letzter Schluss. In R führen viele Wege nach Rom, und wenn Sie mit anderem Code zu den richtigen Ergebnissen kommen, dann ist das völlig in Ordnung.
55.1 Lösung zur Aufgabe 44.11.1
Therapie und Aknereduktion.
# plot()
boxplot(Aknereduktion ~ Therapie, data=df)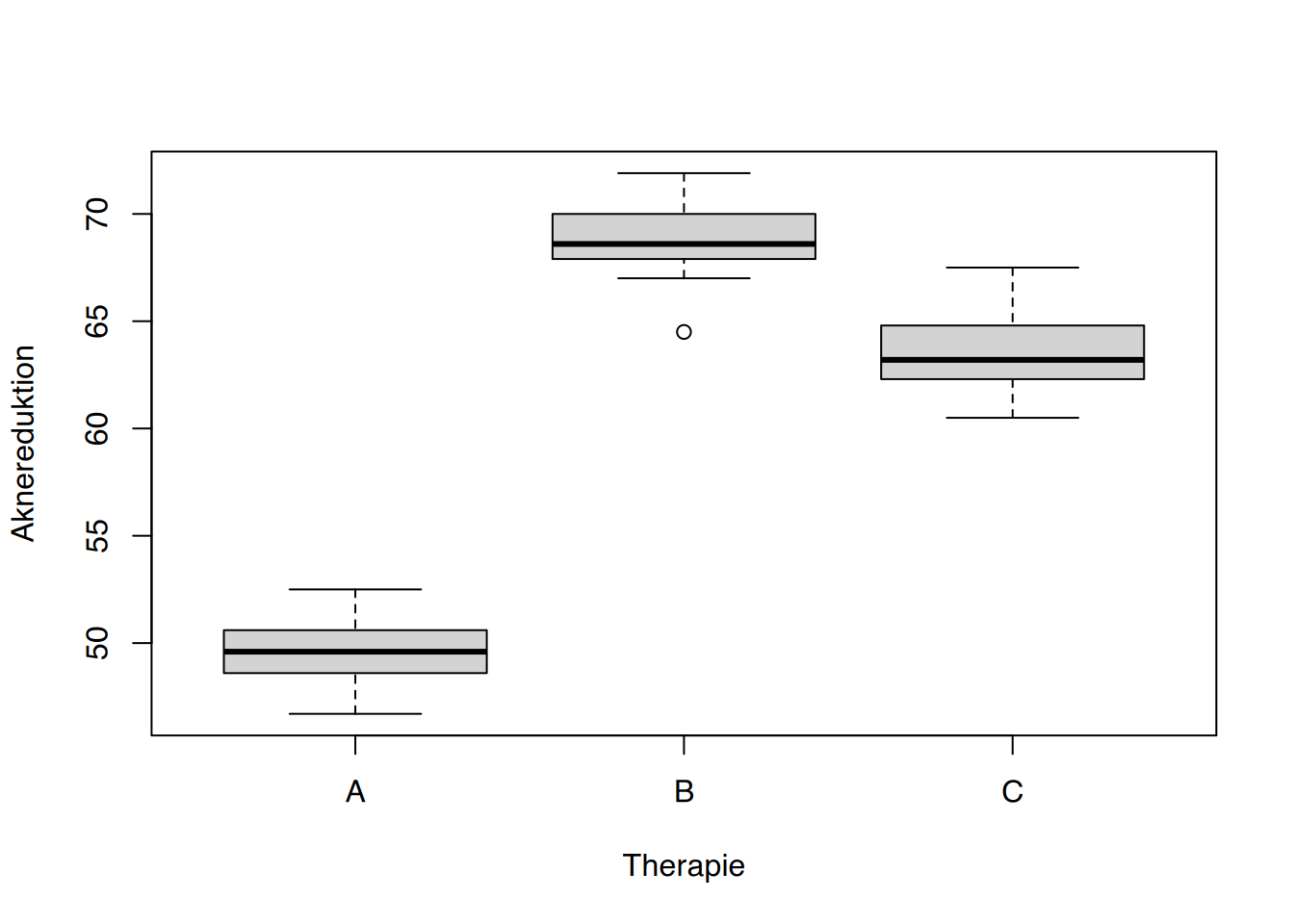
# ggplot()
ggplot(df, aes(x=Therapie, y=Aknereduktion)) +
geom_boxplot(aes(fill=Therapie))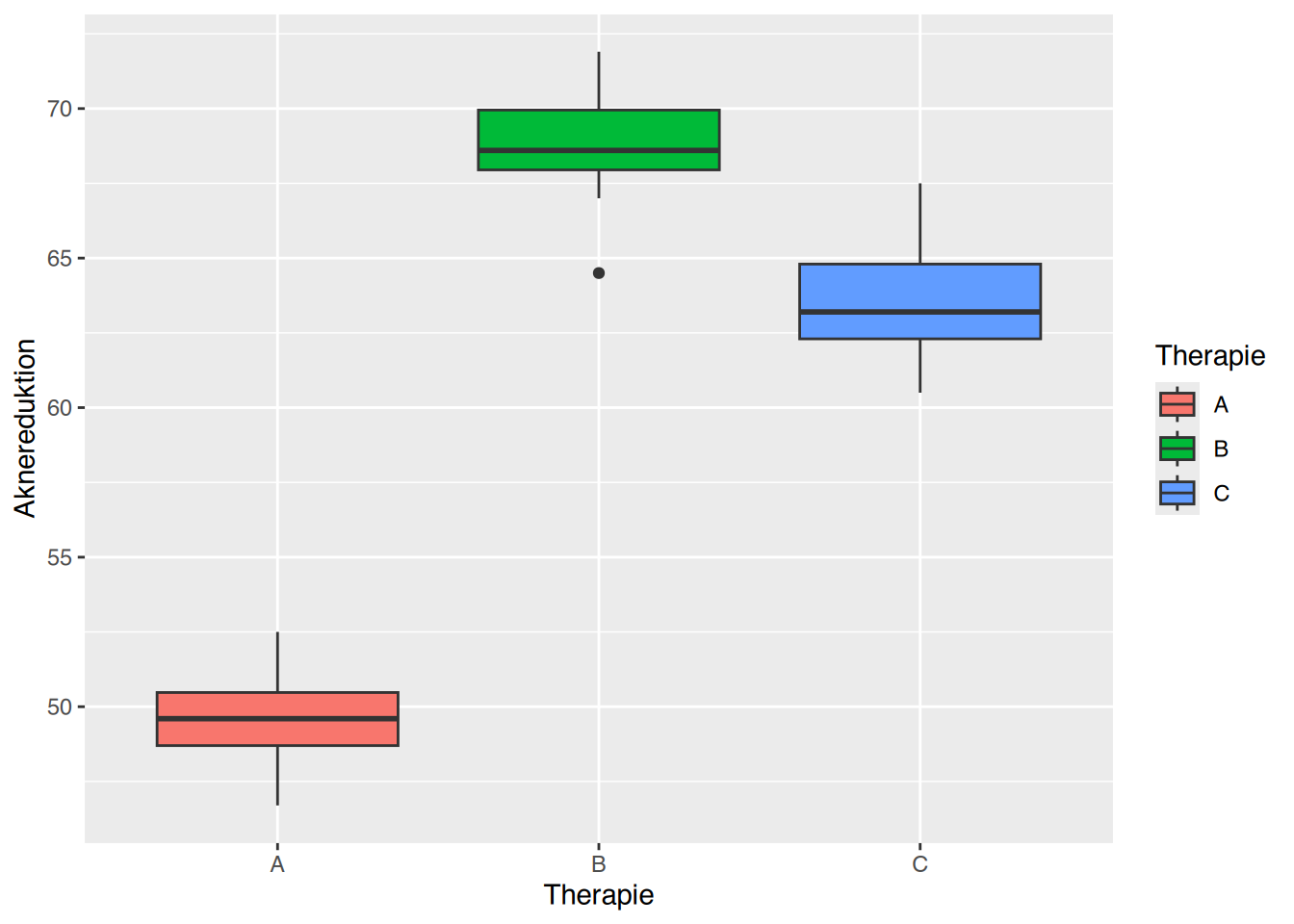
Therapie A unterscheidet sich deutlich von B und C.
fit <- aov(Aknereduktion ~ Therapie, data=df)
summary(fit) Df Sum Sq Mean Sq F value Pr(>F)
Therapie 2 2133.7 1066.8 262 <2e-16 ***
Residuals 32 130.3 4.1
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Es gibt Unterschiede. Schauen wir genauer hin.
pairwise.t.test(df$Aknereduktion, df$Therapie, p.adjust="bonferroni")
Pairwise comparisons using t tests with pooled SD
data: df$Aknereduktion and df$Therapie
A B
B < 2e-16 -
C < 2e-16 1.2e-06
P value adjustment method: bonferroni Alle Gruppen unterscheiden sich jeweils voneinander.
ab <- t.test(A,B)
ac <- t.test(A,C)
cb <- t.test(C,B)
# Konfidenzintervalle
as.numeric(ab$conf.int)[1] -20.93395 -17.61271as.numeric(ac$conf.int)[1] -15.85486 -12.42514as.numeric(cb$conf.int)[1] -6.880637 -3.386029Keines der Intervalle schließt die 0 ein. Alle Therapien unterscheiden sich jeweils voneinander.
# Als Datenframe für ggplot zusammenbauen
plotdf <- data.frame(unten = c(ab$conf.int[1], ac$conf.int[1], cb$conf.int[1]),
oben = c(ab$conf.int[2], ac$conf.int[2], cb$conf.int[2]),
mitte = c(ab$estimate[1]-ab$estimate[2],
ac$estimate[1]-ac$estimate[2],
cb$estimate[1]-cb$estimate[2] ),
Therapie = c("A", "B", "C"))
# ggplot
ggplot(plotdf, aes(x=Therapie, color=Therapie)) +
geom_point(aes(y=mitte)) +
geom_segment(aes(xend=Therapie, y=unten, yend=oben))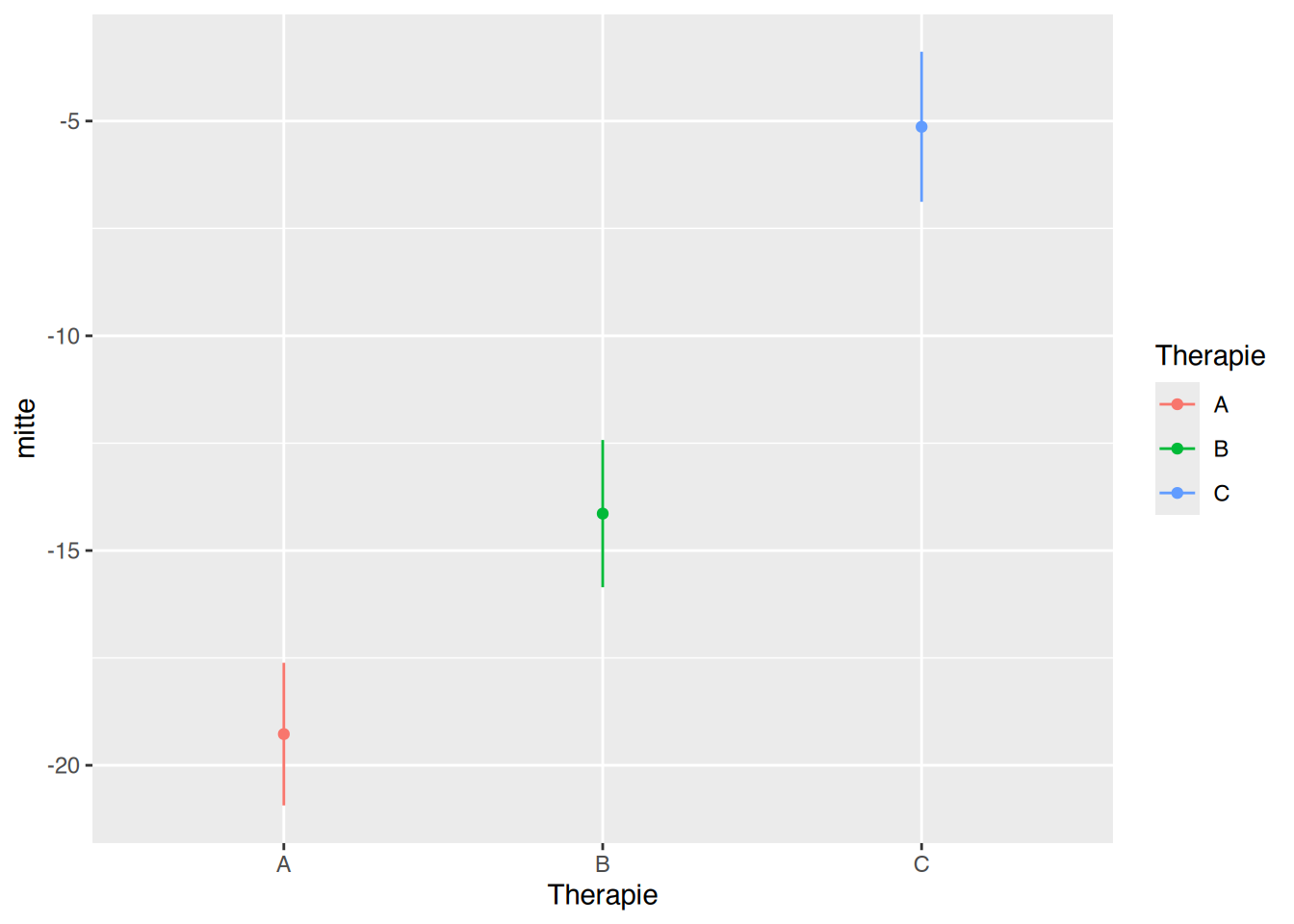
Keines der Intervalle schließt die 0 ein. Alle Therapien unterscheiden sich jeweils voneinander.
55.2 Lösung zur Aufgabe 44.11.2
Schule und Punkte.
# Erstellen des tibbles mit tribble()
df <- tribble(
~A, ~B, ~C, ~D, ~E,
5.5, 6.1, 4.9, 3.2, 6.7,
5.2, 7.2, 5.5, 3.3, 5.8,
5.9, 5.5, 6.1, 5.5, 5.4,
7.1, 6.7, 6.1, 5.7, 5.5,
6.2, 7.6, 6.2, 6.0, 4.9,
5.9, 5.9, 6.4, 6.1, 6.2,
5.3, 8.1, 6.9, 4.7, 6.1,
6.2, 8.3, 4.5, 5.1, 7.0
) %>% # und pivot_longer
pivot_longer(A:E, names_to="Schule", values_to="Punkte")# plot()
boxplot(Punkte ~ Schule, data=df)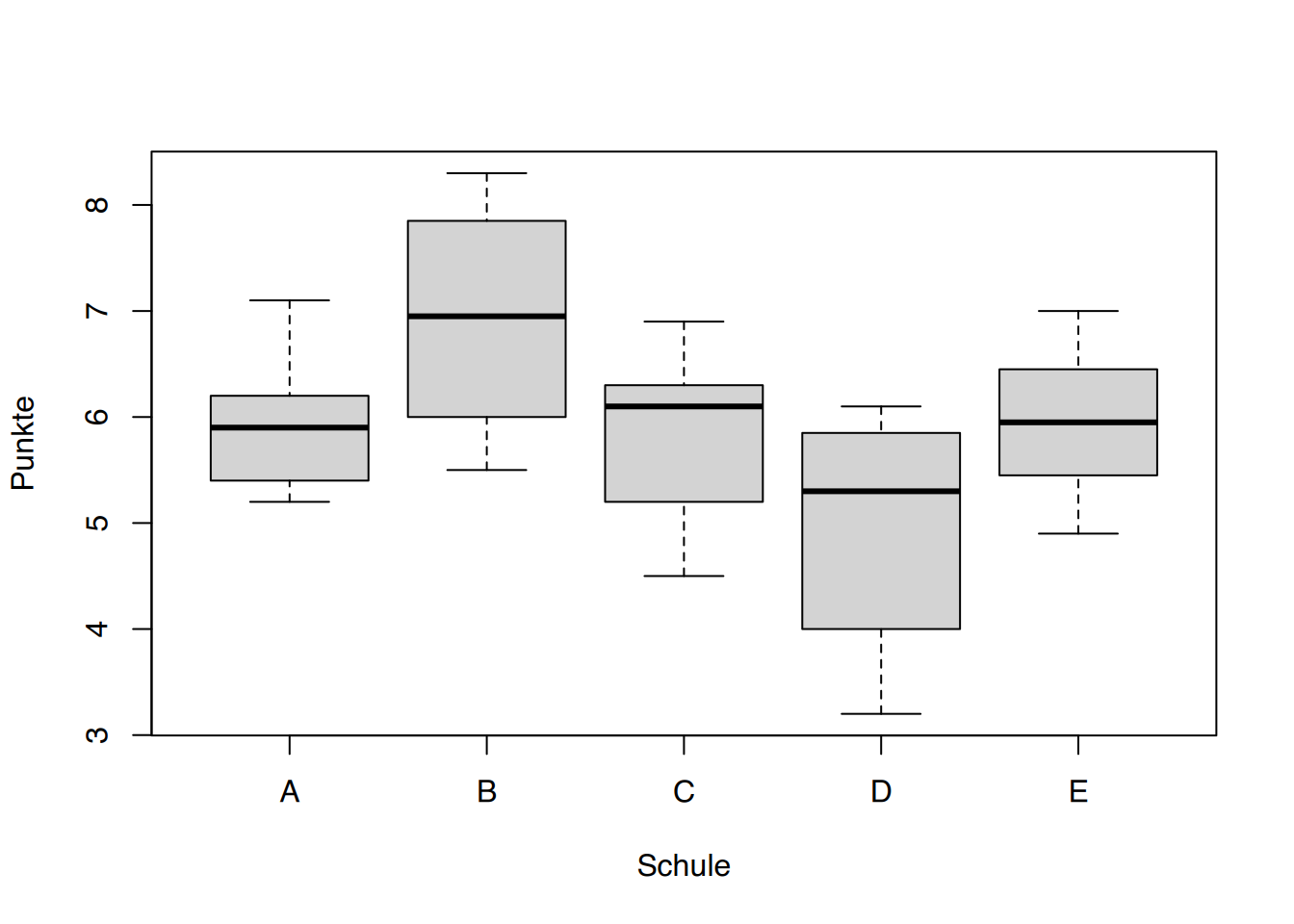
# ggplot()
ggplot(df, aes(x=Schule, fill=Schule, y=Punkte)) +
geom_boxplot() +
# whiskers
stat_boxplot(geom="errorbar")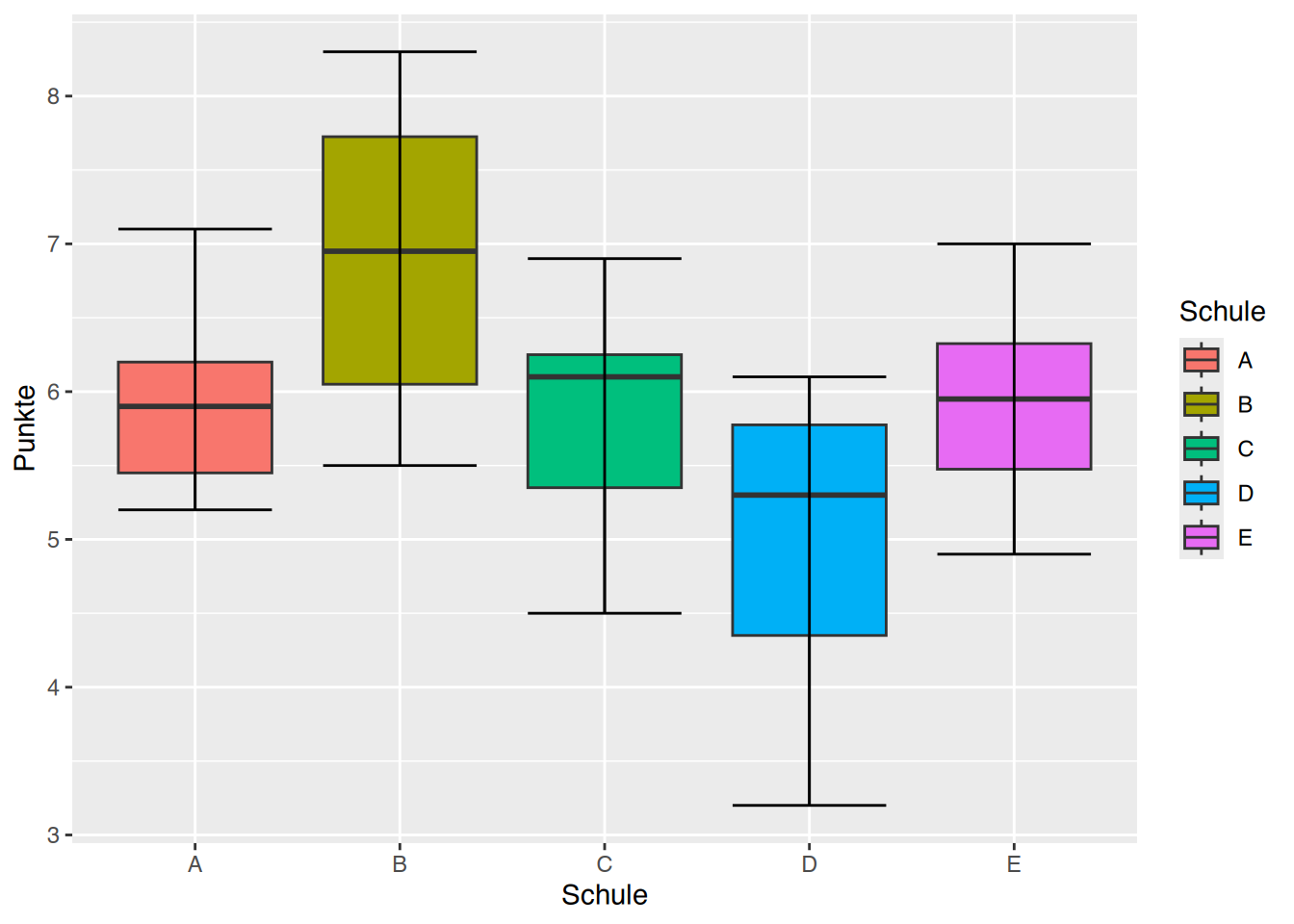
fit <- aov(Punkte ~ Schule, data=df)
summary(fit) Df Sum Sq Mean Sq F value Pr(>F)
Schule 4 15.69 3.921 5.031 0.00261 **
Residuals 35 27.28 0.779
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1pairwise.t.test(df$Punkte, df$Schule, p.adjust="bonferroni")
Pairwise comparisons using t tests with pooled SD
data: df$Punkte and df$Schule
A B C D
B 0.27923 - - -
C 1.00000 0.17589 - -
D 0.36032 0.00078 0.55345 -
E 1.00000 0.33829 1.00000 0.29780
P value adjustment method: bonferroni Es gibt einen Unterschied zwischen Schule B und Schule D. In Schule B sind die Leistungen besser als in Schule D. Die Leistungen in B sind aber nicht “die besten”, da kein Unterschied zu den anderen Schulen gezeigt werden kann. Die Mittelwerte sind in B aber höher.
55.3 Lösung zur Aufgabe 44.11.3
# Erstellen des tibbles mit tribble()
df <- tribble(
~Kontrolle, ~AnginaP, ~Arrhytmia, ~Herzinfarkt,
83, 81, 75, 61,
61, 65, 68, 75,
80, 77, 80, 78,
63, 87, 80, 80,
67, 95, 74, 68,
89, 89, 78, 65,
71, 103, 69, 68,
73, 89, 72, 69,
70, 78, 76, 70,
66, 83, 75, 79,
57, 91, 69, 61
) %>% # und pivot_longer
pivot_longer(1:4, names_to="Gruppe", values_to="Puls")
# ANOVa
fit <- aov(Puls ~ Gruppe, data=df)
summary(fit) Df Sum Sq Mean Sq F value Pr(>F)
Gruppe 3 1587 529.1 8.043 0.00026 ***
Residuals 40 2631 65.8
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1# paarweise
pairwise.t.test(df$Puls, df$Gruppe, p.adjust="bonferroni")
Pairwise comparisons using t tests with pooled SD
data: df$Puls and df$Gruppe
AnginaP Arrhytmia Herzinfarkt
Arrhytmia 0.01584 - -
Herzinfarkt 0.00062 1.00000 -
Kontrolle 0.00100 1.00000 1.00000
P value adjustment method: bonferroni Patienten mit Angina Pectoris unterscheiden sich von allen anderen Patientengruppen.
55.4 Lösung zur Aufgabe 44.11.4
# Erstellen des tibbles mit tribble()
df <- tribble(
~Low, ~Medium, ~High,
36, 43, 45,
33, 38, 39,
35, 41, 33,
39, 34, 39,
41, 28, 33,
41, 44, 26,
44, 30, 39,
45, 31, 29
) %>% # und pivot_longer
pivot_longer(1:3, names_to="Konzentration", values_to="Atemfrequenz")
# ANOVa
fit <- aov(Atemfrequenz ~ Konzentration, data=df)
summary(fit) Df Sum Sq Mean Sq F value Pr(>F)
Konzentration 2 67.6 33.79 1.056 0.366
Residuals 21 672.2 32.01 # paarweise
pairwise.t.test(df$Atemfrequenz, df$Konzentration, p.adjust="bonferroni")
Pairwise comparisons using t tests with pooled SD
data: df$Atemfrequenz and df$Konzentration
High Low
Low 0.56 -
Medium 1.00 0.85
P value adjustment method: bonferroni Es kann kein signifikanter Unterschied festgestellt werden.