# c() fällt auf den "kleinsten gemeinsamen Nenner" zurück
c(1, 2, 3, 4, "fünf", "sechs", TRUE, TRUE, FALSE)## [1] "1" "2" "3" "4" "fünf" "sechs" "TRUE" "TRUE" "FALSE"Die Datentypen können wiederum in Datenklassen gespeichert werden. Für uns wichtige Datenklassen sind:
Vektoren
Matrizen
Faktoren (Gruppen, Rangfolge)
Datenframes
Listen
Vektoren haben wir schon kennengelernt. Ein Vektor ist eine einfache Wertereihe vom selben Datentyp. So erzeugt die Funktion c() einen Datenvektor. Alle Werte der Datenklasse Vektor müssen Werte des selben Wertetyps enthalen. Kombinieren wir numerische, character und logische Werte in einem Vektor, so wandeln sich alle Werte in den kleinsten gemeinsamen Datentyp (nämlich character) um.
# c() fällt auf den "kleinsten gemeinsamen Nenner" zurück
c(1, 2, 3, 4, "fünf", "sechs", TRUE, TRUE, FALSE)## [1] "1" "2" "3" "4" "fünf" "sechs" "TRUE" "TRUE" "FALSE"Alle Werte sind nun vom Datentyp character, erkennbar an den Anführungszeichen.
Auf die Werte kann man zugreifen, indem man den Variablennamen eingibt:
# erzeuge einen Vektor
vektor <- seq(1, 20, 1)
# anzeigen
vektor## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20Auf die einzelnen Werte des Vektors kann man Zugreifen, indem man die gewünschte Position in eckigen Klammern an den Variablennamen anhgängt.
# Zeige den ersten Wert von "vektor"
vektor[1]## [1] 1Auch hier können wir Positionsbereiche mit einem : angeben
# Zeige die Werte an Position 4 bis 15
vektor[4:15]## [1] 4 5 6 7 8 9 10 11 12 13 14 15Mit einem Minuszeichen können auch bestimmte Werte oder Wertbereiche ausgelassen werden.
# Zeige "vektor" OHNE den ersten Wert
vektor[-1]## [1] 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20# Zeige "vektor" OHNE die Werte 10 bis 14
vektor[-(10:14)]## [1] 1 2 3 4 5 6 7 8 9 15 16 17 18 19 20Wieviele Werte ein Vektor enthält erfährt man mit der Funktion lenght().
# wieviele Werte hat "vektor"?
length(vektor)## [1] 20Der Vektor vektor beinhaltet 20 Werte.
Mit der Funktion is.vector() kann geprüft werden, ob ein Objekt ein Vektor ist.
# ist "vektor" ein Vektor?
is.vector(vektor)## [1] TRUEMatrizen sind zweidimensionale Strukturen (Tabellen) und werden von R-intern durch Vektoren dargestellt. Dies impliziert, dass alle Werte der Matrix vom selben Datentyp (z.B. numerisch) sein müssen, genau so wie bei Vektoren.
Um besser zu erklären, wie Matrizen funktionieren, erzeugen wir zunächst ein paar Beispielvektoren.
# Erzeuge Testwertereihen
a <- c(11, 12, 13, 14, 15)
b <- c(21, 22, 23, 24, 25)
c <- c(31, 32, 33, 34, 35)
d <- c(41, 42, 43, 44, 45)
e <- c(51, 52, 53, 54, 55)
f <- c("eins", "zwei", "drei", "vier", "fünf")
# Füge alle Zahlen zu einem Vektor zusammen
alle <- c(a, b, c, d, e)
# anzeigen
alle## [1] 11 12 13 14 15 21 22 23 24 25 31 32 33 34 35 41 42 43 44 45 51 52 53 54 55Die Funktoin martix() setzt aus einem Vektor eine Matrix zusammen. Lässt man alle Parameter im Funktionsaufruf matrix() leer, wird eine Matrix mit 1 Spalte erzeugt.
# Erzeuge eine Matrix aus Vektor "a"
matrix(a)## [,1]
## [1,] 11
## [2,] 12
## [3,] 13
## [4,] 14
## [5,] 15Mit dem Parameter ncol kann die gewünschte Anzahl an Spalten übergeben werden:
# Erzeuge eine Matrix aus Vektoren "a" und "b"
# mit 2 Spalten
matrix(c(a,b), ncol=2)## [,1] [,2]
## [1,] 11 21
## [2,] 12 22
## [3,] 13 23
## [4,] 14 24
## [5,] 15 25Mit dem Parameter nrow kann die gewünschte Anzahl an Zeilen übergeben werden:
# Erzeuge eine Matrix aus Vektoren "a" und "b"
# mit 2 Zeilen
matrix(c(a,b), nrow=2)## [,1] [,2] [,3] [,4] [,5]
## [1,] 11 13 15 22 24
## [2,] 12 14 21 23 25Achten Sie auf die Reihenfolge, in der die Werte in der Matrix angelegt wurden. Das ist wahrscheinlich nicht das Ergebnis, das Sie erwartet haben. Die Funktion matrix() arbeitet standardmäßig die Werte pro Spalte (spaltenorientiert) ab. Wir können das mit dem Parameter byrow ändern:
# Erzeuge eine Matrix aus Vektoren "a" und "b"
# mit 2 Zeilen, diesmal zeilenorientiert
matrix(c(a,b), nrow=2, byrow=TRUE)## [,1] [,2] [,3] [,4] [,5]
## [1,] 11 12 13 14 15
## [2,] 21 22 23 24 25Sobald eine Matrix mit 2 oder mehr Spalten angelegt werden soll, muss der Datenvektor so viele Werte enthalten, dass die gewünschte Matrix vollständig erstellt werden kann. Sollten zu wenige Werte vorhanden sein, gibt R eine Warnmeldung aus. Für eine Matrix mit 2 Spalten muss also eine gerade Anzahl an Werten vorhanden sein.
# Matrix mit 2 Spalten benötigt gerade Anzahl an Werten
# daher gibt R (mit nur 9 Werten) eine Warnmeldung aus
matrix(1:9, nrow=2)## Warning in matrix(1:9, nrow = 2): data length [9] is not a sub-multiple or
## multiple of the number of rows [2]## [,1] [,2] [,3] [,4] [,5]
## [1,] 1 3 5 7 9
## [2,] 2 4 6 8 1Wie Sie sehen versucht R selbstständig die Matrix zu vervollständigen. Hierfür wiederholt R die angegebenen Werte so lange, bis die Matrix “voll” ist. Im obigen Beispiel wurde nach der 9 wieder eine 1 eingetragen.
Wie bereits erwähnt müssen die Werte vom selben Datentyp sein. Mischt man numeric mit character, fällt auch Matrix auf den kleinsten gemeinsamen Datentyp (character) zurück.
# Erzeuge eine Matrix aus Vektoren "a" und "f"
# fällt auch Typ "char" zurück
matrix(c(a,f), nrow=2, byrow=TRUE)## [,1] [,2] [,3] [,4] [,5]
## [1,] "11" "12" "13" "14" "15"
## [2,] "eins" "zwei" "drei" "vier" "fünf"Neben matrix() können auch die Befehle rbind() (für rowbind) und cbind() (für columnbind) verwendet werden, um eine Matrix zu erzeugen.
Der Befehl cbind() fügt die übergebenen Vektoren spaltenorientiert zu einer Matrix zusammen.
# Erzeuge eine Matrix aus Vektoren "c" und "d"
# spaltenorientiert
cbind(c, d)## c d
## [1,] 31 41
## [2,] 32 42
## [3,] 33 43
## [4,] 34 44
## [5,] 35 45Der Befehl rbind() fügt die übergebenen Vektoren zeilenorientiert zu einer Matrix zusammen.
# Erzeuge eine Matrix aus Vektoren "c" und "d"
# zeilenorientiert
rbind(c, d)## [,1] [,2] [,3] [,4] [,5]
## c 31 32 33 34 35
## d 41 42 43 44 45Wir speichern eine Matrix in einer Variable:
# Erzeuge eine Matrix aus Vektor "alle"
# mit 5 Spalten
mymatrix <- matrix(alle, ncol=5)
# ausgeben
mymatrix## [,1] [,2] [,3] [,4] [,5]
## [1,] 11 21 31 41 51
## [2,] 12 22 32 42 52
## [3,] 13 23 33 43 53
## [4,] 14 24 34 44 54
## [5,] 15 25 35 45 55An der Ausgabe der Spalten- und Zeilentitel lässt sich erahnen, wie die einzelnen Werte einer Matrix refenziert werden können. Bei Vektoren können die einzelnen Werte abgerufen werden, indem in eckigen Klammern die gewünschte Position angegeben wird. Dies funktioniert bei Matrizen ähnlich, jedoch muss innerhalb der eckigen Klammer zwischen Zeilen und Spalten unterschieden werden. Dies geschieht mit einem Komma, wobei vor dem Komma die Zeilen, und nach dem Komma die Spalten referenziert werden.
# zeige die 1. Zeile der Matrix
# die Zahl vor dem Komma repräsentiert die Zeilen
mymatrix[1,]## [1] 11 21 31 41 51# zeige die 1. Spalte der Matrix
# die Zahl nach dem Komma repräsentiert die Spalten
mymatrix[,1]## [1] 11 12 13 14 15Wie in einem Koordinatensystem können nun gezielt einzelne Werte oder Wertbereiche referenziert werden.
# zeige den Wert in Zeile 3 und Spalte 2
mymatrix[3,2]## [1] 23# zeige die Werte aus Zeile 2 bis 4
# und Spalte 1 bis 3
mymatrix[2:4,1:3]## [,1] [,2] [,3]
## [1,] 12 22 32
## [2,] 13 23 33
## [3,] 14 24 34Mit der Funktion t() kann die Matrix transponiert werden, das beduetet, es werden Zeilen und Spalten diagonal gespiegelt.
# zeige meine Matrix
mymatrix## [,1] [,2] [,3] [,4] [,5]
## [1,] 11 21 31 41 51
## [2,] 12 22 32 42 52
## [3,] 13 23 33 43 53
## [4,] 14 24 34 44 54
## [5,] 15 25 35 45 55# transponiere meine Matrix
t(mymatrix)## [,1] [,2] [,3] [,4] [,5]
## [1,] 11 12 13 14 15
## [2,] 21 22 23 24 25
## [3,] 31 32 33 34 35
## [4,] 41 42 43 44 45
## [5,] 51 52 53 54 55Mit den Funktionen colnames() und rownames() können Spalten und Zeilen noch betitelt werden.
Wir benennen unsere Spalten von “a” bis “e”:
# benenne Spalten der Matrix
colnames(mymatrix) <- c("a", "b", "c", "d", "e")
# anzeigen
mymatrix## a b c d e
## [1,] 11 21 31 41 51
## [2,] 12 22 32 42 52
## [3,] 13 23 33 43 53
## [4,] 14 24 34 44 54
## [5,] 15 25 35 45 55Wir benennen unsere Zeilen von römisch I bis V:
# benenne Spalten der Matrix
rownames(mymatrix) <- c("I", "II", "III", "IV", "V")
# anzeigen
mymatrix## a b c d e
## I 11 21 31 41 51
## II 12 22 32 42 52
## III 13 23 33 43 53
## IV 14 24 34 44 54
## V 15 25 35 45 55Mit der Funktion class() kann die Datenklasse angezeigt werden.
# welcher Datentyp ist Variable "mymatrix"
class(mymatrix)## [1] "matrix" "array"Mit der Funktion is.matrix() kann zudem geprüft werden, ob ein Objekt eine Matrix ist.
# ist "mymatrix" eine Matrix?
is.matrix(mymatrix)## [1] TRUE# ist "vector" eine Matrix?
is.matrix(vector)## [1] FALSEAls Übung können wir nun beispielsweise die Anzahl der Beschäftigten in Pflegeberufen aus dem “Pflegethermometer 2018” (Isfort et al., 2018) (siehe Abbildung 8.1), als Matrix in R übertragen.
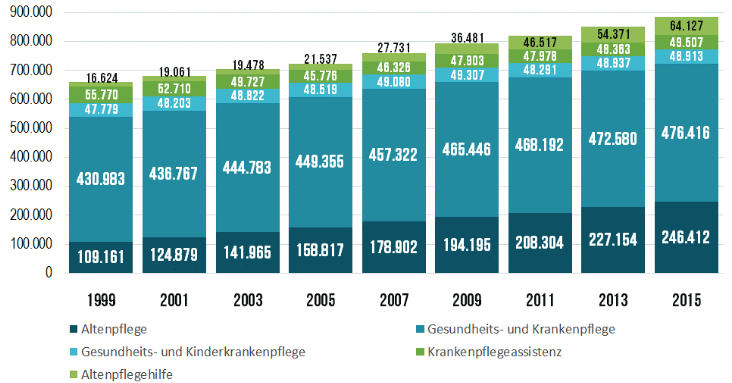
# Schreibe die Zahlen reihenweise aus der Grafik ab
Pflegeberufe <- c(16624, 19061, 19478, 21537, 27731, 36481, 46517, 54371, 64127,
55770,52710, 49727, 45776, 48326, 47903, 47978, 48363, 49507,
47779, 48203, 48822, 48519, 49080, 49307, 48291, 48937, 48913,
430983, 436767, 444783, 449355, 457322, 465446, 468192, 472580, 476416,
109161, 124879, 141965, 158817, 178902, 194195, 208304, 227154, 246412 )
# überführe in Matrix mit 9 Spalten
# Die Werte kommen reihenweise
Pflegeberufe <- matrix(Pflegeberufe, byrow=T,ncol=9)
# benenne die Spalten
colnames(Pflegeberufe) <- c(1999, 2001, 2003, 2005, 2007, 2009, 2011, 2013, 2015)
# benenne die Reihen
rownames(Pflegeberufe) <- c("Krankenpflegeassistenz", "Altenpflegehilfe", "Kinderkrankenpflege", "Krankenpflege", "Altenpflege")
# zeige Tabelle
Pflegeberufe## 1999 2001 2003 2005 2007 2009 2011 2013
## Krankenpflegeassistenz 16624 19061 19478 21537 27731 36481 46517 54371
## Altenpflegehilfe 55770 52710 49727 45776 48326 47903 47978 48363
## Kinderkrankenpflege 47779 48203 48822 48519 49080 49307 48291 48937
## Krankenpflege 430983 436767 444783 449355 457322 465446 468192 472580
## Altenpflege 109161 124879 141965 158817 178902 194195 208304 227154
## 2015
## Krankenpflegeassistenz 64127
## Altenpflegehilfe 49507
## Kinderkrankenpflege 48913
## Krankenpflege 476416
## Altenpflege 246412Wir werden auf diese Matrix später noch zurückkommen.
Die Datenklasse factor beschreibt gruppierte (nominale) oder ranggeordnete (ordinale) Werte. Gruppierte Werte sind beispielsweise “Beruf”, “Konfession”, “Familienstand” oder “Geschlecht”. Es liegt keine Reihenfolge unter den Gruppen vor. Sie werden mit der Funktion factor() erstellt.
# nominalen Faktoren erstellen
v_fac1 <- factor(c("maennlich", "weiblich", "divers"))
# ausgeben
v_fac1## [1] maennlich weiblich divers
## Levels: divers maennlich weiblichUnsere Variable v_fac1 besteht aus 3 Gruppen, nämlich divers, maennlich und weiblich mit je einem Wert. Erzeugen wir die Daten erneut, diesmal mit mehr Werten und einer abgekürzten Schreibweise für das Geschlecht.
# Faktoren neu erstellen, mit mehr Werten
v_fac1 <- factor(c("m", "w", "d","m", "w", "d","m", "w", "d","m", "w", "d"))
# ausgeben
v_fac1## [1] m w d m w d m w d m w d
## Levels: d m wIn der ersten Zeile sehen wir unsere Datenreihe, in der zweiten Zeile alle Gruppen (Levels), in diesem Falle d, m und w.
Für ordninale (also ranggeordnete) Werte nehmen wir ebenfalls die factor()-Funktion. Versuchen wir also, klassiche Schulnoten abzubilden. Wir stellen uns eine virtuelle Liste mit Schulnoten von 100 SchülerInnen vor. Wir übertragen die Noten von der virtuellen Liste in R, und zwar in der Reihenfolge, wie sie auf unserer virtuellen Notenliste stehen könnten (sprich: unsortiert).
# Faktoren aus Notenliste erstellen
v_fac2 <- factor(c("gut", "ausreichend", "sehr gut", "ausreichend", "befriedigend", "magelhaft", "ungenügend", "gut", "gut", "sehr gut"))
# ausgeben
v_fac2## [1] gut ausreichend sehr gut ausreichend befriedigend
## [6] magelhaft ungenügend gut gut sehr gut
## Levels: ausreichend befriedigend gut magelhaft sehr gut ungenügendIn der ersten Zeile sehen wir unsere Datenreihe, in der letzten Zeile alle Ränge (Levels). Ein Blick auf die Levels zeigt aber auch, dass diese in einer falschen Reihehnfolge angelegt wurden. Der erste Rang ist hier ausreichend, und der zweite befriedigend (es sollte ja eigentlich so sein, dass sehr gut der erste Rang ist, und gut der zweite, usw). Das liegt daran, dass wir die Noten unsortiert aneinandergereiht haben.
R erstellt die Reihenfolge der Levels anhand der Reihenfolge, in der sie eintreffen.
Dies ist ein häufiger Anfängerfehler bei der Erstellung von Faktoren!
Um unsere Schulnoten in der richtigen Levelreihenfolge anzulegen, müssen wir dem Befehl factor() eben diese Reihenfolge über die Option levels mitgeben (mehr Informationen erhalten Sie über die Hilfeseite ?factor). Konkret übergeben wir per levels=c() die Levelnamen von sehr gut bis ungenügend.
# Faktoren aus Notenliste erstellen # diesmal Levelreihenfolge mit "levels=" vorgeben
v_fac2 <- factor(c("gut", "ausreichend", "sehr gut", "ausreichend", "befriedigend", "mangelhaft", "ungenügend", "gut", "gut", "sehr gut"),
levels=c("sehr gut", "gut", "befriedigend", "ausreichend","mangelhaft", "ungenügend"))
# ausgeben
v_fac2## [1] gut ausreichend sehr gut ausreichend befriedigend
## [6] mangelhaft ungenügend gut gut sehr gut
## Levels: sehr gut gut befriedigend ausreichend mangelhaft ungenügendDie Levels sind nun in der korrekten Reihenfolge.
Wir hätten die Daten aber gar nicht neu eingeben müssen. Der Befehl lässt sich verkürzen, indem man einfach die bestehende Variable als Input nutzt und neu überschreibt:
# Levelreihenfolge in "v_fac2" reparieren und überschreiben
v_fac2 <- factor(v_fac2, levels=c("sehr gut", "gut", "befriedigend", "ausreichend","mangelhaft", "ungenügend"))
# ausgeben
v_fac2## [1] gut ausreichend sehr gut ausreichend befriedigend
## [6] mangelhaft ungenügend gut gut sehr gut
## Levels: sehr gut gut befriedigend ausreichend mangelhaft ungenügendSo kann man auch nachträglich die Levelreihenfolge korrigieren.
Mit dem Befehl revalue() aus dem plyr-Zusatzpaket lassen sich die Werte und Levels von Faktoren umändern. Wir ändern unsere Schulnoten von den ausgeschriebenen Noten hin zu Zahlenwerten. Hierfür erzeugen wir eine neue Variable v_fac3.
# Lade Zusatzpaket "plyr"
library(plyr)
# Ändere Levelnamen
v_fac3 <- revalue(v_fac2, c("sehr gut"="1", "gut"="2","befriedigend"="3","ausreichend"="4","mangelhaft"="5","ungenügend"="6"))
# Werte ausgeben
v_fac3## [1] 2 4 1 4 3 5 6 2 2 1
## Levels: 1 2 3 4 5 6Beachten Sie, dass die “Zahlen”werte nur nominaler Natur sind. Wir können mit ihnen nicht rechnen!
# rechnen ist mit factor nicht möglich!
v_fac3 * 100## Warning in Ops.factor(v_fac3, 100): '*' ist nicht sinnvoll für Faktoren## [1] NA NA NA NA NA NA NA NA NA NADie Werte innerhalb einer Faktorenreihe refenziert man so wie bei Vektoren, indem man die gewünschte Position in eckigen Klammern an den Variablennamen anhgängt.
# zeige die Werte von 3 bis 7 von "v_fac3"
v_fac3[3:7]## [1] 1 4 3 5 6
## Levels: 1 2 3 4 5 6Welche Levels in einem Faktor existieren erfährt man mit der Funktion levels().
# welche Levels hat "v_fac3"?
levels(v_fac3)## [1] "1" "2" "3" "4" "5" "6"Durch Kombimation mit der Funktion length() können wir die Anzahl der Levels erfahren.
# wieviele Levels hat "v_fac3"?
length(levels(v_fac3))## [1] 6Die Variable hat 6 Levels.
Welche Level welche Häufigkeit hat erfahren wir mit der Funktion table().
# welche Level hat welche Häufigkeit?
table(v_fac2)## v_fac2
## sehr gut gut befriedigend ausreichend mangelhaft ungenügend
## 2 3 1 2 1 1Mit der Funktion class() kann die Datenklasse angezeigt werden.
# welcher Datentyp ist Variable "v_fac2"
class(v_fac2)## [1] "factor"Mit der Funktion is.factor() kann zudem geprüft werden, ob ein Objekt ein Faktor ist.
# ist "v_fac2" ein Faktor?
is.factor(v_fac2)## [1] TRUE# ist "mymatrix" ein Faktor?
is.factor(mymatrix)## [1] FALSEDie bislang von uns erzeugten Faktoren haben zwar “scheinbar” eine korrekte Levelreihenfolge. Für R handelt es sich dabei aber weiterhin um “einfache” Faktoren, also nominale Daten. Um die Faktoren in ordinale Faktoren umzuwandeln kann die Funktion ordered() verwendet werden. Nutzen wir hierfür unsere Variable v_fac2 mit den Schulnoten.
# wandle in ordinalen Faktor um
ordered(v_fac2)## [1] gut ausreichend sehr gut ausreichend befriedigend
## [6] mangelhaft ungenügend gut gut sehr gut
## 6 Levels: sehr gut < gut < befriedigend < ausreichend < ... < ungenügendDie Levels haben nun eine Rangfolge, welche durch das Kleinerzeichen < dargestellt wird. Leider sind die Levelränge genau verkehrt herum, denn im obigen Falle ist “sehr gut” die kleinste und “ungenügend” die größte Note. Wir ändern also nochmal die Levelreihenfolge. Hierzu übergeben wir die Levelnamen in einen Vektor, und kehren mit der Funktion rev() dessen Reihenfolge um. (die Funktion fct_rev() aus dem forcats-Paket dreht die Levelreihenfolge ebenfalls um, aber derzeit wissen wir ja noch nicht, wie man Zusatzpakete installiert (siehe Kapitel 16)).
# zeige Levelnamen an
levels(v_fac2)## [1] "sehr gut" "gut" "befriedigend" "ausreichend" "mangelhaft"
## [6] "ungenügend"# kehre den Vektor um
rev(levels(v_fac2))## [1] "ungenügend" "mangelhaft" "ausreichend" "befriedigend" "gut"
## [6] "sehr gut"In Kombination mit ordered() erhalten wir so die korrekte ordinale Darstellung.
# überführe die ordinale Reihenfolge in Variable 'v_ord'
v_ord <- ordered(factor(v_fac2,
levels= rev(levels(v_fac2))))
v_ord## [1] gut ausreichend sehr gut ausreichend befriedigend
## [6] mangelhaft ungenügend gut gut sehr gut
## 6 Levels: ungenügend < mangelhaft < ausreichend < befriedigend < ... < sehr gutDie Funktion factor() nimmt zudem den Parameter ordered=TRUE entgegen, der direkt ein ordinales Objekt erzeugt:
# erzeuge direkt einen ordinalen factor
# mit Parameter 'ordered=TRUE'
v_ord <- factor(v_fac2,
levels=rev(levels(v_fac2)),
ordered=TRUE)
v_ord## [1] gut ausreichend sehr gut ausreichend befriedigend
## [6] mangelhaft ungenügend gut gut sehr gut
## 6 Levels: ungenügend < mangelhaft < ausreichend < befriedigend < ... < sehr gutDie Datenklasse Datenframe (Datensatz) ist wohl die wichtigste in R. Datenframes sind ebenso wie Matrizen zweidimensional. Im Unterschied zu einer Matrix können in einem Datenframe unterschiedliche Datentypen, also z.B. numeric, character und factor, zusammengeführt werden. Das Datenframe folgt dabei der Logik “ein Fall pro Zeile” (so genanntes tidy data Format, siehe Kapitel 24). Das bedeutet, dass jede Beobachtung (auch Wiederholungen) in einer eigenen Zeile stehen und die jeweiligen Variablen durch die Spalten repräsentiert werden.
Erzeugen wir uns ein paar Beispielvektoren unterschiedlichen Typs mit je 12 Werten.
# erzeuge Testvektoren "factor", "char", "numeric", "logical"
geschlecht <- factor(rep(c("m", "w", "d"), 4))
spitzname <- c("Hasi", "Ide", "Momsi", "Ryu", "Dave", "Zoid", "Adu", "Efi", "Ole", "Ray", "Sam", "Emi")
hausnummer <- 1:12
angemeldet <- c(TRUE, TRUE, FALSE, T, F, F, F, T, T, T, F, T)Aus den Variablen setzen wir nun mit der Funktion data.frame() ein Datenframe zusammen.
# erzeuge ein Datenframe aus den Testvektoren
data.frame(geschlecht, spitzname, hausnummer, angemeldet)## geschlecht spitzname hausnummer angemeldet
## 1 m Hasi 1 TRUE
## 2 w Ide 2 TRUE
## 3 d Momsi 3 FALSE
## 4 m Ryu 4 TRUE
## 5 w Dave 5 FALSE
## 6 d Zoid 6 FALSE
## 7 m Adu 7 FALSE
## 8 w Efi 8 TRUE
## 9 d Ole 9 TRUE
## 10 m Ray 10 TRUE
## 11 w Sam 11 FALSE
## 12 d Emi 12 TRUEAlternativ können die Werte auch direkt dem Datenframe übergeben werden.
## Wir schreiben die Werte direkt ins Datenframe
## Das Ergebnis ist das selbe
data.frame(geschlecht = factor(rep(c("m", "w", "d"), 4)),
spitzname = c("Hasi", "Ide", "Momsi", "Ryu", "Dave", "Zoid", "Adu", "Efi", "Ole", "Ray", "Sam", "Emi"),
hausnummer = 1:12,
angemeldet = c(TRUE, TRUE, FALSE, T, F, F, F, T, T, T, F, T)
)## geschlecht spitzname hausnummer angemeldet
## 1 m Hasi 1 TRUE
## 2 w Ide 2 TRUE
## 3 d Momsi 3 FALSE
## 4 m Ryu 4 TRUE
## 5 w Dave 5 FALSE
## 6 d Zoid 6 FALSE
## 7 m Adu 7 FALSE
## 8 w Efi 8 TRUE
## 9 d Ole 9 TRUE
## 10 m Ray 10 TRUE
## 11 w Sam 11 FALSE
## 12 d Emi 12 TRUEDas Datenframe speichern wir in die Variable MeinDatenframe.
# speicher Datenframe in Variable
MeinDatenframe <- data.frame(geschlecht, spitzname, hausnummer, angemeldet)
# zeige Datenklasse an
class(MeinDatenframe)## [1] "data.frame"Die Funktion class() weist unsere Variable als Datenframe aus.
Ähnlich wie bei Matrizen müssen die Vektoren jeweils die selbe Anzahl an Werten (die selbe Länge) besitzen, damit das Datenframe vollständig aufgebaut werden kann. Entfernen wir z.B. einen Wert aus der Reihe hausnummer, schlägt der Befehl fehl.
# Datenframe, "hausnummer" ist einen Wert kürzer
data.frame(geschlecht, spitzname, hausnummer[-1], angemeldet)Fehler in data.frame(geschlecht, spitzname, hausnummer[-1], angemeldet) :
Argumente implizieren unterschiedliche Anzahl Zeilen: 12, 11Wenn zwei Datenframes mit den selben Spaltennamen exisiteren, können diese per rbind() zusammengefasst werden. In unserem Beispiel verdoppeln wir einfach unser Datenframe.
# füge 2 Datenframes mittels rbind() zusammen
rbind(MeinDatenframe, MeinDatenframe)## geschlecht spitzname hausnummer angemeldet
## 1 m Hasi 1 TRUE
## 2 w Ide 2 TRUE
## 3 d Momsi 3 FALSE
## 4 m Ryu 4 TRUE
## 5 w Dave 5 FALSE
## 6 d Zoid 6 FALSE
## 7 m Adu 7 FALSE
## 8 w Efi 8 TRUE
## 9 d Ole 9 TRUE
## 10 m Ray 10 TRUE
## 11 w Sam 11 FALSE
## 12 d Emi 12 TRUE
## 13 m Hasi 1 TRUE
## 14 w Ide 2 TRUE
## 15 d Momsi 3 FALSE
## 16 m Ryu 4 TRUE
## 17 w Dave 5 FALSE
## 18 d Zoid 6 FALSE
## 19 m Adu 7 FALSE
## 20 w Efi 8 TRUE
## 21 d Ole 9 TRUE
## 22 m Ray 10 TRUE
## 23 w Sam 11 FALSE
## 24 d Emi 12 TRUEEs funktioniert nicht, wenn eine neue Zeile mit einem Datenvektor übergeben wird, denn in einem Vektor können nur Werte des selben Datentyps vorkommen.
# füge einzelne Zeile mit rbind() hinzu
rbind(MeinDatenframe, c("m", "Joe", 99, TRUE))## geschlecht spitzname hausnummer angemeldet
## 1 m Hasi 1 TRUE
## 2 w Ide 2 TRUE
## 3 d Momsi 3 FALSE
## 4 m Ryu 4 TRUE
## 5 w Dave 5 FALSE
## 6 d Zoid 6 FALSE
## 7 m Adu 7 FALSE
## 8 w Efi 8 TRUE
## 9 d Ole 9 TRUE
## 10 m Ray 10 TRUE
## 11 w Sam 11 FALSE
## 12 d Emi 12 TRUE
## 13 m Joe 99 TRUEZwar sieht es so aus, als sei die neue Zeile korrekt eingetragen worden, wenn wir jedoch das neue Datenframe in einer Variable abspeichern und die Datenklassen überprüfen, stellen wir fest, was falsch gelaufen ist.
# füge einzelne Zeile mit rbind() hinzu
new <- rbind(MeinDatenframe, c("m", "Joe", 99, TRUE))
# überprüfe Datentyp für Spalte "hausnummer"
class(new$hausnummer)## [1] "character"Der Datentyp in Spalte hausnummer ist auf den “kleinsten gemeinsamen Nenner” (character) zurückgefallen. Das liegt daran, dass zunächst der Vektor in c() auf character zurückfällt. Somit sind alle Werte in der c()-Funktion vom Typ character. Bei hausnummer zieht nun dieser neue Wert die gesamte Spalte auf den Typ character zurück. Ebenso verhält es sich bei Variable angemeldet, die eigentlich mal vom Typ logical war.
# überprüfe Datentyp für Spalte "geschlecht"
class(new$angemeldet)## [1] "character"Wenn wir mit der Spalte hausnummer rechnen wollen, schlägt dies fehl.
# multipliziere Spalte "hausnummer" mit 2
new$hausnummer * 2Fehler in new$hausnummer * 2 : nicht-numerisches Argument für binären Operator
rbind()-Befehl kann Ihnen also das gesamte Datenframe “zerschießen”.
Dies ist ein häufiger Anfängerfehler, seien Sie sorgsam, wenn Sie einem Datenframe neue Zeilen hinzufügen!
Um also eine neue Zeile korrekt dem Datenframe hinzuzufügen, muss diese neue Zeile ebenfalls als Datenframe in der selben Struktur (also mit den selben Variablen (Spalten) vorliegen. Die
# neue Zeile
neuezeile <- data.frame( factor("m"), "Joe", 99, TRUE)
# übergebe die Spaltennamen an die neue Zeile
colnames(neuezeile) <- colnames(MeinDatenframe)
# füge zu Datenframe hinzu
new <- rbind(MeinDatenframe, neuezeile)
# anzeigen
new## geschlecht spitzname hausnummer angemeldet
## 1 m Hasi 1 TRUE
## 2 w Ide 2 TRUE
## 3 d Momsi 3 FALSE
## 4 m Ryu 4 TRUE
## 5 w Dave 5 FALSE
## 6 d Zoid 6 FALSE
## 7 m Adu 7 FALSE
## 8 w Efi 8 TRUE
## 9 d Ole 9 TRUE
## 10 m Ray 10 TRUE
## 11 w Sam 11 FALSE
## 12 d Emi 12 TRUE
## 13 m Joe 99 TRUEMit dem Befehl cbind() können dem Datenframe neue Spalten hinzugefügt werden. Hierbei ist wichtig, dass die neue Spalte die selbe Anzahl an Werten aufweist wie die anderen Spalten des Datenframes. Wir erzeugen eine neue Variable und fügen diese als neue Spalte dem Datenframe hinzu.
# neue Variable mit 12 Werten
kinder <- c(1, 4, 3, 1, 2, 3, 2, 1, 4, 2, 3, 4)
# neues Datenframe mit dieser Spalte
new <- cbind(MeinDatenframe, kinder)
# anzeigen
new## geschlecht spitzname hausnummer angemeldet kinder
## 1 m Hasi 1 TRUE 1
## 2 w Ide 2 TRUE 4
## 3 d Momsi 3 FALSE 3
## 4 m Ryu 4 TRUE 1
## 5 w Dave 5 FALSE 2
## 6 d Zoid 6 FALSE 3
## 7 m Adu 7 FALSE 2
## 8 w Efi 8 TRUE 1
## 9 d Ole 9 TRUE 4
## 10 m Ray 10 TRUE 2
## 11 w Sam 11 FALSE 3
## 12 d Emi 12 TRUE 4Wir könnten aber auch einfach schreiben:
MeinDatenframe$kinder <- kinderWenn die Variablen (Spalten) von zwei Datensätzen zusammengefügt werden sollen, kann alternativ auch die Funktion merge() verwendet werden. Die Funktion schaut in beiden Datenframes nach einer ID-Variable, anhand derer sie die Daten einander zuordnen kann. In unserem Beispiel kann das die Variable spitzname sein. Ein weiteres Test-Datenframe könnte so aussehen:
## Wir schreiben die Werte direkt ins Datenframe
## Das Ergebnis ist das selbe
test <- data.frame(kinder = c(1, 4, 3, 1, 2, 3, 2, 1, 4, 2, 3, 4),
spitzname = c( "Emi", "Dave", "Sam", "Hasi", "Zoid", "Ray", "Ide", "Momsi", "Ryu", "Adu", "Efi", "Ole")
)
# anzeigen
test## kinder spitzname
## 1 1 Emi
## 2 4 Dave
## 3 3 Sam
## 4 1 Hasi
## 5 2 Zoid
## 6 3 Ray
## 7 2 Ide
## 8 1 Momsi
## 9 4 Ryu
## 10 2 Adu
## 11 3 Efi
## 12 4 OleWie Sie sehen können, ist die Reihenfolge der Spitznamen eine andere als in MeinDatenframe. Mittels merge() können die beiden Datenframes nun dennoch zusammengefügt werden. Über die Parameter by.x und by.y legen wir fest, anhand welcher Spalten das Zusammenführen ausgerichtet werden soll. In beiden Datensätzen ist dies die Variable spitzname. Daher lautet der Funktionsaufruf:
## Vereine MeinDatenframe und test
## anhand der Spalte spitzname
merge(MeinDatenframe, test,
by.x = "spitzname", by.y = "spitzname")## spitzname geschlecht hausnummer angemeldet kinder
## 1 Adu m 7 FALSE 2
## 2 Dave w 5 FALSE 4
## 3 Efi w 8 TRUE 3
## 4 Emi d 12 TRUE 1
## 5 Hasi m 1 TRUE 1
## 6 Ide w 2 TRUE 2
## 7 Momsi d 3 FALSE 1
## 8 Ole d 9 TRUE 4
## 9 Ray m 10 TRUE 3
## 10 Ryu m 4 TRUE 4
## 11 Sam w 11 FALSE 3
## 12 Zoid d 6 FALSE 2Die Daten wurden korrekt anhand der Spitznamen zusammengefügt.
Ebenso wie bei der Matrix lassen sich die einzelnen Spalten und Zeilen referenzieren, indem wir in eckigen Klammern die gewünschte Position angeben.
# Zeige nur die erste Spalte
MeinDatenframe[,1]## [1] m w d m w d m w d m w d
## Levels: d m wDie einzelnen Spalten des Datenframes lassen sich auch über ihren Namen referenzieren. Hierfür schreiben wir ein Dollarzeichen $ und hängen den Spaltennamen daran.
# Zeige nur Spalte "geschlecht"
MeinDatenframe$geschlecht## [1] m w d m w d m w d m w d
## Levels: d m w# Zeige nur Spalte "angemeldet"
MeinDatenframe$angemeldet## [1] TRUE TRUE FALSE TRUE FALSE FALSE FALSE TRUE TRUE TRUE FALSE TRUESo können wir auch die jeweiligen Datentypen der Spaltenwerte überprüfen.
# welcher Datentyp liegt in Spalte "angemeldet" vor?
class(MeinDatenframe$angemeldet)## [1] "logical"# welcher Datentyp liegt in Spalte "spitzname" vor?
class(MeinDatenframe$spitzname)## [1] "character"# welcher Datentyp liegt in der 1. Spalte vor?
class(MeinDatenframe[, 1])## [1] "factor"Möchten wir uns die Fälle (also die Zeilen) ausgeben lassen, erfolgt dies mit
# Zeige Fall Nummer 4
MeinDatenframe[4,]## geschlecht spitzname hausnummer angemeldet
## 4 m Ryu 4 TRUEoder für Fallserien per
# Zeige Fälle Nummer 2 bis 5
MeinDatenframe[2:5,]## geschlecht spitzname hausnummer angemeldet
## 2 w Ide 2 TRUE
## 3 d Momsi 3 FALSE
## 4 m Ryu 4 TRUE
## 5 w Dave 5 FALSEWir können auch bedingte Ausgaben erzeugen.
# Zeige nur die Fälle mit "hausnummer" kleiner als 5
MeinDatenframe[MeinDatenframe$hausnummer<5, ]## geschlecht spitzname hausnummer angemeldet
## 1 m Hasi 1 TRUE
## 2 w Ide 2 TRUE
## 3 d Momsi 3 FALSE
## 4 m Ryu 4 TRUE# Zeige nur die Fälle mit "angemeldet" = TRUE
MeinDatenframe[MeinDatenframe$angemeldet==T, ]## geschlecht spitzname hausnummer angemeldet
## 1 m Hasi 1 TRUE
## 2 w Ide 2 TRUE
## 4 m Ryu 4 TRUE
## 8 w Efi 8 TRUE
## 9 d Ole 9 TRUE
## 10 m Ray 10 TRUE
## 12 d Emi 12 TRUEDies kann auch verknüpft werden:
# Zeige nur die Fälle mit "angemeldet" = TRUE # und "hausnummer" größer 6
MeinDatenframe[(MeinDatenframe$angemeldet==T) & (MeinDatenframe$hausnummer>6),]## geschlecht spitzname hausnummer angemeldet
## 8 w Efi 8 TRUE
## 9 d Ole 9 TRUE
## 10 m Ray 10 TRUE
## 12 d Emi 12 TRUEBeachten Sie, dass wir jedes Mal, wenn wir auf eine Spalte des Datenframes referenzieren möchten, den Datenframe-Namen mit einem Dollarzeichen $ schreiben müssen. Tun wir das nicht, sucht R nach einer Variable im Workspace, und nutzt dann deren Werte. Dies ist ein häufiger Anfängerfehler!
Die ständige Angabe des Datenframes macht die Befehle recht lang.
Mit der Funktion with() können wir uns die Referenzierung mit $ sparen. Wir übergeben der Funktion with() unser Datenframe, und sagen dann, was damit getan werden soll.
# mit Funktion with() wird es leichter
with(MeinDatenframe, MeinDatenframe[hausnummer>4 & angemeldet==F, ])## geschlecht spitzname hausnummer angemeldet
## 5 w Dave 5 FALSE
## 6 d Zoid 6 FALSE
## 7 m Adu 7 FALSE
## 11 w Sam 11 FALSE# mit Funktion with() wird es leichter
with(MeinDatenframe, spitzname[hausnummer>4 & angemeldet==F])## [1] "Dave" "Zoid" "Adu" "Sam"Das funktioniert in Kombination mit jeder anderen Funktion.
# Summe der Hausnummern
with(MeinDatenframe, sum(hausnummer))## [1] 78# Häufigkeit von geschlecht
with(MeinDatenframe, table(geschlecht))## geschlecht
## d m w
## 4 4 4Ähnlich wie bei Matrizen können wir die Zeilen- und Spaltentitel anpassen. Mit colnames() können wir die Spalten umbenennen.
# benenne Spalten des Datenframes
colnames(MeinDatenframe) <- c("Sex", "Nickname", "House", "confirmed")
# anzeigen
MeinDatenframe## Sex Nickname House confirmed
## 1 m Hasi 1 TRUE
## 2 w Ide 2 TRUE
## 3 d Momsi 3 FALSE
## 4 m Ryu 4 TRUE
## 5 w Dave 5 FALSE
## 6 d Zoid 6 FALSE
## 7 m Adu 7 FALSE
## 8 w Efi 8 TRUE
## 9 d Ole 9 TRUE
## 10 m Ray 10 TRUE
## 11 w Sam 11 FALSE
## 12 d Emi 12 TRUEDementsprechend können mit rownames() die Zeilen umbenannt werden.
# benenne Spalten des Datenframes
rownames(MeinDatenframe) <- c("Eins", "Zwei", "Drei", "Vier", "Fünf", "Sechs", "Sieben", "Acht", "Neun", "Zehn", "Elf", "Zwölf")
# anzeigen
MeinDatenframe## Sex Nickname House confirmed
## Eins m Hasi 1 TRUE
## Zwei w Ide 2 TRUE
## Drei d Momsi 3 FALSE
## Vier m Ryu 4 TRUE
## Fünf w Dave 5 FALSE
## Sechs d Zoid 6 FALSE
## Sieben m Adu 7 FALSE
## Acht w Efi 8 TRUE
## Neun d Ole 9 TRUE
## Zehn m Ray 10 TRUE
## Elf w Sam 11 FALSE
## Zwölf d Emi 12 TRUEDas sollte bei einem Datenframe nach dem Tidy Data Prinzip (siehe Kapitel 24))) aber niemals notwendig sein.
Mit der Funktion class() kann die Datenklasse angezeigt werden.
# welcher Datentyp ist "MeinDatenframe"
class(MeinDatenframe)## [1] "data.frame"Mit der Funktion is.data.frame() kann zudem geprüft werden, ob ein Objekt ein Faktor ist.
# ist "MeinDatenframe" ein Datenframe?
is.data.frame(MeinDatenframe)## [1] TRUE# ist "mymatrix" ein Datenframe?
is.data.frame(mymatrix)## [1] FALSEIn RStudio werden die Variablen und Datensätze im Datenfenster oben rechts angezeigt (Abbildung 8.2).
Wenn Sie hier auf einen Datensatz klicken, z.B. auf MeinDatenframe, so werden Ihnen die Inhalte (also die Werte) des Datensatzes im Scriptfenster angezeigt (Abbildung 8.3).
Wir können mit der Funktion as.data.frame() Objekte in ein Datenframe umwandeln. Schauen wir uns erneut die auf Seite erzeugte Matrix der Beschäftigten in den Pflegeberufen an.
# zeige Matrix
Pflegeberufe## 1999 2001 2003 2005 2007 2009 2011 2013
## Krankenpflegeassistenz 16624 19061 19478 21537 27731 36481 46517 54371
## Altenpflegehilfe 55770 52710 49727 45776 48326 47903 47978 48363
## Kinderkrankenpflege 47779 48203 48822 48519 49080 49307 48291 48937
## Krankenpflege 430983 436767 444783 449355 457322 465446 468192 472580
## Altenpflege 109161 124879 141965 158817 178902 194195 208304 227154
## 2015
## Krankenpflegeassistenz 64127
## Altenpflegehilfe 49507
## Kinderkrankenpflege 48913
## Krankenpflege 476416
## Altenpflege 246412Mit der Funktion as.data.frame() wandeln wir die Matrix in ein Datenframe um.
# wandle Matrix in Datenframe um
Pflegeframe <- as.data.frame(Pflegeberufe)
# anzeigen
Pflegeframe## 1999 2001 2003 2005 2007 2009 2011 2013
## Krankenpflegeassistenz 16624 19061 19478 21537 27731 36481 46517 54371
## Altenpflegehilfe 55770 52710 49727 45776 48326 47903 47978 48363
## Kinderkrankenpflege 47779 48203 48822 48519 49080 49307 48291 48937
## Krankenpflege 430983 436767 444783 449355 457322 465446 468192 472580
## Altenpflege 109161 124879 141965 158817 178902 194195 208304 227154
## 2015
## Krankenpflegeassistenz 64127
## Altenpflegehilfe 49507
## Kinderkrankenpflege 48913
## Krankenpflege 476416
## Altenpflege 246412Kommen wir noch einmal auf das Konzept Tidy Data zurück (siehe Kapitel 24)). Ein Datenframe sollte möglichst so aufgebaut sein, dass jeweils ein Fall pro Zeile abgebildet wird. Das ist bei unserem Datenframe Pflegeframe aber nicht der Fall.
Ein Datenframe würde sich aus der Matrix jede Beschäftigtenzahl einzeln vornehmen, um dann zu fragen “aus welchem Jahr stammt diese Zahl?” und “aus welcher Berufsgruppe stammt diese Zahl?”. Das heisst, es würde hinterher dieser Struktur folgen:
Jahr Berufsgruppe Wert
1 1999 Krankenpflege 430983
2 2001 Krankenpflege 436767
3 2001 Altenpflege 124879
4 2003 Altenpflegehilfe 49727
( . . . )Man spricht in diesem Zusammenhang von long table und wide table. Die Matrix der Pflegeberufe stellt dabei die wide table, die breite Tabelle dar. “Breit” bedeutet, dass die Tabelle, wenn wir ihr nun 10 weitere Jahrgänge hinzufügen würden, immer breiter und breiter werden würde.
Unser angestrebtes Tidy-Data-Datenframe ist vom Typ long table, da die Tabelle, wenn wir ihr Daten hinzufügen würden, immer länger und länger werden würde.
Wie formen wir unsere Matrix in ein Tidy Data-Datenframe, also in eine long table, um?
Mit der Funktion expand.grid() kann ein Datenframe mit Wertepaaren erzeugt werden. Für unser Beispiel mit den Pflegeberufen brauchen wir im Datenframe je eine Zeile für alle möglichen Kombinationen aus Jahr und Berufsgruppe. Die Funktion expand.grid() erzeugt genau solche Paarungen. Idealerweise sind die benötigten Werte (alle Jahre und alle Berufsgruppen) als Zeilen- und Spaltennamen in der Matrix Pflegeberufe vorhanden.
# erzeuge ein Tidy-Data-Dataframe
# mit allen möglichen Kombinationen
# aus Jahren und Berufsgruppen
new <- expand.grid( colnames(Pflegeberufe), rownames(Pflegeberufe))
# anzeigen
new## Var1 Var2
## 1 1999 Krankenpflegeassistenz
## 2 2001 Krankenpflegeassistenz
## 3 2003 Krankenpflegeassistenz
## 4 2005 Krankenpflegeassistenz
## 5 2007 Krankenpflegeassistenz
## 6 2009 Krankenpflegeassistenz
## 7 2011 Krankenpflegeassistenz
## 8 2013 Krankenpflegeassistenz
## 9 2015 Krankenpflegeassistenz
## 10 1999 Altenpflegehilfe
## 11 2001 Altenpflegehilfe
## 12 2003 Altenpflegehilfe
## 13 2005 Altenpflegehilfe
## 14 2007 Altenpflegehilfe
## 15 2009 Altenpflegehilfe
## 16 2011 Altenpflegehilfe
## 17 2013 Altenpflegehilfe
## 18 2015 Altenpflegehilfe
## 19 1999 Kinderkrankenpflege
## 20 2001 Kinderkrankenpflege
## 21 2003 Kinderkrankenpflege
## 22 2005 Kinderkrankenpflege
## 23 2007 Kinderkrankenpflege
## 24 2009 Kinderkrankenpflege
## 25 2011 Kinderkrankenpflege
## 26 2013 Kinderkrankenpflege
## 27 2015 Kinderkrankenpflege
## 28 1999 Krankenpflege
## 29 2001 Krankenpflege
## 30 2003 Krankenpflege
## 31 2005 Krankenpflege
## 32 2007 Krankenpflege
## 33 2009 Krankenpflege
## 34 2011 Krankenpflege
## 35 2013 Krankenpflege
## 36 2015 Krankenpflege
## 37 1999 Altenpflege
## 38 2001 Altenpflege
## 39 2003 Altenpflege
## 40 2005 Altenpflege
## 41 2007 Altenpflege
## 42 2009 Altenpflege
## 43 2011 Altenpflege
## 44 2013 Altenpflege
## 45 2015 AltenpflegeMit der Funktion cbind() können wir nun die Zahlenwerte aus der Matrix als neue Spalte an das Datenframe anhängen. Dafür müssen die Werte in Form eines Vektors vorliegen.
Um die Matrix als Vektor auszugeben nutzen wir die Funktion as.vector()
# stelle die Matrix als Vektor dar
as.vector(Pflegeberufe)## [1] 16624 55770 47779 430983 109161 19061 52710 48203 436767 124879
## [11] 19478 49727 48822 444783 141965 21537 45776 48519 449355 158817
## [21] 27731 48326 49080 457322 178902 36481 47903 49307 465446 194195
## [31] 46517 47978 48291 468192 208304 54371 48363 48937 472580 227154
## [41] 64127 49507 48913 476416 246412Wie Sie sehen, überführt R die Matrix spaltenweise in den Vektor. Für unser neues Datenframe bräuchten wir aber einen zeilenorientierten Vektor, damit er mit der Reihenfolge der Einträge (Paarung aus Jahr und Berufsgruppe) übereinstimmt. Um einen reihenorientierten Vektor zu erzeugen muss die Matrix mit der Funktion t() transpoiniert werden.
# stelle die Matrix als Vektor dar
# zeilenorientiert
as.vector(t(Pflegeberufe))## [1] 16624 19061 19478 21537 27731 36481 46517 54371 64127 55770
## [11] 52710 49727 45776 48326 47903 47978 48363 49507 47779 48203
## [21] 48822 48519 49080 49307 48291 48937 48913 430983 436767 444783
## [31] 449355 457322 465446 468192 472580 476416 109161 124879 141965 158817
## [41] 178902 194195 208304 227154 246412Diesen Vektor fügen wir nun per cbind() dem Datenframe als neue Spalte hinzu
# füge Spalte hinzu
Pflegeframe <- cbind(new, as.vector(t(Pflegeberufe)))
# benenne die Spalten neu
colnames(Pflegeframe) <- c("Jahr", "Berufsgruppe", "Anzahl")
# zeige an
Pflegeframe## Jahr Berufsgruppe Anzahl
## 1 1999 Krankenpflegeassistenz 16624
## 2 2001 Krankenpflegeassistenz 19061
## 3 2003 Krankenpflegeassistenz 19478
## 4 2005 Krankenpflegeassistenz 21537
## 5 2007 Krankenpflegeassistenz 27731
## 6 2009 Krankenpflegeassistenz 36481
## 7 2011 Krankenpflegeassistenz 46517
## 8 2013 Krankenpflegeassistenz 54371
## 9 2015 Krankenpflegeassistenz 64127
## 10 1999 Altenpflegehilfe 55770
## 11 2001 Altenpflegehilfe 52710
## 12 2003 Altenpflegehilfe 49727
## 13 2005 Altenpflegehilfe 45776
## 14 2007 Altenpflegehilfe 48326
## 15 2009 Altenpflegehilfe 47903
## 16 2011 Altenpflegehilfe 47978
## 17 2013 Altenpflegehilfe 48363
## 18 2015 Altenpflegehilfe 49507
## 19 1999 Kinderkrankenpflege 47779
## 20 2001 Kinderkrankenpflege 48203
## 21 2003 Kinderkrankenpflege 48822
## 22 2005 Kinderkrankenpflege 48519
## 23 2007 Kinderkrankenpflege 49080
## 24 2009 Kinderkrankenpflege 49307
## 25 2011 Kinderkrankenpflege 48291
## 26 2013 Kinderkrankenpflege 48937
## 27 2015 Kinderkrankenpflege 48913
## 28 1999 Krankenpflege 430983
## 29 2001 Krankenpflege 436767
## 30 2003 Krankenpflege 444783
## 31 2005 Krankenpflege 449355
## 32 2007 Krankenpflege 457322
## 33 2009 Krankenpflege 465446
## 34 2011 Krankenpflege 468192
## 35 2013 Krankenpflege 472580
## 36 2015 Krankenpflege 476416
## 37 1999 Altenpflege 109161
## 38 2001 Altenpflege 124879
## 39 2003 Altenpflege 141965
## 40 2005 Altenpflege 158817
## 41 2007 Altenpflege 178902
## 42 2009 Altenpflege 194195
## 43 2011 Altenpflege 208304
## 44 2013 Altenpflege 227154
## 45 2015 Altenpflege 246412Der vollständige Code, ohne Hilfsdatenframe new, zur Überführung der Matrix Pflegeberufe in das Tidy-Data-Datenframe Pflegeframe sieht also so aus:
# füge Spalte hinzu
Pflegeframe <- cbind(expand.grid( colnames(Pflegeberufe), rownames(Pflegeberufe)), as.vector(t(Pflegeberufe)))
# benenne die Spalten neu
colnames(Pflegeframe) <- c("Jahr", "Berufsgruppe", "Anzahl")
# zeige erste 12 Fälle an
head(Pflegeframe, 12)## Jahr Berufsgruppe Anzahl
## 1 1999 Krankenpflegeassistenz 16624
## 2 2001 Krankenpflegeassistenz 19061
## 3 2003 Krankenpflegeassistenz 19478
## 4 2005 Krankenpflegeassistenz 21537
## 5 2007 Krankenpflegeassistenz 27731
## 6 2009 Krankenpflegeassistenz 36481
## 7 2011 Krankenpflegeassistenz 46517
## 8 2013 Krankenpflegeassistenz 54371
## 9 2015 Krankenpflegeassistenz 64127
## 10 1999 Altenpflegehilfe 55770
## 11 2001 Altenpflegehilfe 52710
## 12 2003 Altenpflegehilfe 49727In der Datenklasse Listen können beliebige Datenobjekte (Vektor, Faktor, Datenframe, Matrizen) zusammengefasst werden. Listen sind also eine Ansammlung an Datenobjekten, ähnlich wie ein Schrank oder ein Koffer, in welchem man “sein Zeug” ablegt. Wir generieren testweise eine Liste aus den Datenobjekten, die wir bislang erzeugt haben. Dies erfolgt in mit der Funktion list().
# erzeuge eine Liste aus den Datenobjekten
# "MeinDatenframe", "mymatrix", "geschlecht" und "logical"
MeineListe <- list(MeinDatenframe, mymatrix, geschlecht, logical)
# anzeigen
MeineListe## [[1]]
## geschlecht spitzname hausnummer angemeldet
## Eins m Hasi 1 TRUE
## Zwei w Ide 2 TRUE
## Drei d Momsi 3 FALSE
## Vier m Ryu 4 TRUE
## Fünf w Dave 5 FALSE
## Sechs d Zoid 6 FALSE
## Sieben m Adu 7 FALSE
## Acht w Efi 8 TRUE
## Neun d Ole 9 TRUE
## Zehn m Ray 10 TRUE
## Elf w Sam 11 FALSE
## Zwölf d Emi 12 TRUE
##
## [[2]]
## a b c d e
## I 11 21 31 41 51
## II 12 22 32 42 52
## III 13 23 33 43 53
## IV 14 24 34 44 54
## V 15 25 35 45 55
##
## [[3]]
## [1] m w d m w d m w d m w d
## Levels: d m w
##
## [[4]]
## function (length = 0L)
## .Internal(vector("logical", length))
## <bytecode: 0x6235e93fc498>
## <environment: namespace:base>Wie Sie sehen, werden die einzelnen Positionen der Datenobjekte durch doppelte eckige Klammer angezeigt ([[1]] ist unser Datenframe, [[2]] unsere Matrix, usw.) und können über diese auch referenziert werden.
# zeige Objekt 1 (= unser Datenframe)
MeineListe[[1]]## geschlecht spitzname hausnummer angemeldet
## Eins m Hasi 1 TRUE
## Zwei w Ide 2 TRUE
## Drei d Momsi 3 FALSE
## Vier m Ryu 4 TRUE
## Fünf w Dave 5 FALSE
## Sechs d Zoid 6 FALSE
## Sieben m Adu 7 FALSE
## Acht w Efi 8 TRUE
## Neun d Ole 9 TRUE
## Zehn m Ray 10 TRUE
## Elf w Sam 11 FALSE
## Zwölf d Emi 12 TRUE# zeige Objekt 3 (= variable "geschlecht")
MeineListe[[3]]## [1] m w d m w d m w d m w d
## Levels: d m wDie Werte der jeweiligen Objekte können anschließend wie gewohnt referenziert werden.
# zeige Objekt 3, aber nur den 5. Wert
MeineListe[[3]][5]## [1] w
## Levels: d m w# zeige Objekt 2, aber nur die 3. Spalte
MeineListe[[2]][, 3]## I II III IV V
## 31 32 33 34 35