# a)
(15.4 + 0.2) * (7-10.2) / 9[1] -5.546667# b)
5/10 + 11/7 - 8/3[1] -0.5952381# c)
(13+2)^3 * (17-8)^2 / 9[1] 30375# d)
sqrt( ((1+3)*25) / (5*5-15)^2 )[1] 1Gerade als Anfänger:in sollten Sie zumindest versuchen, die Aufgaben selbstständig zu lösen, bevor Sie sich die Lösungswege anschauen. Kopf hoch, Sie schaffen das!
# a)
(15.4 + 0.2) * (7-10.2) / 9[1] -5.546667# b)
5/10 + 11/7 - 8/3[1] -0.5952381# c)
(13+2)^3 * (17-8)^2 / 9[1] 30375# d)
sqrt( ((1+3)*25) / (5*5-15)^2 )[1] 1zahlen <- c(1:100)
#anschauen
zahlen [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18
[19] 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36
[37] 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54
[55] 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72
[73] 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90
[91] 91 92 93 94 95 96 97 98 99 100worte <- c("Apfel", "Birne", "Postauto")
# anschauen
worte[1] "Apfel" "Birne" "Postauto"# mit rep() 30mal "worte" wiederholen
worte30 <- rep(worte, 30)
# anschauen
worte30 [1] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[7] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[13] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[19] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[25] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[31] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[37] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[43] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[49] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[55] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[61] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[67] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[73] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[79] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"
[85] "Apfel" "Birne" "Postauto" "Apfel" "Birne" "Postauto"sample(1:500, 200, replace = FALSE) [1] 373 78 221 158 261 442 56 376 432 247 203 377 217 133 323 15 186 193
[19] 255 171 219 174 187 422 183 30 18 101 288 406 305 81 5 146 396 96
[37] 296 170 403 359 139 6 163 453 455 349 467 93 497 251 130 385 107 167
[55] 457 145 430 102 273 151 425 120 380 220 334 300 117 161 360 346 192 65
[73] 405 50 206 231 476 289 336 419 58 375 489 152 287 340 142 264 155 4
[91] 282 368 233 108 24 185 471 483 447 350 268 331 386 236 190 475 382 123
[109] 309 257 320 482 495 262 119 212 99 214 112 116 256 277 383 246 181 141
[127] 498 82 10 259 254 325 434 118 411 143 59 196 160 134 176 191 88 124
[145] 54 458 345 281 433 195 414 222 57 35 95 362 358 480 302 48 204 301
[163] 90 424 464 22 129 69 469 402 384 487 165 83 20 466 470 61 303 164
[181] 232 55 201 179 280 381 311 32 244 378 182 105 286 110 128 485 395 198
[199] 344 113sample(1:500, 200, replace = TRUE) [1] 162 354 400 184 358 10 374 366 339 130 21 152 145 496 458 465 483 140
[19] 276 441 436 398 252 111 476 146 76 81 148 354 57 413 297 280 284 24
[37] 374 189 363 157 65 269 72 441 19 266 227 142 302 352 131 265 291 41
[55] 177 165 139 227 427 13 305 261 108 357 379 163 256 2 132 437 232 246
[73] 231 413 157 436 263 332 401 82 104 230 240 152 77 147 481 259 190 75
[91] 162 40 21 68 128 218 132 207 95 245 153 133 441 316 412 263 194 378
[109] 358 461 184 255 204 39 197 21 93 108 450 3 172 479 283 25 434 257
[127] 58 355 236 351 3 492 348 468 51 34 156 135 396 57 8 443 262 483
[145] 466 151 191 139 258 369 489 442 224 274 306 311 457 56 102 348 10 365
[163] 149 264 98 180 274 407 324 88 360 265 399 282 447 463 32 56 355 81
[181] 433 495 246 35 307 236 36 358 430 202 22 54 458 457 254 282 259 30
[199] 89 252KHAufenthalte.
KHAufenthalte <- c(1,0,0,3,1,5,1,2,2,0,1,0,5,2,1,0,1,0,0,4,0,1,1,3,0,
1,1,1,3,1,0,1,4,2,0,3,1,1,7,2,0,2,1,3,0,0,0,0,6,1,
1,2,1,0,1,0,3,0,1,3,0,5,2,1,0,2,4,0,1,1,3,0,1,2,1,
1,1,1,2,2,0,3,0,1,0,1,0,0,0,5,0,4,1,2,2,7,1,3,1,5)
#anschauen
KHAufenthalte [1] 1 0 0 3 1 5 1 2 2 0 1 0 5 2 1 0 1 0 0 4 0 1 1 3 0 1 1 1 3 1 0 1 4 2 0 3 1
[38] 1 7 2 0 2 1 3 0 0 0 0 6 1 1 2 1 0 1 0 3 0 1 3 0 5 2 1 0 2 4 0 1 1 3 0 1 2
[75] 1 1 1 1 2 2 0 3 0 1 0 1 0 0 0 5 0 4 1 2 2 7 1 3 1 5# ersten und dritten Wert enfernen
KHAufenthalte <- KHAufenthalte[-c(1,3)]
#anschauen
KHAufenthalte [1] 0 3 1 5 1 2 2 0 1 0 5 2 1 0 1 0 0 4 0 1 1 3 0 1 1 1 3 1 0 1 4 2 0 3 1 1 7 2
[39] 0 2 1 3 0 0 0 0 6 1 1 2 1 0 1 0 3 0 1 3 0 5 2 1 0 2 4 0 1 1 3 0 1 2 1 1 1 1
[77] 2 2 0 3 0 1 0 1 0 0 0 5 0 4 1 2 2 7 1 3 1 5# 7 und 2 hinzufügen
KHAufenthalte <- c(KHAufenthalte, 7, 2)
#anschauen
KHAufenthalte [1] 0 3 1 5 1 2 2 0 1 0 5 2 1 0 1 0 0 4 0 1 1 3 0 1 1 1 3 1 0 1 4 2 0 3 1 1 7
[38] 2 0 2 1 3 0 0 0 0 6 1 1 2 1 0 1 0 3 0 1 3 0 5 2 1 0 2 4 0 1 1 3 0 1 2 1 1
[75] 1 1 2 2 0 3 0 1 0 1 0 0 0 5 0 4 1 2 2 7 1 3 1 5 7 2hospital.stays um.
# umbenennen
hospital.stays <- KHAufenthaltecut()-Funktion in die Klassen \(0\), \(1-2\) und \(>2\) Aufenthalte.
# cut
cut(hospital.stays, breaks=c(0,1,3,Inf), right=FALSE) [1] [0,1) [3,Inf) [1,3) [3,Inf) [1,3) [1,3) [1,3) [0,1) [1,3)
[10] [0,1) [3,Inf) [1,3) [1,3) [0,1) [1,3) [0,1) [0,1) [3,Inf)
[19] [0,1) [1,3) [1,3) [3,Inf) [0,1) [1,3) [1,3) [1,3) [3,Inf)
[28] [1,3) [0,1) [1,3) [3,Inf) [1,3) [0,1) [3,Inf) [1,3) [1,3)
[37] [3,Inf) [1,3) [0,1) [1,3) [1,3) [3,Inf) [0,1) [0,1) [0,1)
[46] [0,1) [3,Inf) [1,3) [1,3) [1,3) [1,3) [0,1) [1,3) [0,1)
[55] [3,Inf) [0,1) [1,3) [3,Inf) [0,1) [3,Inf) [1,3) [1,3) [0,1)
[64] [1,3) [3,Inf) [0,1) [1,3) [1,3) [3,Inf) [0,1) [1,3) [1,3)
[73] [1,3) [1,3) [1,3) [1,3) [1,3) [1,3) [0,1) [3,Inf) [0,1)
[82] [1,3) [0,1) [1,3) [0,1) [0,1) [0,1) [3,Inf) [0,1) [3,Inf)
[91] [1,3) [1,3) [1,3) [3,Inf) [1,3) [3,Inf) [1,3) [3,Inf) [3,Inf)
[100] [1,3)
Levels: [0,1) [1,3) [3,Inf)# mit custom labels
cut(hospital.stays, breaks=c(0,1,3,Inf), right=FALSE,
labels=c("0", "1-2", "mehr als 2")) [1] 0 mehr als 2 1-2 mehr als 2 1-2 1-2
[7] 1-2 0 1-2 0 mehr als 2 1-2
[13] 1-2 0 1-2 0 0 mehr als 2
[19] 0 1-2 1-2 mehr als 2 0 1-2
[25] 1-2 1-2 mehr als 2 1-2 0 1-2
[31] mehr als 2 1-2 0 mehr als 2 1-2 1-2
[37] mehr als 2 1-2 0 1-2 1-2 mehr als 2
[43] 0 0 0 0 mehr als 2 1-2
[49] 1-2 1-2 1-2 0 1-2 0
[55] mehr als 2 0 1-2 mehr als 2 0 mehr als 2
[61] 1-2 1-2 0 1-2 mehr als 2 0
[67] 1-2 1-2 mehr als 2 0 1-2 1-2
[73] 1-2 1-2 1-2 1-2 1-2 1-2
[79] 0 mehr als 2 0 1-2 0 1-2
[85] 0 0 0 mehr als 2 0 mehr als 2
[91] 1-2 1-2 1-2 mehr als 2 1-2 mehr als 2
[97] 1-2 mehr als 2 mehr als 2 1-2
Levels: 0 1-2 mehr als 2Groesse und Gewicht.
Groesse <- c(1.68, 1.87, 1.95, 1.74, 1.80,
1.75, 1.59, 1.77, 1.82, 1.74)
Gewicht <- c(78500, 110100, 97500, 69200, 82500,
71500, 81500, 87200, 75500, 65500)
# anzeigen
Groesse [1] 1.68 1.87 1.95 1.74 1.80 1.75 1.59 1.77 1.82 1.74Gewicht [1] 78500 110100 97500 69200 82500 71500 81500 87200 75500 65500Kilogramm.
# Rechne Gramm in Kilogramm um
Kilogramm <- Gewicht/1000
# anzeigen
Kilogramm [1] 78.5 110.1 97.5 69.2 82.5 71.5 81.5 87.2 75.5 65.5BMI.
# BMI berechnen
BMI <- Kilogramm / (Groesse^2)
# anzeigen
BMI [1] 27.81321 31.48503 25.64103 22.85639 25.46296 23.34694 32.23765 27.83364
[9] 22.79314 21.63430Groesse, Gewicht (aber in Kilogramm) und BMI zu einem Datenframe zusammen.
# Datenframe erzeugen
df <- data.frame(Groesse, Gewicht=Kilogramm, BMI)
# anzeigen
df Groesse Gewicht BMI
1 1.68 78.5 27.81321
2 1.87 110.1 31.48503
3 1.95 97.5 25.64103
4 1.74 69.2 22.85639
5 1.80 82.5 25.46296
6 1.75 71.5 23.34694
7 1.59 81.5 32.23765
8 1.77 87.2 27.83364
9 1.82 75.5 22.79314
10 1.74 65.5 21.63430df[c(4, 7, 9),] Groesse Gewicht BMI
4 1.74 69.2 22.85639
7 1.59 81.5 32.23765
9 1.82 75.5 22.79314df[df$Gewicht > 80 , ] Groesse Gewicht BMI
2 1.87 110.1 31.48503
3 1.95 97.5 25.64103
5 1.80 82.5 25.46296
7 1.59 81.5 32.23765
8 1.77 87.2 27.83364Monate, in welcher die 12 ausgeschriebenen Monatsnamen in korrekter Levelreihenfolge enthalten sind.
# ordinaler Faktor
Monate <- factor(c("Januar", "Februar", "März", "April", "Mai", "Juni",
"Juli", "August", "September", "Oktober", "November",
"Dezember"),
levels= c("Januar", "Februar", "März", "April", "Mai",
"Juni", "Juli", "August", "September", "Oktober",
"November", "Dezember"),
ordered=TRUE )
# anzeigen
Monate [1] Januar Februar März April Mai Juni Juli
[8] August September Oktober November Dezember
12 Levels: Januar < Februar < März < April < Mai < Juni < Juli < ... < DezemberWir können uns aber auch ein bisschen Schreibarbeit ersparen.
# Hilfsvektor erzeugen
dummy <- c("Januar", "Februar", "März", "April", "Mai", "Juni", "Juli",
"August", "September", "Oktober", "November", "Dezember")
# ordinaler Faktor
Monate <- factor(dummy, levels=dummy, ordered=TRUE)
# anzeigen
Monate [1] Januar Februar März April Mai Juni Juli
[8] August September Oktober November Dezember
12 Levels: Januar < Februar < März < April < Mai < Juni < Juli < ... < DezemberSchulnoten, in welcher die 6 ausgeschriebenen Schulnoten in korrekter Levelreihenfolge enthalten sind.
# ordinaler Faktor
# Achten Sie auf die Reihenfolge der Schulnoten,
# wir müssen mit der schlechtesten anfangen.
Schulnoten <- c("ungenügend", "mangelhaft", "ausreichend", "befriedigend",
"gut", "sehr gut")
Schulnoten <- factor(Schulnoten, levels=Schulnoten, ordered=TRUE)
# anzeigen
Schulnoten[1] ungenügend mangelhaft ausreichend befriedigend gut
[6] sehr gut
6 Levels: ungenügend < mangelhaft < ausreichend < befriedigend < ... < sehr gutwoche, welcher die Wochentagen von Montag bis Sonntag mit korrekter Levelreihenfolge enthält.
Um etwas Schreibarbeit zu sparen, schreiben wir die Tage in ein eigenes Objekt. Dieses kann dann für factor() und levels genutzt werden.
# Tage in Objekt speichern
tage <- c("Montag", "Dienstag", "Mittwoch",
"Donnerstag", "Freitag", "Samstag", "Sonntag")
# Dieses Objekt dann wiederverwenden
woche <- factor(tage,
ordered = TRUE,
levels=tage)
# anzeigen
woche[1] Montag Dienstag Mittwoch Donnerstag Freitag Samstag Sonntag
7 Levels: Montag < Dienstag < Mittwoch < Donnerstag < Freitag < ... < Sonntaglevels(woche) <- c("Monday", "Tuesday", "Wednesday", "Thursday",
"Friday", "Saturday", "Sunday")
# anzeigen
woche[1] Monday Tuesday Wednesday Thursday Friday Saturday Sunday
7 Levels: Monday < Tuesday < Wednesday < Thursday < Friday < ... < Sunday# ordinaler Faktor
f <- factor(c("vielleicht", "glaube nicht", "nein", "glaube nicht",
"ja", "glaube schon", "vielleicht", "nein", "glaube nicht",
"ja", "ja", "glaube schon", "ja", "ja", "nein",
"glaube nicht", "glaube schon", "vielleicht", "vielleicht",
"glaube nicht", "vielleicht", "glaube nicht", "nein",
"glaube nicht", "ja", "glaube schon", "vielleicht", "nein",
"glaube nicht", "ja", "ja", "glaube schon", "ja", "ja",
"nein", "glaube nicht", "glaube schon", "vielleicht",
"vielleicht", "glaube nicht"),
levels=c("nein", "glaube nicht", "vielleicht", "glaube schon", "ja"),
ordered=TRUE)
# anzeigen
f [1] vielleicht glaube nicht nein glaube nicht ja
[6] glaube schon vielleicht nein glaube nicht ja
[11] ja glaube schon ja ja nein
[16] glaube nicht glaube schon vielleicht vielleicht glaube nicht
[21] vielleicht glaube nicht nein glaube nicht ja
[26] glaube schon vielleicht nein glaube nicht ja
[31] ja glaube schon ja ja nein
[36] glaube nicht glaube schon vielleicht vielleicht glaube nicht
Levels: nein < glaube nicht < vielleicht < glaube schon < ja-2, -1, 0, 1, 2.
# Levelnamen ändern
levels(f) <- c("-2", "-1", "0", "1", "2")
# anzeigen
f [1] 0 -1 -2 -1 2 1 0 -2 -1 2 2 1 2 2 -2 -1 1 0 0 -1 0 -1 -2 -1 2
[26] 1 0 -2 -1 2 2 1 2 2 -2 -1 1 0 0 -1
Levels: -2 < -1 < 0 < 1 < 2Name”, “Alter” und “Geschlecht” und fügen Sie drei Beispielzeilen mit Daten hinzu.
df <- data.frame(Name = c("Max", "Lisa", "Tom"),
Alter = c(22, 25, 30),
Geschlecht = factor(c("m", "w", "m")))
# anzeigen
df Name Alter Geschlecht
1 Max 22 m
2 Lisa 25 w
3 Tom 30 mHobbys” hinzu und füllen Sie diese mit drei Beispielwerten.
df$Hobbys <- c("Sport", "Musik", "Reisen")
# anzeigen
df Name Alter Geschlecht Hobbys
1 Max 22 m Sport
2 Lisa 25 w Musik
3 Tom 30 m Reisendf$Name[2] <- "Kunigunde"
# anzeigen
df Name Alter Geschlecht Hobbys
1 Max 22 m Sport
2 Kunigunde 25 w Musik
3 Tom 30 m ReisenStudiengaenge mit den Variablen “Fachbereich”, “Studiengang” und “Niveau” (Bachelor/Master), und überführen Sie die oben stehenden Daten in das Datenframe.
# FB06 und FB10 haben jeweils 6 Studiengänge
# schreibe direkt als factor()
Fachbereich <- factor(c("FB06", "FB06", "FB06", "FB06", "FB06", "FB06",
"FB10", "FB10", "FB10", "FB10", "FB10", "FB10"))
## oder auch so:
Fachbereich <- factor(c(rep("FB06", 6), rep("FB10", 6)))
# Zuerst die Studiengänge von FB06, dann von FB10
# schreibe direkt als factor()
Studiengang = factor(c("BA Soziale Arbeit", "BA Kulturpädagogik",
"BA Kindheitspädagogik", "MA Soziale Arbeit",
"MA Kulturpädagogik & Kulturmanagement",
"MA Sozialmanagement", "BA Health Care Management",
"BA Medizinische Informatik",
"BA Angewandte Therapiewissenschaften", "BA Pflege",
"BA Angewandte Hebammenwissenschaft", "MA Health Care"))
# FB06 hat 3xBachelor und 3xMaster, FB010 hat 5xBachelor und 1xMaster
Niveau = factor(c("Bachelor", "Bachelor", "Bachelor", "Master", "Master",
"Master", "Bachelor", "Bachelor", "Bachelor", "Bachelor",
"Bachelor", "Master"))
# oder auch so:
Niveau = factor(c(rep("Bachelor", 3), rep("Master", 3),
rep("Bachelor", 5), "Master"))
# Füge zu Datenframe zusammen
Studiengaenge <- data.frame(Fachbereich,
Studiengang,
Niveau)
# anzeigen
Studiengaenge Fachbereich Studiengang Niveau
1 FB06 BA Soziale Arbeit Bachelor
2 FB06 BA Kulturpädagogik Bachelor
3 FB06 BA Kindheitspädagogik Bachelor
4 FB06 MA Soziale Arbeit Master
5 FB06 MA Kulturpädagogik & Kulturmanagement Master
6 FB06 MA Sozialmanagement Master
7 FB10 BA Health Care Management Bachelor
8 FB10 BA Medizinische Informatik Bachelor
9 FB10 BA Angewandte Therapiewissenschaften Bachelor
10 FB10 BA Pflege Bachelor
11 FB10 BA Angewandte Hebammenwissenschaft Bachelor
12 FB10 MA Health Care MasterKurse, in welchem die Daten aus den Tabellenspalten Haus und Kurs enthalten sind.
# Daten übertragen
Kurse <- data.frame(
Haus = c("Gryffindor", "Gryffindor", "Gryffindor", "Gryffindor",
"Hufflepuff", "Hufflepuff", "Hufflepuff", "Hufflepuff",
"Ravenclaw", "Ravenclaw", "Ravenclaw", "Ravenclaw",
"Slytherin", "Slytherin", "Slytherin", "Slytherin"),
Kurs = c("Verteidigung gegen die dunklen Künste", "Zauberkunst",
"Verwandlung", "Besenflugunterricht",
"Kräuterkunde", "Pflege magischer Geschöpfe",
"Geschichte der Zauberei", "Alte Runen",
"Arithmantik", "Astronomie",
"Verwandlung", "Verteidigung gegen die dunklen Künste",
"Zaubertränke", "Zauberkunst",
"Dunkle Künste", "Legilimentik")
)
# anzeigen
Kurse Haus Kurs
1 Gryffindor Verteidigung gegen die dunklen Künste
2 Gryffindor Zauberkunst
3 Gryffindor Verwandlung
4 Gryffindor Besenflugunterricht
5 Hufflepuff Kräuterkunde
6 Hufflepuff Pflege magischer Geschöpfe
7 Hufflepuff Geschichte der Zauberei
8 Hufflepuff Alte Runen
9 Ravenclaw Arithmantik
10 Ravenclaw Astronomie
11 Ravenclaw Verwandlung
12 Ravenclaw Verteidigung gegen die dunklen Künste
13 Slytherin Zaubertränke
14 Slytherin Zauberkunst
15 Slytherin Dunkle Künste
16 Slytherin Legilimentik# unique()
unique(Kurse$Kurs) [1] "Verteidigung gegen die dunklen Künste"
[2] "Zauberkunst"
[3] "Verwandlung"
[4] "Besenflugunterricht"
[5] "Kräuterkunde"
[6] "Pflege magischer Geschöpfe"
[7] "Geschichte der Zauberei"
[8] "Alte Runen"
[9] "Arithmantik"
[10] "Astronomie"
[11] "Zaubertränke"
[12] "Dunkle Künste"
[13] "Legilimentik" length(unique(Kurse$Kurs))[1] 13Es sind 13 Kurse in der Liste.
subset() für jedes Haus ein eigenes Datenframe
# Subsets erstellen
gryffindor <- subset(Kurse, Haus=="Gryffindor")
hufflepuff <- subset(Kurse, Haus=="Hufflepuff")
ravenclaw <- subset(Kurse, Haus=="Ravenclaw")
slytherin <- subset(Kurse, Haus=="Slytherin")# Subsets erstellen
gryffindor$Kurs <- factor(gryffindor$Kurs)
gryffindor$Haus <- factor(gryffindor$Haus)
hufflepuff$Kurs <- factor(hufflepuff$Kurs)
hufflepuff$Haus <- factor(hufflepuff$Haus)
ravenclaw$Kurs <- factor(ravenclaw$Kurs)
ravenclaw$Haus <- factor(ravenclaw$Haus)
slytherin$Kurs <- factor(slytherin$Kurs)
slytherin$Haus <- factor(slytherin$Haus)Hogwarts zusammen, in der Reihenfolge Ravenclaw, Gryffindor, Syltherin und Hufflepuff. Ändern Sie anschließend den Kurs “Geschichte der Zauberei” in “Geisterkunde” um.
# Zusammenführen
Hogwarts <- rbind(ravenclaw, gryffindor, slytherin, hufflepuff)
# Level ändern
levels(Hogwarts$Kurs)[levels(Hogwarts$Kurs)=="Geschichte der Zauberei"] <- "Geisterkunde"
# anzeigen
Hogwarts$Kurs [1] Arithmantik Astronomie
[3] Verwandlung Verteidigung gegen die dunklen Künste
[5] Verteidigung gegen die dunklen Künste Zauberkunst
[7] Verwandlung Besenflugunterricht
[9] Zaubertränke Zauberkunst
[11] Dunkle Künste Legilimentik
[13] Kräuterkunde Pflege magischer Geschöpfe
[15] Geisterkunde Alte Runen
13 Levels: Arithmantik Astronomie ... Pflege magischer GeschöpfeWenn wir “einfach so” die order()-Funktion nutzen, erhalten wir eine falsche Ausgabe.
# wird nicht korrekt sortiert
Hogwarts[order(Hogwarts$Kurs),] Haus Kurs
9 Ravenclaw Arithmantik
10 Ravenclaw Astronomie
12 Ravenclaw Verteidigung gegen die dunklen Künste
1 Gryffindor Verteidigung gegen die dunklen Künste
11 Ravenclaw Verwandlung
3 Gryffindor Verwandlung
4 Gryffindor Besenflugunterricht
2 Gryffindor Zauberkunst
14 Slytherin Zauberkunst
15 Slytherin Dunkle Künste
16 Slytherin Legilimentik
13 Slytherin Zaubertränke
8 Hufflepuff Alte Runen
7 Hufflepuff Geisterkunde
5 Hufflepuff Kräuterkunde
6 Hufflepuff Pflege magischer GeschöpfeDas liegt daran, dass Hogwarts$Kurs als Factor vorliegt, und somit nach Levelreihenfolge sortiert wird.
# Datenklasse Factor
class(Hogwarts$Kurs)[1] "factor"Wir müssen daher die Funktion as.character() um die Variable wickeln, um eine alphabetische Sortierung zu erzwingen.
# jetzt klappt es
Hogwarts[order(as.character(Hogwarts$Kurs)),] Haus Kurs
8 Hufflepuff Alte Runen
9 Ravenclaw Arithmantik
10 Ravenclaw Astronomie
4 Gryffindor Besenflugunterricht
15 Slytherin Dunkle Künste
7 Hufflepuff Geisterkunde
5 Hufflepuff Kräuterkunde
16 Slytherin Legilimentik
6 Hufflepuff Pflege magischer Geschöpfe
12 Ravenclaw Verteidigung gegen die dunklen Künste
1 Gryffindor Verteidigung gegen die dunklen Künste
11 Ravenclaw Verwandlung
3 Gryffindor Verwandlung
2 Gryffindor Zauberkunst
14 Slytherin Zauberkunst
13 Slytherin Zaubertränkesorted, und reparieren Sie die Zeilennummerierung von sorted.
# sortiert speichern
sorted <- Hogwarts[order(as.character(Hogwarts$Kurs)),]
# Zeilennummerierung reparieren
rownames(sorted) <- 1:length(sorted$Kurs)
# anzeigen
sorted Haus Kurs
1 Hufflepuff Alte Runen
2 Ravenclaw Arithmantik
3 Ravenclaw Astronomie
4 Gryffindor Besenflugunterricht
5 Slytherin Dunkle Künste
6 Hufflepuff Geisterkunde
7 Hufflepuff Kräuterkunde
8 Slytherin Legilimentik
9 Hufflepuff Pflege magischer Geschöpfe
10 Ravenclaw Verteidigung gegen die dunklen Künste
11 Gryffindor Verteidigung gegen die dunklen Künste
12 Ravenclaw Verwandlung
13 Gryffindor Verwandlung
14 Gryffindor Zauberkunst
15 Slytherin Zauberkunst
16 Slytherin Zaubertränkechol.
# Daten übertragen
chol <- data.frame(Name = c("Anna Tomie", "Bud Zillus", "Dieter Mietenplage",
"Hella Scheinwerfer", "Inge Danken", "Jason Zufall"),
Geschlecht = c("W", "M", "M", "W", "W", "M"),
Gewicht = c(85, 115, 79, 60, 57, 96),
Größe = c(179, 173, 181, 170, 158, 174),
Cholesterol = c(182, 232, 191, 200, 148, 249)
)
# anzeigen
chol Name Geschlecht Gewicht Größe Cholesterol
1 Anna Tomie W 85 179 182
2 Bud Zillus M 115 173 232
3 Dieter Mietenplage M 79 181 191
4 Hella Scheinwerfer W 60 170 200
5 Inge Danken W 57 158 148
6 Jason Zufall M 96 174 249Alter, die zwischen Name und Geschlecht liegt
# Daten übertragen
alter <- c(18, 32, 24, 35, 46, 68)
# zwischen Name und Geschlecht einfügen
chol <- data.frame(Name=chol$Name, Alter=alter, Geschlecht=chol$Geschlecht,
Gewicht=chol$Gewicht, Größe=chol$Größe,
Cholesterol=chol$Cholesterol)
# anzeigen
chol Name Alter Geschlecht Gewicht Größe Cholesterol
1 Anna Tomie 18 W 85 179 182
2 Bud Zillus 32 M 115 173 232
3 Dieter Mietenplage 24 M 79 181 191
4 Hella Scheinwerfer 35 W 60 170 200
5 Inge Danken 46 W 57 158 148
6 Jason Zufall 68 M 96 174 249# Daten übertragen
neu <- data.frame(Name="Mitch Mackes", Alter=44, Geschlecht="M", Gewicht=92,
Größe=178, Cholesterol=220)
# zusammenfügen
chol <- rbind(chol, neu)
# anzeigen
chol Name Alter Geschlecht Gewicht Größe Cholesterol
1 Anna Tomie 18 W 85 179 182
2 Bud Zillus 32 M 115 173 232
3 Dieter Mietenplage 24 M 79 181 191
4 Hella Scheinwerfer 35 W 60 170 200
5 Inge Danken 46 W 57 158 148
6 Jason Zufall 68 M 96 174 249
7 Mitch Mackes 44 M 92 178 220BMI (\(\text{BMI}=\frac{kg}{m^2}\)).
# BMI hinzufügen
# Größe muss in Meter umgerechnet werden
chol$BMI <- chol$Gewicht / (chol$Größe/100)^2
# anzeigen
chol Name Alter Geschlecht Gewicht Größe Cholesterol BMI
1 Anna Tomie 18 W 85 179 182 26.52851
2 Bud Zillus 32 M 115 173 232 38.42427
3 Dieter Mietenplage 24 M 79 181 191 24.11404
4 Hella Scheinwerfer 35 W 60 170 200 20.76125
5 Inge Danken 46 W 57 158 148 22.83288
6 Jason Zufall 68 M 96 174 249 31.70828
7 Mitch Mackes 44 M 92 178 220 29.03674Adipositas hinzu, in welcher Sie die BMI-Werte klassieren
Ein Klassierung kann auf mehrere Weisen erfolgen.
# bedingtes Referenzieren
chol$Adipositas[chol$BMI < 18.5] <- "Untergewicht"
chol$Adipositas[chol$BMI >= 18.5 & chol$BMI < 24.5] <- "Normalgewicht"
chol$Adipositas[chol$BMI >= 24.5 & chol$BMI < 30] <- "Übergewicht"
chol$Adipositas[chol$BMI >= 30] <- "Adipositas"
# anzeigen
chol Name Alter Geschlecht Gewicht Größe Cholesterol BMI
1 Anna Tomie 18 W 85 179 182 26.52851
2 Bud Zillus 32 M 115 173 232 38.42427
3 Dieter Mietenplage 24 M 79 181 191 24.11404
4 Hella Scheinwerfer 35 W 60 170 200 20.76125
5 Inge Danken 46 W 57 158 148 22.83288
6 Jason Zufall 68 M 96 174 249 31.70828
7 Mitch Mackes 44 M 92 178 220 29.03674
Adipositas
1 Übergewicht
2 Adipositas
3 Normalgewicht
4 Normalgewicht
5 Normalgewicht
6 Adipositas
7 ÜbergewichtAlternativ kann die cut()-Funktion verwendet werden.
# cut-Funktion
chol$Adipositas <- cut(chol$BMI, breaks = c(0, 18.5, 24.5, 30, Inf),
labels = c("Untergewicht", "Normalgewicht",
"Übergewicht", "Adipositas"),
right = FALSE)
# anzeigen
chol Name Alter Geschlecht Gewicht Größe Cholesterol BMI
1 Anna Tomie 18 W 85 179 182 26.52851
2 Bud Zillus 32 M 115 173 232 38.42427
3 Dieter Mietenplage 24 M 79 181 191 24.11404
4 Hella Scheinwerfer 35 W 60 170 200 20.76125
5 Inge Danken 46 W 57 158 148 22.83288
6 Jason Zufall 68 M 96 174 249 31.70828
7 Mitch Mackes 44 M 92 178 220 29.03674
Adipositas
1 Übergewicht
2 Adipositas
3 Normalgewicht
4 Normalgewicht
5 Normalgewicht
6 Adipositas
7 Übergewichtmale erhalten, welcher nur die Daten der Männer beinhaltet.
# subset erzeugen
male <- subset(chol, Geschlecht=="M")
# anzeigen
male Name Alter Geschlecht Gewicht Größe Cholesterol BMI
2 Bud Zillus 32 M 115 173 232 38.42427
3 Dieter Mietenplage 24 M 79 181 191 24.11404
6 Jason Zufall 68 M 96 174 249 31.70828
7 Mitch Mackes 44 M 92 178 220 29.03674
Adipositas
2 Adipositas
3 Normalgewicht
6 Adipositas
7 Übergewichtjgsbook mit allen Abhängigkeiten
# installiere inkl Abhängigkeiten
install.packages("jgsbook", dependencies = TRUE)Der folgende Befehl öffnet einen neuen Tab in RStudio:
# Zeige die enthaltenen Datensätze graphisch an
data(package = "jgsbook")Für die Ausgabe auf der Konsole können wir so vorgehen.
# Zeige die enthaltenen Datensätze auf Konsole
a <- data(package = "jgsbook")
as.data.frame(a$results[, 3:4]) Item
1 Faktorenbogen
2 MarioANOVA
3 Messwiederholung
4 Pflegeberufe
5 epa
6 mma
7 nw (Nachtwachen)
8 nw_labelled (nw)
9 ordinalSample (OrdinalSample)
10 pf8
Title
1 Datatable of the Faktorenbogen Example for factor analysis
2 Datatable of the SuperMario Example for Friedman-ANOVA
3 Datatable of the Messwiederholung Example for ANOVA
4 Matrix of Pflegeberufe by Isfort et al. 2018
5 Datatable of the epa Example
6 Dataset of a work sampling study
7 Dataset of the German Nachtwachen study with labelled variables
8 Dataset of the German Nachtwachen study with labelled variables
9 Datatable of an Ordinal Sample
10 Dataset of the PF8 example.pf8 aus dem jgsbook in das Objekt df. Welche Variablen sind im Datensatz enthalten?
df <- jgsbook::pf8
# anzeigen
str(df)'data.frame': 731 obs. of 16 variables:
$ Standort : Factor w/ 5 levels "Rheine","Münster",..: 2 2 2 2 2 2 2 2 2 2 ...
$ Alter : int 18 67 60 61 24 21 59 56 82 52 ...
$ Geschlecht : Factor w/ 3 levels "männlich","weiblich",..: 2 2 2 1 1 2 2 2 1 2 ...
$ Größe : int 172 165 175 182 173 177 168 156 184 166 ...
$ Gewicht : num 69 67 NA 90 68 60 80 60 NA 60 ...
$ Bildung : Factor w/ 7 levels "keinen","Hauptschule",..: 6 3 7 3 6 6 3 4 3 5 ...
$ Beruf : Factor w/ 104 levels ""," Produktionsleiter",..: 46 81 22 13 93 93 6 69 49 49 ...
$ Familienstand : Factor w/ 6 levels "ledig","Partnerschaft",..: 2 4 2 1 1 2 3 4 3 3 ...
$ Kinder : int 0 0 0 0 0 0 0 2 0 1 ...
$ Wohnort : Factor w/ 2 levels "städtisch","ländlich": 2 2 1 2 1 1 2 1 1 1 ...
$ Rauchen : Factor w/ 2 levels "nein","ja": 1 1 1 1 1 2 1 2 1 1 ...
$ SportHäufig : num NA 2 2 4 4 1 2 1 1 2 ...
$ SportMinuten : num NA 60 45 120 60 60 45 90 NA 45 ...
$ SportWie : Factor w/ 3 levels "Allein","Gruppe",..: 1 2 3 1 1 2 2 3 2 1 ...
$ SportWarum : Factor w/ 8 levels "0","Vorbeugung",..: 6 2 2 4 3 4 2 2 4 2 ...
$ LebenZufrieden: num 5 7 7 2 9 8 5 8 10 8 ...jgsbook-Paket auf.
help(package = "jgsbook")freqTable() aus dem Paket jgsbook auf die Variable df$Kinder an, ohne das Paket vorher per library() zu aktivieren.
# Funktion aufrufen ohne Paket zu laden
jgsbook::freqTable(df$Kinder) Wert Haeufig Hkum Relativ Rkum
1 0 563 563 77.02 77.02
2 1 81 644 11.08 88.10
3 2 60 704 8.21 96.31
4 3 21 725 2.87 99.18
5 4 1 726 0.14 99.32
6 5 1 727 0.14 99.46Datentabelle.txt
# Lese Daten ein
a <- read.table("https://www.produnis.de/trainingslager/data/Datentabelle.txt", header=TRUE)# Datenklassen anschauen
str(a)'data.frame': 10 obs. of 4 variables:
$ Geschlecht: chr "m" "w" "w" "m" ...
$ Alter : int 28 18 25 29 21 19 27 26 31 22
$ Gewicht : int 80 55 74 101 84 74 65 56 88 78
$ Groesse : int 170 174 183 190 185 178 169 163 189 184# Geschlecht anpassen
a$Geschlecht <- factor(a$Geschlecht)
# anschaeun
str(a)'data.frame': 10 obs. of 4 variables:
$ Geschlecht: Factor w/ 2 levels "m","w": 1 2 2 1 1 2 2 2 1 1
$ Alter : int 28 18 25 29 21 19 27 26 31 22
$ Gewicht : int 80 55 74 101 84 74 65 56 88 78
$ Groesse : int 170 174 183 190 185 178 169 163 189 184anwesenheitnoten.csv
In der Datei werden Dezimalstellen mit “,” und Feldtrenner mit “;” angegeben. Entsprechend lautet der Aufruf von read.table():
# Lese Daten ein
b <- read.table("https://www.produnis.de/trainingslager/data/anwesenheitnoten.csv",
header=TRUE, dec=",", sep=";")# Datenklassen anschauen
str(b)'data.frame': 27 obs. of 2 variables:
$ Anwesenheit: num 90.9 40.9 100 81.8 0 100 27.3 27.3 54.5 54.5 ...
$ Note : num 1.7 5 1 NA 5 2 3.7 5 3.3 NA ...Alle Datenklassen sind korrekt (numerisch).
Testdatumdaten.xlsx
# Lese Daten ein
d <- openxlsx::read.xlsx("https://www.produnis.de/trainingslager/data/Testdatumdaten.xlsx")# Datenklassen anschauen
str(d)tibble [38 × 4] (S3: tbl_df/tbl/data.frame)
$ Vornme : chr [1:38] "Anima" "Annika" "Farhad" "Michèle" ...
$ Geschlecht : chr [1:38] "weiblich" "weiblich" "männlich" "weiblich" ...
$ Geburtstag : chr [1:38] "25.02.2001" "19.10.1995" "10.11.1999" "23.08.1993" ...
$ Lieblingsfarbe: chr [1:38] "blau" "grün" "gelb" "blau" ...# Datenklassen anpassen
d$Vornme <- factor(d$Vornme)
d$Geschlecht <- factor(d$Geschlecht)
d$Lieblingsfarbe <- factor(d$Lieblingsfarbe)
# Zeitformat
d$Geburtstag <- lubridate::dmy(d$Geburtstag, tz="CET")
# anschauen
str(d)tibble [38 × 4] (S3: tbl_df/tbl/data.frame)
$ Vornme : Factor w/ 38 levels "Alexander","Anima",..: 2 4 13 30 38 35 10 3 5 15 ...
$ Geschlecht : Factor w/ 2 levels "männlich","weiblich": 2 2 1 2 2 2 2 2 2 1 ...
$ Geburtstag : POSIXct[1:38], format: "2001-02-25" "1995-10-19" ...
$ Lieblingsfarbe: Factor w/ 4 levels "blau","gelb",..: 1 3 2 1 3 3 1 4 4 2 ...v <- 1:10median(v)[1] 5.5mean(v)[1] 5.5v <- c(v, 45881)
# anschauen
v [1] 1 2 3 4 5 6 7 8 9 10 45881median(v)[1] 6mean(v)[1] 4176Es fällt auf, dass der Mittelwert viel stärker von dem neuen Wert (45881) beinflusst wird als der Median.
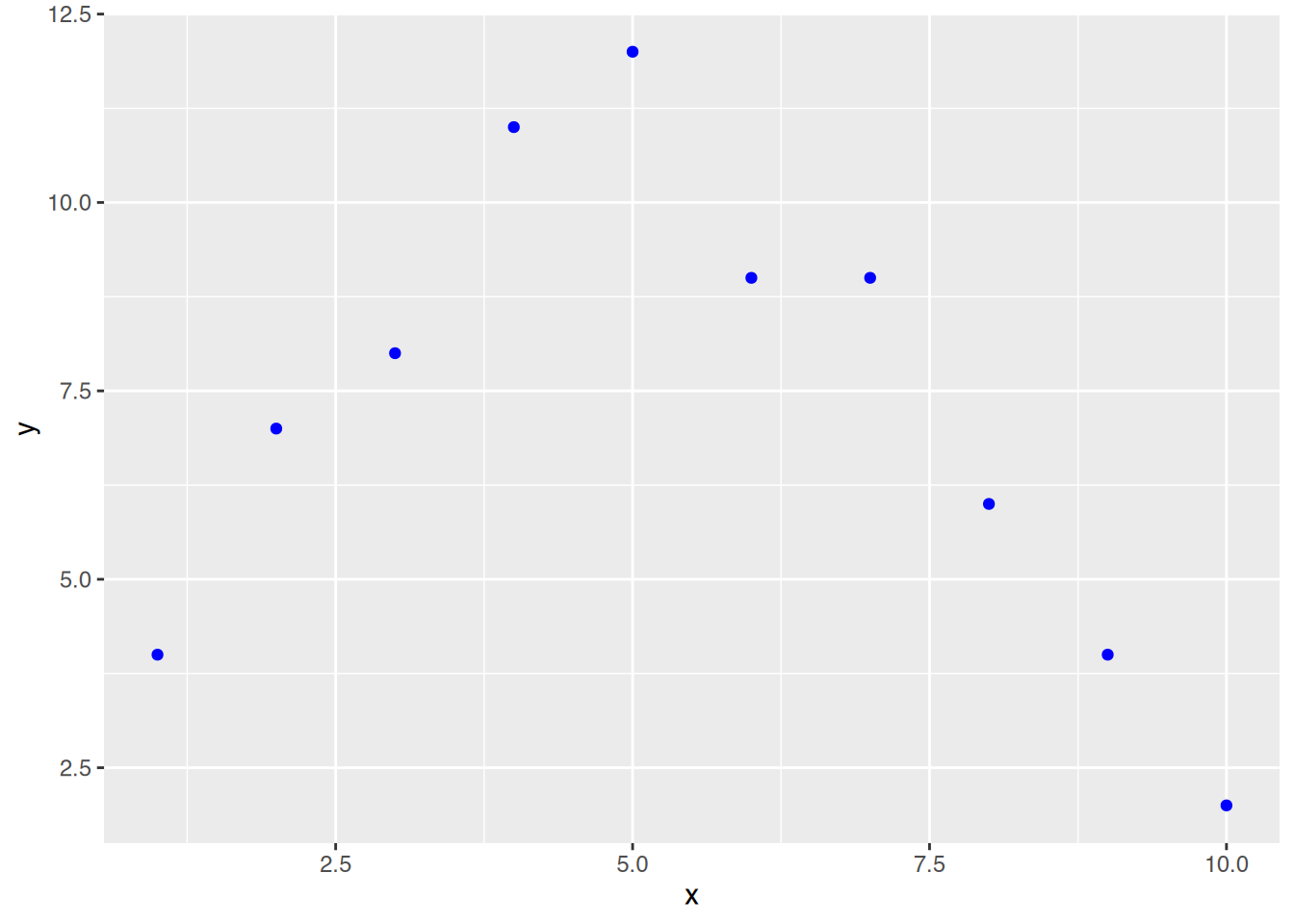
df <- data.frame(x = c(1:10),
y = c(4, 7, 8, 11, 12, 9, 9, 6, 4, 2))# Rbase
plot(df, col="blue")
# ggplot
library(ggplot2)
ggplot(df, aes(x=x, y=y)) +
geom_point(color="blue")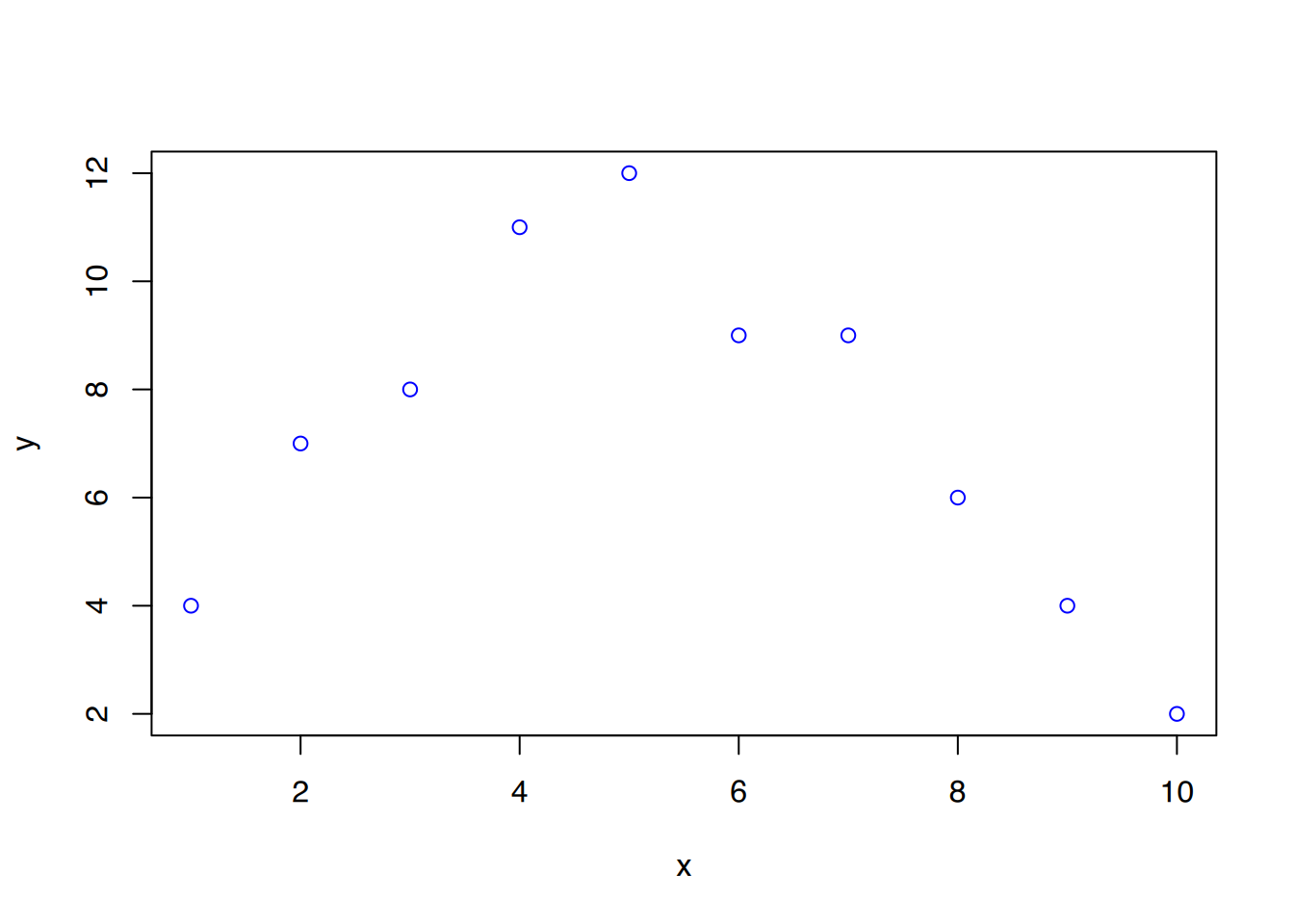
# R-base
barplot(df$y, names.arg = df$x,
col="skyblue",
horiz = TRUE)
# ggplot
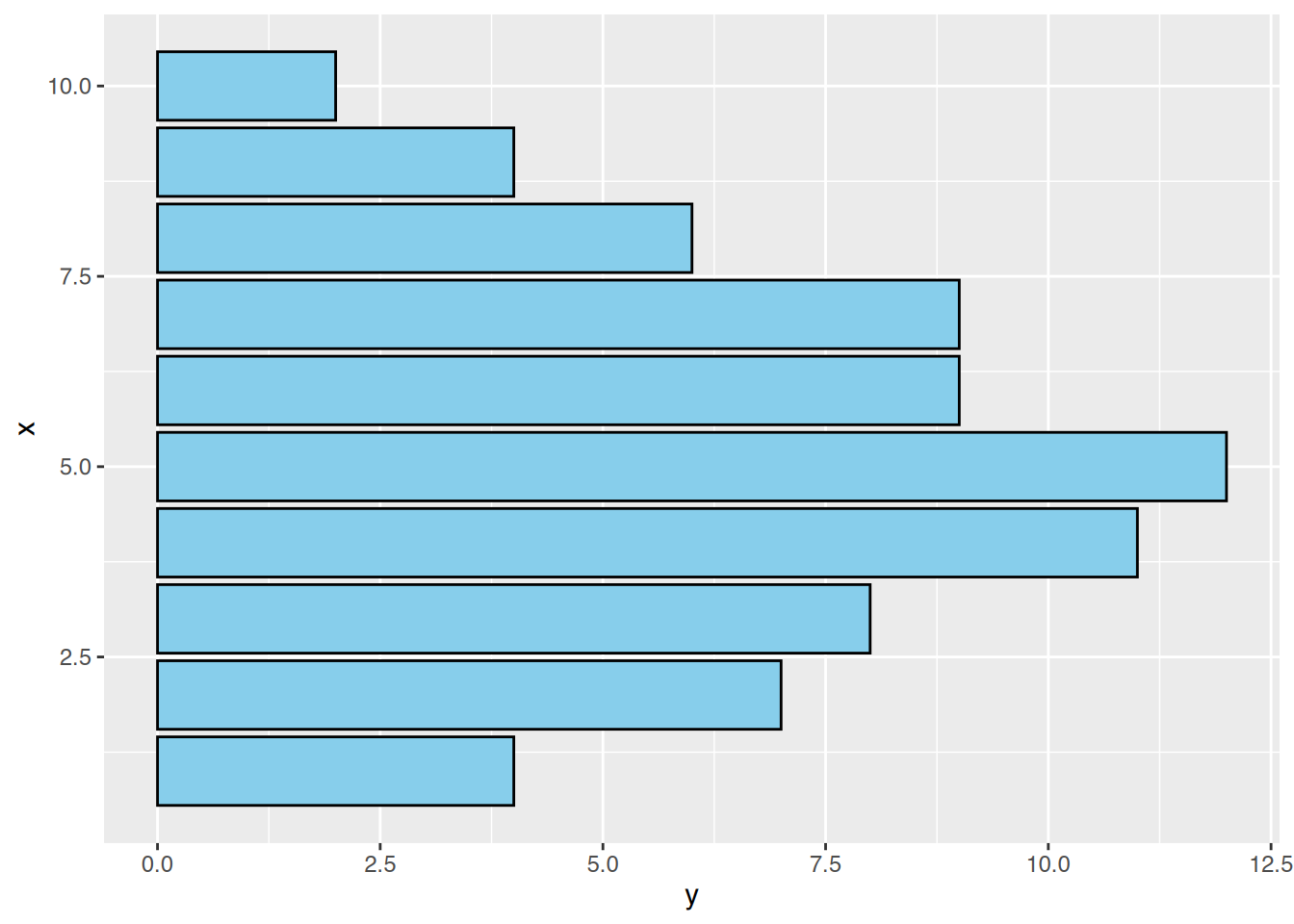
ggplot(df, aes(x=x, y=y)) +
geom_bar(stat="identity",
fill="skyblue", col="black") +
coord_flip()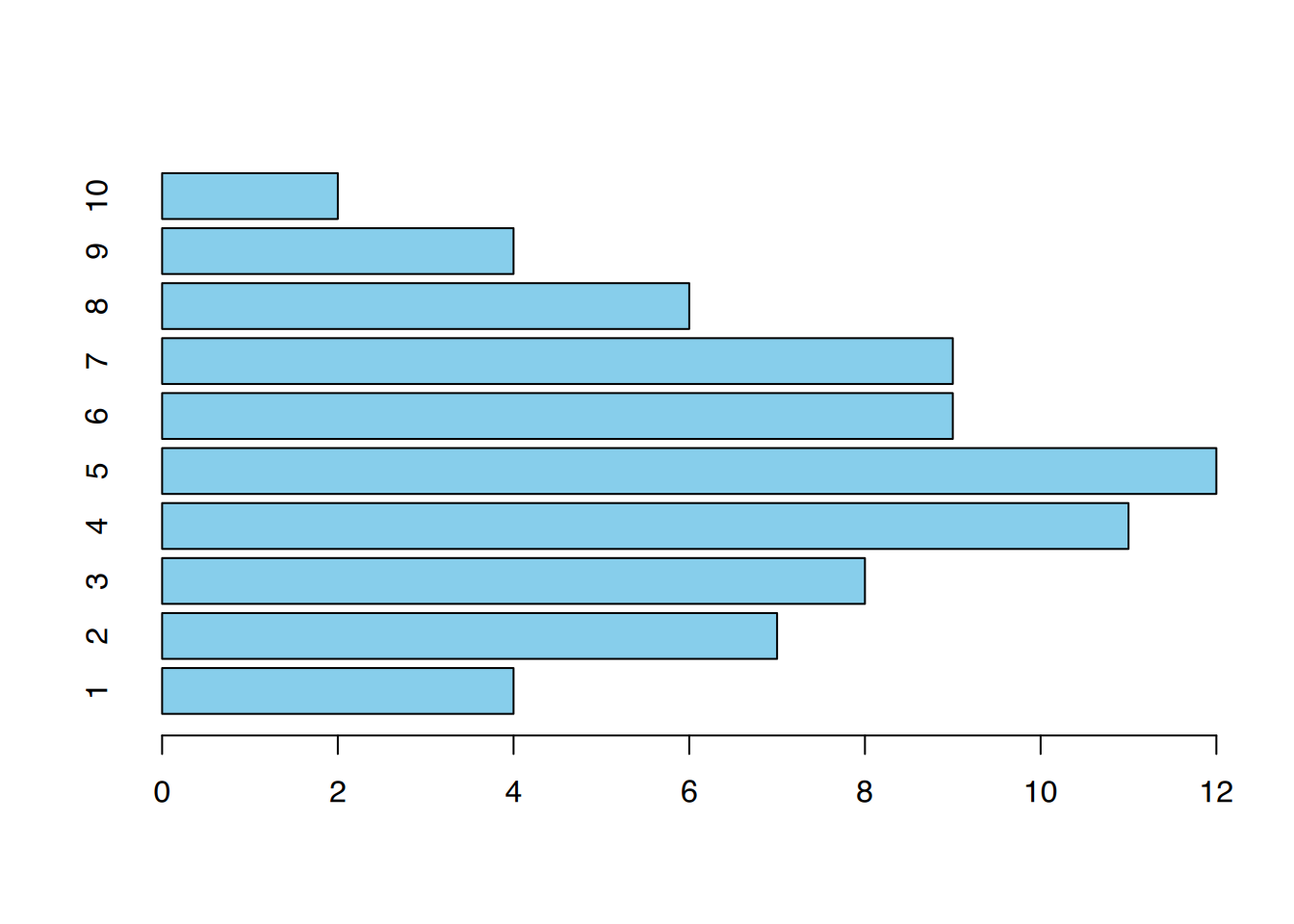
taylor_swift_spotify2024.csv in Ihre R-Session. Nennen Sie Ihr Datenframe dabei ts.
ts <- read.csv("https://www.produnis.de/trainingslager/data/taylor_swift_spotify2024.csv",
sep=",", header=TRUE)str() und summary() einen Überblick über die enthaltenen Daten.
str(ts)'data.frame': 582 obs. of 18 variables:
$ X : int 0 1 2 3 4 5 6 7 8 9 ...
$ name : chr "Fortnight (feat. Post Malone)" "The Tortured Poets Department" "My Boy Only Breaks His Favorite Toys" "Down Bad" ...
$ album : chr "THE TORTURED POETS DEPARTMENT: THE ANTHOLOGY" "THE TORTURED POETS DEPARTMENT: THE ANTHOLOGY" "THE TORTURED POETS DEPARTMENT: THE ANTHOLOGY" "THE TORTURED POETS DEPARTMENT: THE ANTHOLOGY" ...
$ release_date : chr "2024-04-19" "2024-04-19" "2024-04-19" "2024-04-19" ...
$ track_number : int 1 2 3 4 5 6 7 8 9 10 ...
$ id : chr "6dODwocEuGzHAavXqTbwHv" "4PdLaGZubp4lghChqp8erB" "7uGYWMwRy24dm7RUDDhUlD" "1kbEbBdEgQdQeLXCJh28pJ" ...
$ uri : chr "spotify:track:6dODwocEuGzHAavXqTbwHv" "spotify:track:4PdLaGZubp4lghChqp8erB" "spotify:track:7uGYWMwRy24dm7RUDDhUlD" "spotify:track:1kbEbBdEgQdQeLXCJh28pJ" ...
$ acousticness : num 0.502 0.0483 0.137 0.56 0.73 0.384 0.624 0.178 0.607 0.315 ...
$ danceability : num 0.504 0.604 0.596 0.541 0.423 0.521 0.33 0.533 0.626 0.606 ...
$ energy : num 0.386 0.428 0.563 0.366 0.533 0.72 0.483 0.573 0.428 0.338 ...
$ instrumentalness: num 1.53e-05 0.00 0.00 1.00e-06 2.64e-03 0.00 0.00 0.00 0.00 0.00 ...
$ liveness : num 0.0961 0.126 0.302 0.0946 0.0816 0.135 0.111 0.309 0.0921 0.106 ...
$ loudness : num -10.98 -8.44 -7.36 -10.41 -11.39 ...
$ speechiness : num 0.0308 0.0255 0.0269 0.0748 0.322 0.104 0.0399 0.138 0.0261 0.048 ...
$ tempo : num 192 110.3 97.1 159.7 160.2 ...
$ valence : num 0.281 0.292 0.481 0.168 0.248 0.438 0.34 0.398 0.487 0.238 ...
$ popularity : int 82 79 80 82 80 81 78 79 82 81 ...
$ duration_ms : int 228965 293048 203801 261228 262974 340428 210789 215463 254365 334084 ...summary(ts) X name album release_date
Min. : 0.0 Length:582 Length:582 Length:582
1st Qu.:145.2 Class :character Class :character Class :character
Median :290.5 Mode :character Mode :character Mode :character
Mean :290.5
3rd Qu.:435.8
Max. :581.0
track_number id uri acousticness
Min. : 1.00 Length:582 Length:582 Min. :0.000182
1st Qu.: 5.00 Class :character Class :character 1st Qu.:0.037325
Median :10.00 Mode :character Mode :character Median :0.184500
Mean :11.42 Mean :0.333185
3rd Qu.:15.00 3rd Qu.:0.660000
Max. :46.00 Max. :0.971000
danceability energy instrumentalness liveness
Min. :0.1750 Min. :0.1180 Min. :0.0000000 Min. :0.03350
1st Qu.:0.5150 1st Qu.:0.4180 1st Qu.:0.0000000 1st Qu.:0.09652
Median :0.5935 Median :0.5710 Median :0.0000019 Median :0.11450
Mean :0.5808 Mean :0.5658 Mean :0.0033933 Mean :0.16113
3rd Qu.:0.6530 3rd Qu.:0.7190 3rd Qu.:0.0000581 3rd Qu.:0.16100
Max. :0.8970 Max. :0.9480 Max. :0.3330000 Max. :0.93100
loudness speechiness tempo valence
Min. :-17.932 Min. :0.02310 Min. : 68.10 Min. :0.0384
1st Qu.: -9.401 1st Qu.:0.03030 1st Qu.: 96.89 1st Qu.:0.2300
Median : -7.353 Median :0.03760 Median :119.05 Median :0.3740
Mean : -7.662 Mean :0.05648 Mean :122.40 Mean :0.3910
3rd Qu.: -5.495 3rd Qu.:0.05480 3rd Qu.:143.94 3rd Qu.:0.5225
Max. : -1.927 Max. :0.91200 Max. :208.92 Max. :0.9430
popularity duration_ms
Min. : 0.00 Min. : 83253
1st Qu.:45.00 1st Qu.:211823
Median :62.00 Median :235433
Mean :57.86 Mean :240011
3rd Qu.:70.00 3rd Qu.:260820
Max. :93.00 Max. :613026 Mindestens album kann als Factor angesehen werden. Streggenommen aber auch name, id und uri.
ts$name <- factor(ts$name)
ts$album <- factor(ts$album)
ts$release_date <- as.Date(ts$release_date)
ts$id <- factor(ts$id)
ts$uri <- factor(ts$uri)
summary(ts) X name
Min. : 0.0 The Story Of Us : 4
1st Qu.:145.2 22 : 3
Median :290.5 All Too Well : 3
Mean :290.5 Anti-Hero : 3
3rd Qu.:435.8 august : 3
Max. :581.0 Back To December: 3
(Other) :563
album
reputation Stadium Tour Surprise Song Playlist : 46
folklore: the long pond studio sessions (from the Disney+ special) [deluxe edition]: 34
THE TORTURED POETS DEPARTMENT: THE ANTHOLOGY : 31
Red (Taylor's Version) : 30
Fearless (Taylor's Version) : 26
Midnights (The Til Dawn Edition) : 23
(Other) :392
release_date track_number id
Min. :2006-10-24 Min. : 1.00 00vJzaoxM3Eja1doBUhX0P: 1
1st Qu.:2012-10-22 1st Qu.: 5.00 0108kcWLnn2HlH2kedi1gn: 1
Median :2020-08-18 Median :10.00 01K4zKU104LyJ8gMb7227B: 1
Mean :2018-04-17 Mean :11.42 01QdEx6kFr78ZejhQtWR5m: 1
3rd Qu.:2022-10-22 3rd Qu.:15.00 02Zkkf2zMkwRGQjZ7T4p8f: 1
Max. :2024-04-19 Max. :46.00 045ZeOHPIzhxxsm8bq5kyE: 1
(Other) :576
uri acousticness danceability
spotify:track:00vJzaoxM3Eja1doBUhX0P: 1 Min. :0.000182 Min. :0.1750
spotify:track:0108kcWLnn2HlH2kedi1gn: 1 1st Qu.:0.037325 1st Qu.:0.5150
spotify:track:01K4zKU104LyJ8gMb7227B: 1 Median :0.184500 Median :0.5935
spotify:track:01QdEx6kFr78ZejhQtWR5m: 1 Mean :0.333185 Mean :0.5808
spotify:track:02Zkkf2zMkwRGQjZ7T4p8f: 1 3rd Qu.:0.660000 3rd Qu.:0.6530
spotify:track:045ZeOHPIzhxxsm8bq5kyE: 1 Max. :0.971000 Max. :0.8970
(Other) :576
energy instrumentalness liveness loudness
Min. :0.1180 Min. :0.0000000 Min. :0.03350 Min. :-17.932
1st Qu.:0.4180 1st Qu.:0.0000000 1st Qu.:0.09652 1st Qu.: -9.401
Median :0.5710 Median :0.0000019 Median :0.11450 Median : -7.353
Mean :0.5658 Mean :0.0033933 Mean :0.16113 Mean : -7.662
3rd Qu.:0.7190 3rd Qu.:0.0000581 3rd Qu.:0.16100 3rd Qu.: -5.495
Max. :0.9480 Max. :0.3330000 Max. :0.93100 Max. : -1.927
speechiness tempo valence popularity
Min. :0.02310 Min. : 68.10 Min. :0.0384 Min. : 0.00
1st Qu.:0.03030 1st Qu.: 96.89 1st Qu.:0.2300 1st Qu.:45.00
Median :0.03760 Median :119.05 Median :0.3740 Median :62.00
Mean :0.05648 Mean :122.40 Mean :0.3910 Mean :57.86
3rd Qu.:0.05480 3rd Qu.:143.94 3rd Qu.:0.5225 3rd Qu.:70.00
Max. :0.91200 Max. :208.92 Max. :0.9430 Max. :93.00
duration_ms
Min. : 83253
1st Qu.:211823
Median :235433
Mean :240011
3rd Qu.:260820
Max. :613026
# Mittelwert
mean(ts$duration_ms)/1000[1] 240.0112# Median
median(ts$duration_ms)/1000[1] 235.433# Umrechnen in Sekunden
songlänge <- ts$duration_ms / 1000
# Rbase
hist(songlänge, col="pink",
xlab="Länge in Sekunden",
ylab="Anzahl der Songs",
main= "Talyor Swift Songs 2024")
# ggplot
library(ggplot2)
songs <- as.data.frame(songlänge)
ggplot(songs, aes(x=songlänge)) +
geom_histogram(fill="pink", col="black",
bins=10) +
labs(title = "Taylor Swift Songs 2024",
x = "Länge in Sekunden",
y = "Anzahl der Songs")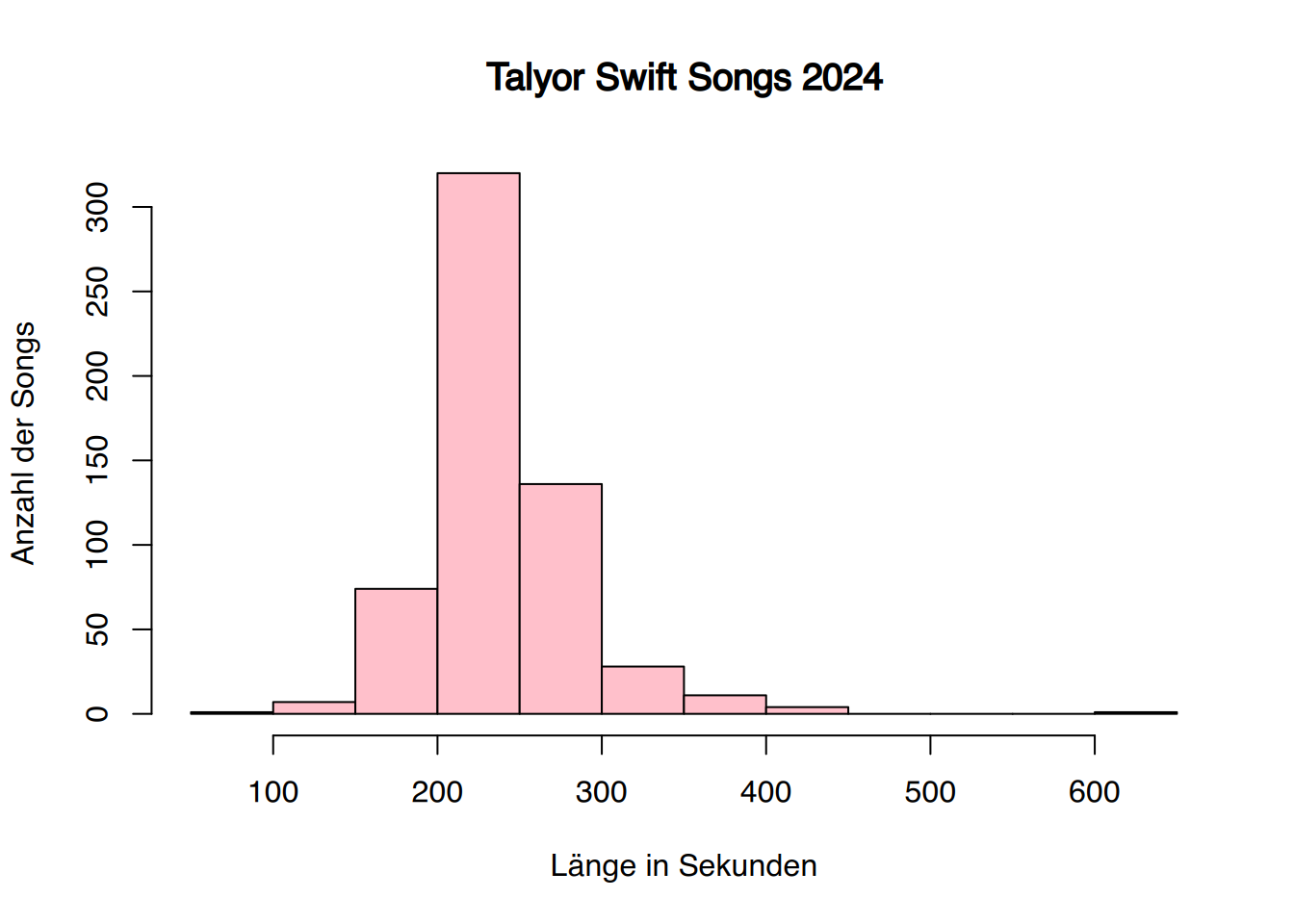
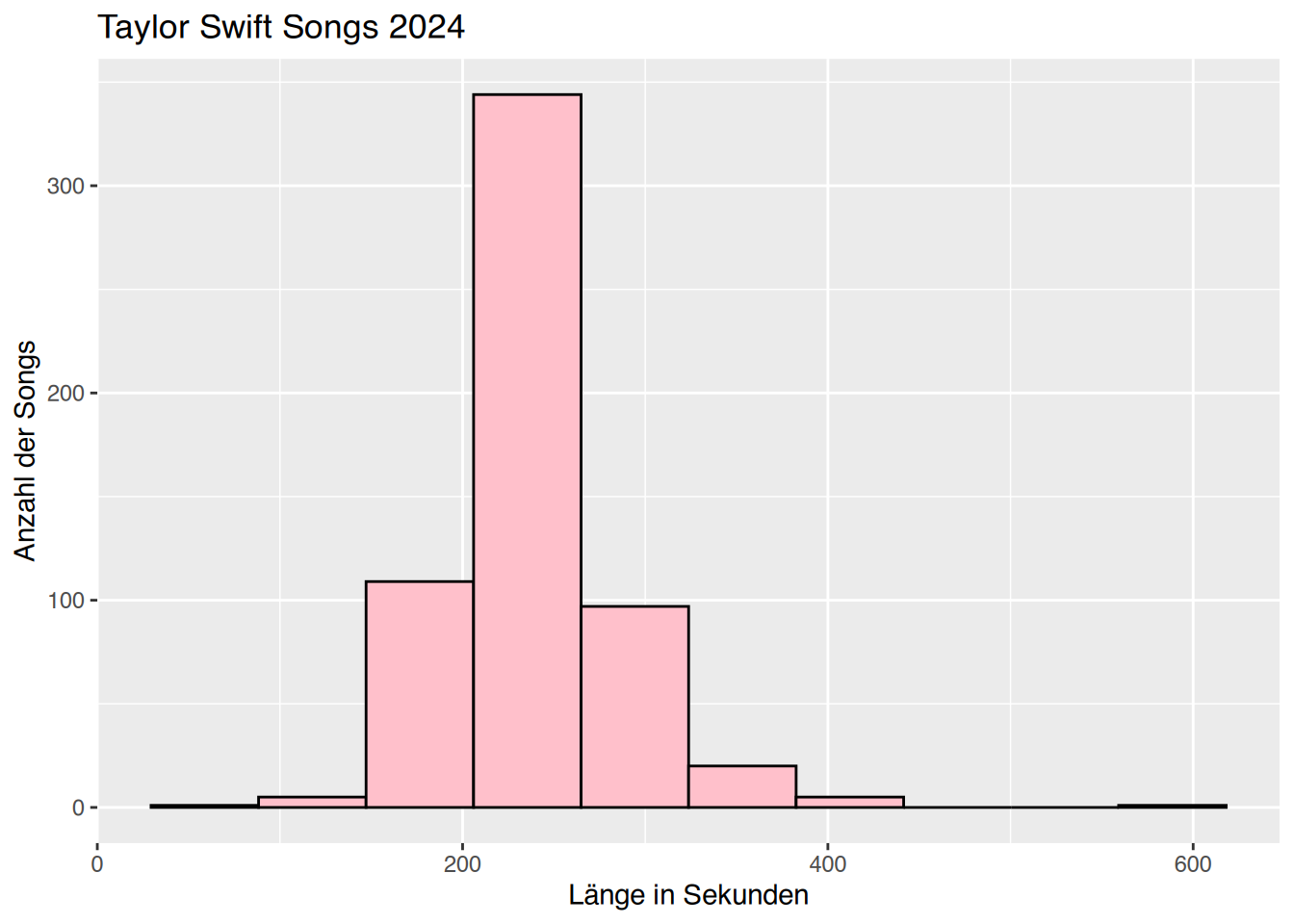
Neben min() und max() können mittels summary() die Werte über Min. und Max. abgelesen werden.
## populärste
ts[ts$popularity==93, ]
...1 name album release_date track_number id uri acousticness1 324 Cruel… Lover 2019-08-23 2 1Bxf… spot… 0.117
# oder
ts$name[ts$popularity==93][1] Cruel Summer
363 Levels: ...Ready For It? ... You're On Your Own, Kid# oder
ts$name[ts$popularity==max(ts$popularity)][1] Cruel Summer
363 Levels: ...Ready For It? ... You're On Your Own, Kid## längste
ts[ts$duration_ms==613026, ]
...1 name album release_date track_number id uri acousticness1 197 All Too… Red … 2021-11-12 30 5enx… spot… 0.274
# oder
ts$name[ts$duration_ms==613026][1] All Too Well (10 Minute Version) (Taylor's Version) (From The Vault)
363 Levels: ...Ready For It? ... You're On Your Own, Kid# oder
ts$name[ts$duration_ms==max(ts$duration_ms)][1] All Too Well (10 Minute Version) (Taylor's Version) (From The Vault)
363 Levels: ...Ready For It? ... You're On Your Own, Kid## langsamste
ts[ts$tempo==68.097, ]
...1 name album release_date track_number id uri acousticness1 281 this is … folk… 2020-11-25 9 1gaL… spot… 0.928
# oder
ts$name[ts$tempo==68.097][1] this is me trying - the long pond studio sessions
363 Levels: ...Ready For It? ... You're On Your Own, Kid# oder
ts$name[ts$tempo==min(ts$tempo)][1] this is me trying - the long pond studio sessions
363 Levels: ...Ready For It? ... You're On Your Own, Kid# die meisten Songs
sort(table(ts$album), decreasing=TRUE)[1]reputation Stadium Tour Surprise Song Playlist
46 # die wenigsten Songs
sort(table(ts$album))[1]Live From Clear Channel Stripped 2008
8 Um die Datumsangaben auf der X-Achse in korrekten Abständen anzuzeigen erstellen wir zunächst ein Datenframe der Häufigkeitstabelle von ts$release_date. Anschließend werden die Datumsangaben per as.Date() in die richtige Klasse überführt.
# erstelle Datenframe der Häufigkeitstabelle
df <- as.data.frame(table(ts$release_date))
# anzeigen
df Var1 Freq
1 2006-10-24 15
2 2008-06-28 8
3 2008-11-11 35
4 2010-01-01 22
5 2010-10-25 30
6 2012-10-22 38
7 2014-01-01 32
8 2017-11-09 46
9 2017-11-10 15
10 2019-08-23 18
11 2020-07-24 16
12 2020-08-18 17
13 2020-11-25 34
14 2020-12-11 15
15 2021-01-07 17
16 2021-04-09 26
17 2021-11-12 30
18 2022-10-21 13
19 2022-10-22 20
20 2023-05-26 23
21 2023-07-07 22
22 2023-10-26 21
23 2023-10-27 22
24 2024-04-18 16
25 2024-04-19 31# überführe in Datumsklasse
df$Var1 <- as.Date(df$Var1)# plotte mit Rbase
plot(df, type="b",
col="purple",
ylab="Anzahl Tracks",
xlab="Datum",
main="Taylor Swift Alben")
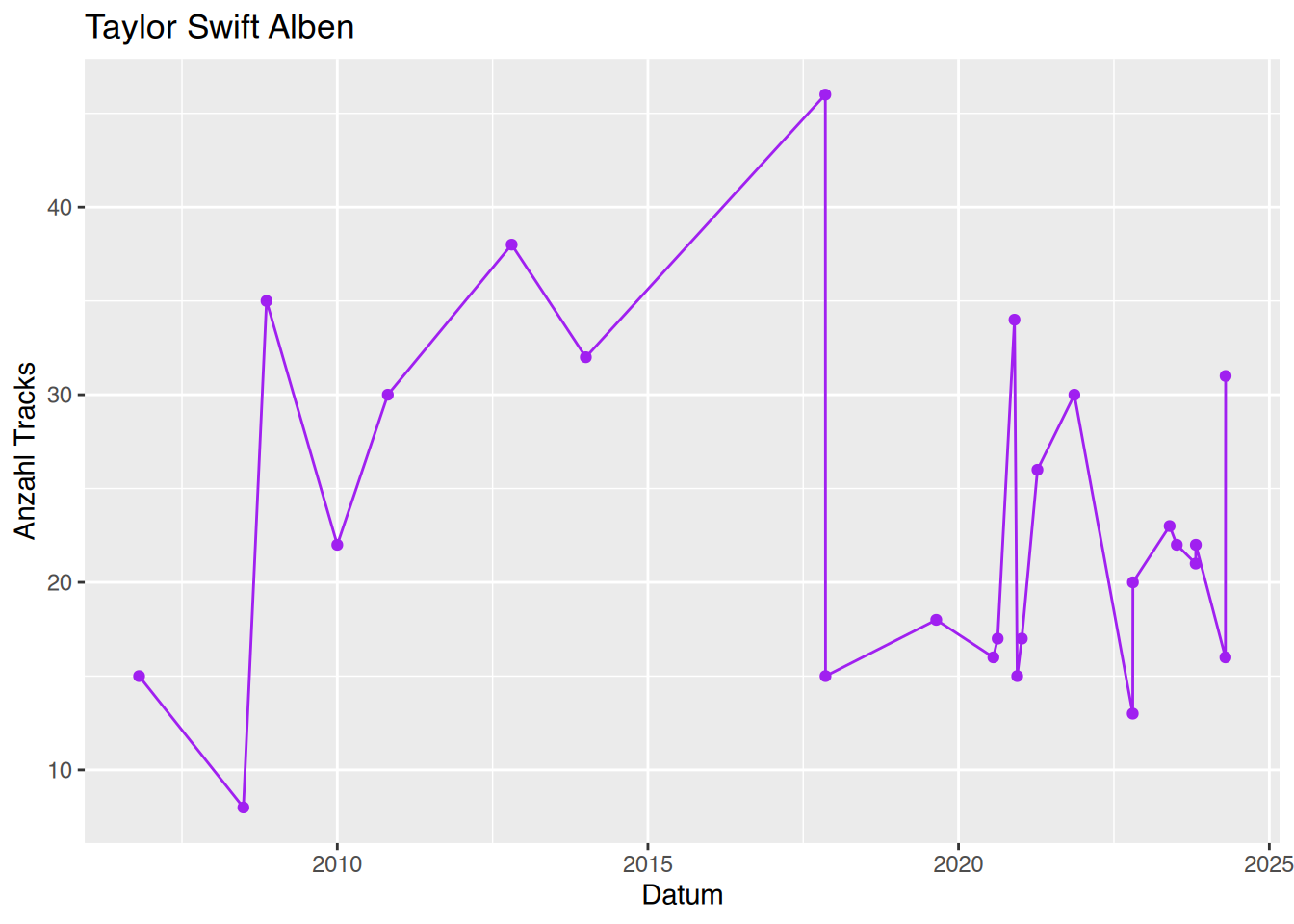
# plott mit ggplot
ggplot(df, aes(x = Var1, y = Freq)) +
geom_line(color = "purple") +
geom_point(color = "purple") +
labs(x = "Datum",
y = "Anzahl Tracks",
title = "Taylor Swift Alben")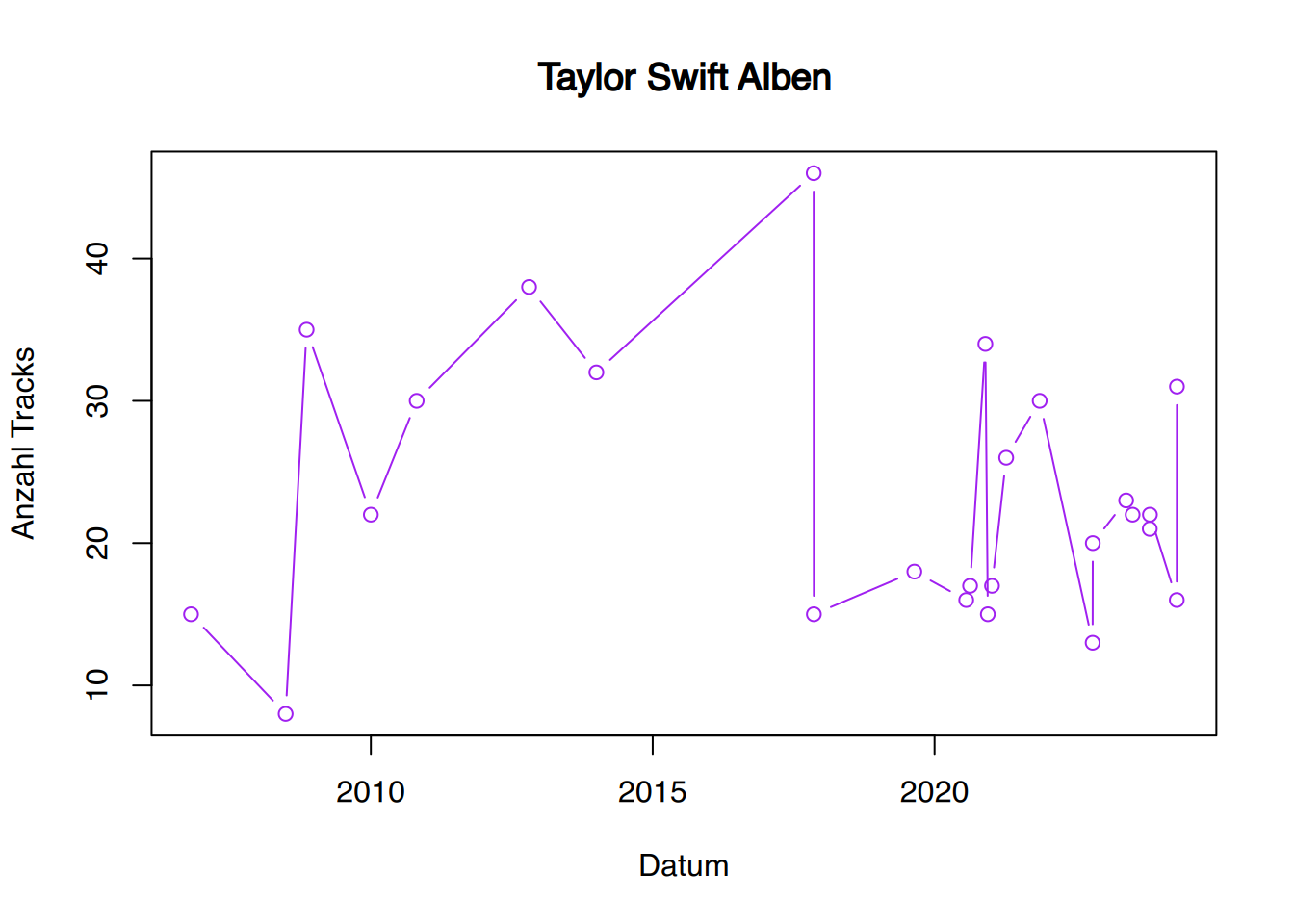
chol.
# gebe die Werte ein
serumchol <- c(4.5, 4.9, 7.3, 5.2, 5.8, 6.2, 5.0, 5.6, 6.4, 7.6,
5.4, 4.4, 6.6, 5.3, 5.7, 4.7, 8.2, 6.7, 4.8, 5.9)
# erstelle Datenframe
df <- data.frame(chol= serumchol)Die Klassierung erfoglt entweder “von Hand”:
# erstelle Werteklassen in eigener Variablenspalte
df$cholklass[df$chol < 5] <- "4.0-4.9" # alle Werte kleiner 5
df$cholklass[df$chol < 6 & df$chol > 4.9] <- "5.0-5.9" # Werte kleiner 6 und größer 4
df$cholklass[df$chol < 7 & df$chol > 5.9] <- "6.0-6.9" # Werte kleiner 7 und größer 5
df$cholklass[df$chol < 8 & df$chol > 6.9] <- "7.0-7.9" # Werte kleiner 8 und größer 6
df$cholklass[df$chol < 9 & df$chol > 7.9] <- "8.0-8.9" # Werte kleiner 9 und größer 7
df$cholklass <- factor(df$cholklass, ordered=T)
# neue Variable anschauen
df$cholklass [1] 4.0-4.9 4.0-4.9 7.0-7.9 5.0-5.9 5.0-5.9 6.0-6.9 5.0-5.9 5.0-5.9 6.0-6.9
[10] 7.0-7.9 5.0-5.9 4.0-4.9 6.0-6.9 5.0-5.9 5.0-5.9 4.0-4.9 8.0-8.9 6.0-6.9
[19] 4.0-4.9 5.0-5.9
Levels: 4.0-4.9 < 5.0-5.9 < 6.0-6.9 < 7.0-7.9 < 8.0-8.9…oder mittels cut().
df$cholklass2 <- cut(df$chol, breaks=c(4:9),
right=FALSE,
ordered_result = TRUE)
# anzeigen
df chol cholklass cholklass2
1 4.5 4.0-4.9 [4,5)
2 4.9 4.0-4.9 [4,5)
3 7.3 7.0-7.9 [7,8)
4 5.2 5.0-5.9 [5,6)
5 5.8 5.0-5.9 [5,6)
6 6.2 6.0-6.9 [6,7)
7 5.0 5.0-5.9 [5,6)
8 5.6 5.0-5.9 [5,6)
9 6.4 6.0-6.9 [6,7)
10 7.6 7.0-7.9 [7,8)
11 5.4 5.0-5.9 [5,6)
12 4.4 4.0-4.9 [4,5)
13 6.6 6.0-6.9 [6,7)
14 5.3 5.0-5.9 [5,6)
15 5.7 5.0-5.9 [5,6)
16 4.7 4.0-4.9 [4,5)
17 8.2 8.0-8.9 [8,9)
18 6.7 6.0-6.9 [6,7)
19 4.8 4.0-4.9 [4,5)
20 5.9 5.0-5.9 [5,6)# erzeuge eine Häufigkeitstabelle
jgsbook::freqTable(df$cholklass) Wert Haeufig Hkum Relativ Rkum
1 4.0-4.9 5 5 25 25
2 5.0-5.9 8 13 40 65
3 6.0-6.9 4 17 20 85
4 7.0-7.9 2 19 10 95
5 8.0-8.9 1 20 5 100# allgemein:
summary(df$chol) Min. 1st Qu. Median Mean 3rd Qu. Max.
4.400 4.975 5.650 5.810 6.450 8.200 # speziell
psych::describe(df$chol,
IQR=TRUE,
skew=FALSE,
quant = c(.10, 0.25, 0.75, .90)
) vars n mean sd median min max range se IQR Q0.1 Q0.25 Q0.75 Q0.9
X1 1 20 5.81 1.06 5.65 4.4 8.2 3.8 0.24 1.48 4.68 4.97 6.45 7.33# Was fehlt noch:
# Varianz
var(df$chol)[1] 1.124105# und Median
median(df$chol)[1] 5.65Wir können “von Hand” rechnen…
mittelwert <- mean(serumchol)
# SQA
sum((serumchol - mittelwert)^2)[1] 21.358… oder wir machen uns die Tatsache zu nutze, dass die Formel für die Varianz \(\frac{\sum(x_{i}-\bar{x})^2}{n-1}\) lautet. Folglich müssen wir die Varianz mit \((n-1)\) multiplizieren, und erhalten so die SQA.
var(serumchol) * (length(serumchol)-1)[1] 21.358boxplot(df$chol)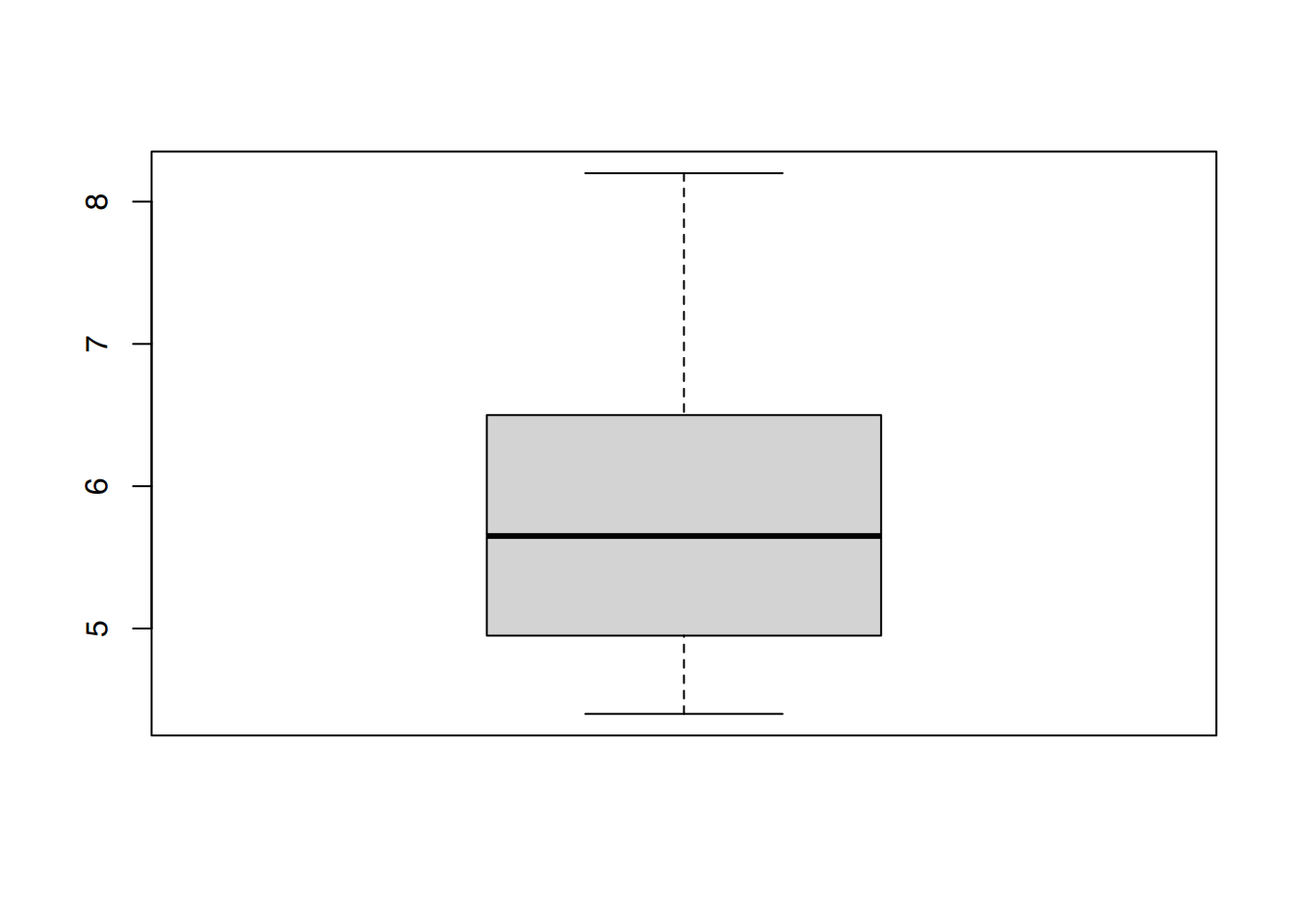
# hübscher
boxplot(df$chol, main="Serumcholesterinspiegel in mmol/l",
col="cyan", border="blue")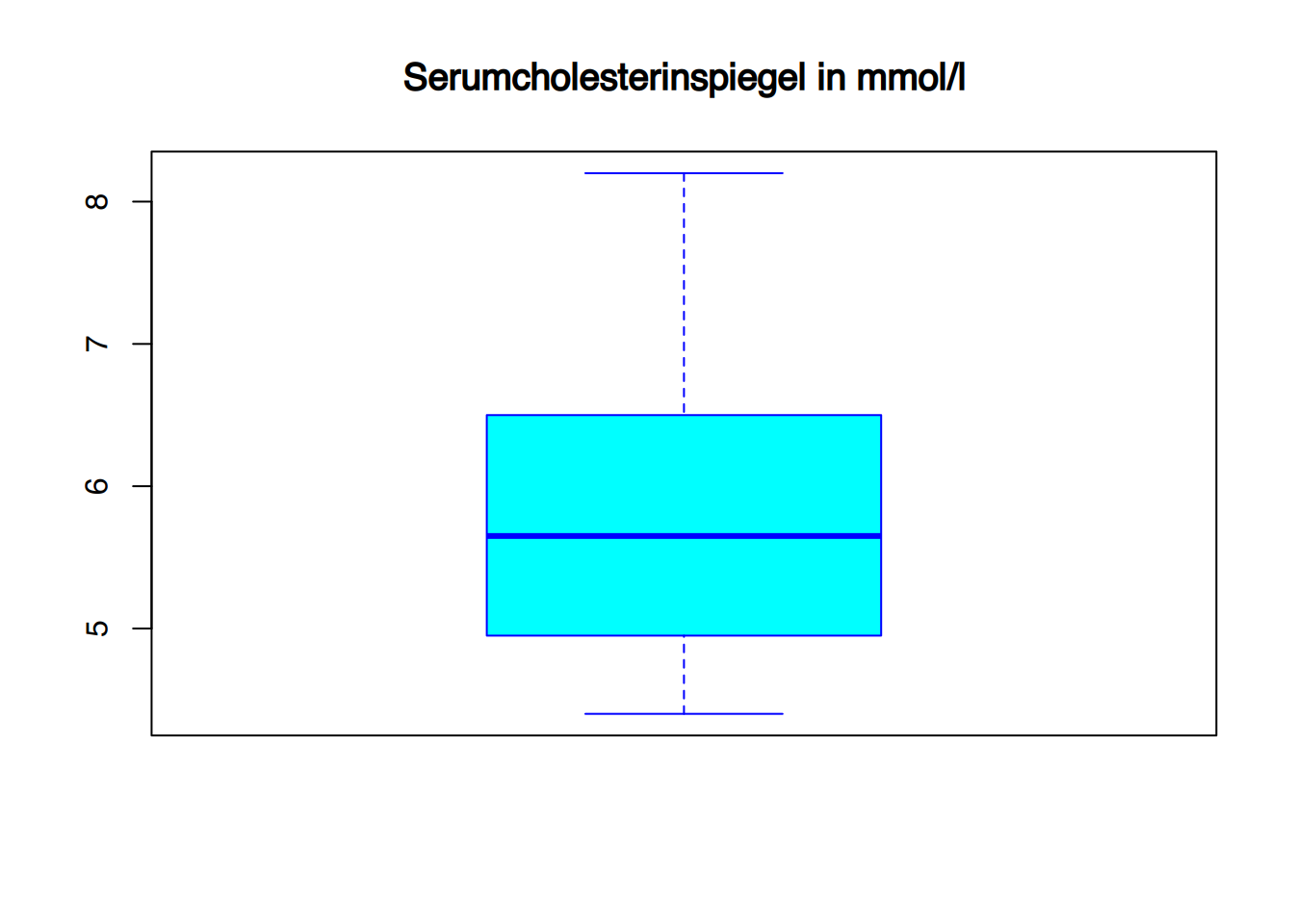
# Histogram
hist(df$chol)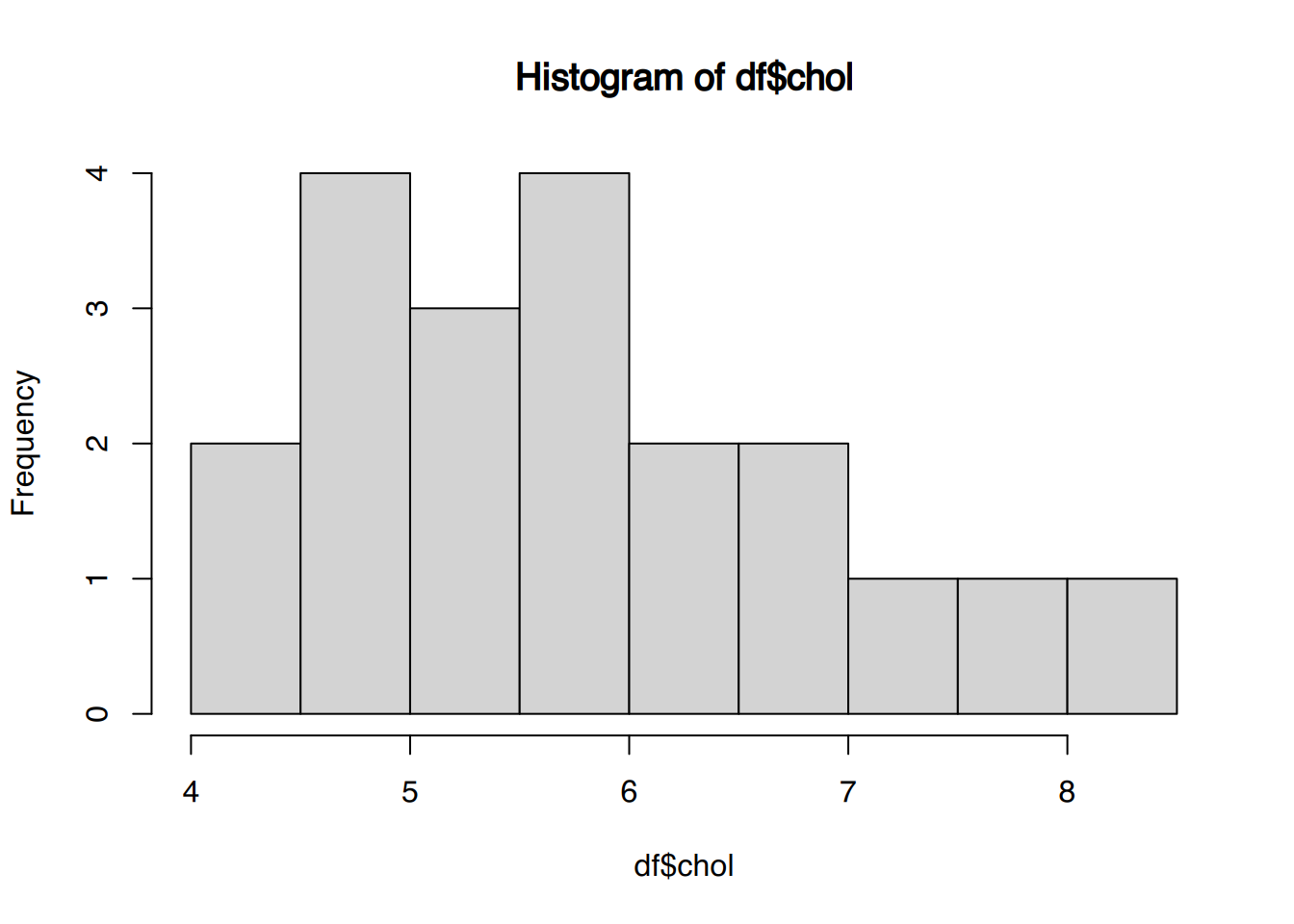
# Histogram mit 5 "breaks und etwas hübscher
hist(df$chol, breaks=5,
col="skyblue2",
main="Histogram",
xlab="Serumcholesterin",
ylab="Häufigkeit")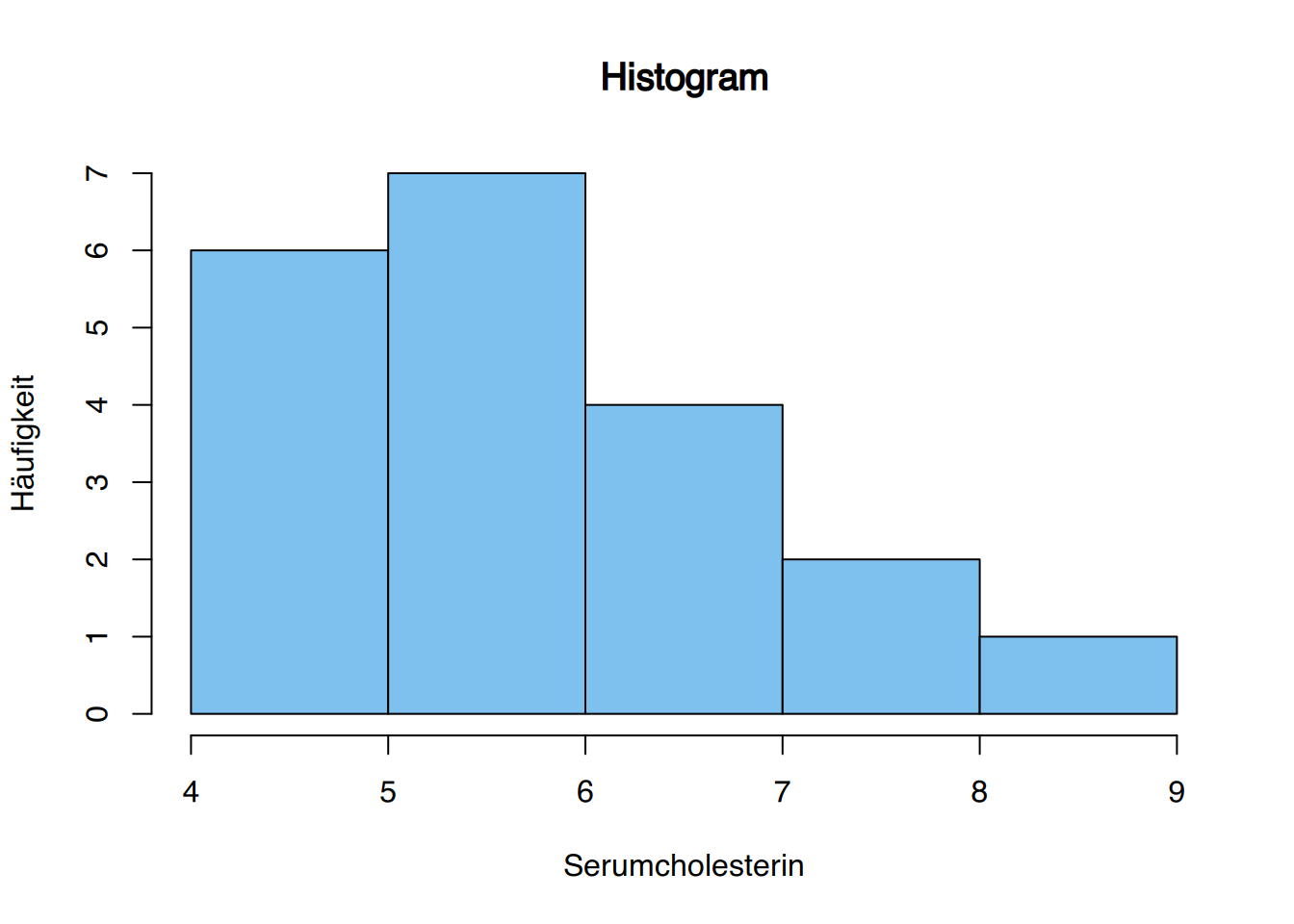
# "Schiefe" berechnen
psych::skew(df$chol)[1] 0.6286707# "Spitzigkeit" berechnen
psych::kurtosi(df$chol)[1] -0.6340307Die Skewness ist positiv, d.h. die Verteilung ist linksgipflig (aka rechtschief).
Die Kurtosis von -0,63 zeigt an, dass die Daten flacher und breiter als eine Normalverteilung sind.
alter <- c(4, 7 ,8, 9, 11, 12, 13, 14, 15, 16, 16, 20, 20,
22, 25, 26, 26, 28, 29, 34)
geschlecht <- c(1, 2, 2, 2, 1, 1, 2, 2, 2, 1, 1, 2, 2, 2, 1, 0, 2, 1, 2, 0)
# Erzeuge Datenframe
df <- data.frame(alter, geschlecht)Geschlecht” um
# wandle "Geschlecht"-Einträge um
df$geschlecht[df$geschlecht == "0"] <- "divers"
df$geschlecht[df$geschlecht == "1"] <- "männlich"
df$geschlecht[df$geschlecht == "2"] <- "weiblich"
# wandle in factor() um
df$geschlecht <- factor(df$geschlecht)Die Altersklassierung erfolgt entweder “von Hand”…
# klassiere die Daten in eigener Spaltenvariable
df$alterk[df$alter < 6] <- "0-5" # alle Werte kleiner 6
df$alterk[df$alter < 11 & df$alter > 5] <- "6-10" # Werte kleiner 11 und größer 5
df$alterk[df$alter < 16 & df$alter > 10] <- "11-15" # Werte kleiner 16 und größer 10
df$alterk[df$alter < 21 & df$alter > 15] <- "16-20" # Werte kleiner 21 und größer 15
df$alterk[df$alter < 26 & df$alter > 20] <- "21-25" # Werte kleiner 26 und größer 20
df$alterk[df$alter < 31 & df$alter > 25] <- "26-30" # Werte kleiner 31 und größer 25
df$alterk[df$alter > 30] <- "31-35" # Werte größer 30 werden zu "31-35"
# ordinaler Faktor der Werteklassen
df$alterk <- factor(df$alterk,
levels=c("0-5", "6-10", "11-15", "16-20", "21-25",
"26-30", "31-35"),
ordered=TRUE)… oder per cut()-Funktion.
df$alterk2 <- cut(df$alter, breaks = seq(0,35,by=5),
ordered=TRUE)
#anzeigen
df alter geschlecht alterk alterk2
1 4 männlich 0-5 (0,5]
2 7 weiblich 6-10 (5,10]
3 8 weiblich 6-10 (5,10]
4 9 weiblich 6-10 (5,10]
5 11 männlich 11-15 (10,15]
6 12 männlich 11-15 (10,15]
7 13 weiblich 11-15 (10,15]
8 14 weiblich 11-15 (10,15]
9 15 weiblich 11-15 (10,15]
10 16 männlich 16-20 (15,20]
11 16 männlich 16-20 (15,20]
12 20 weiblich 16-20 (15,20]
13 20 weiblich 16-20 (15,20]
14 22 weiblich 21-25 (20,25]
15 25 männlich 21-25 (20,25]
16 26 divers 26-30 (25,30]
17 26 weiblich 26-30 (25,30]
18 28 männlich 26-30 (25,30]
19 29 weiblich 26-30 (25,30]
20 34 divers 31-35 (30,35]Alter’.
# allgemein
summary(df$alter) Min. 1st Qu. Median Mean 3rd Qu. Max.
4.00 11.75 16.00 17.75 25.25 34.00 # Minimum
min(df$alter)[1] 4# Perzentile und Quartile
quantile(df$alter, probs = c(0.05, 0.25, 0.75, 0.95)) 5% 25% 75% 95%
6.85 11.75 25.25 29.25 # Perzentile und Quartile
# mit SPSS-Rechenmethode (type=6)
quantile(df$alter, probs = c(0.05, 0.25, 0.75, 0.95), type=6) 5% 25% 75% 95%
4.15 11.25 25.75 33.75 # Median
median(df$alter)[1] 16# ar.Mittel
mean(df$alter)[1] 17.75# Maximum
max(df$alter)[1] 34# Interquartilsabstand
IQR(df$alter)[1] 13.5# Interquartilsabstand
# SPSS-Rechenmethode (type=6)
IQR(df$alter, type=6)[1] 14.5Berechne (fast) alles auf einmal:
# oder einfach
psych::describe(df$alter,
quant = c(0.05, 0.25, 0.75, 0.95),
skew=FALSE,
IQR=TRUE) vars n mean sd median min max range se IQR Q0.05 Q0.25 Q0.75 Q0.95
X1 1 20 17.75 8.33 16 4 34 30 1.86 13.5 6.85 11.75 25.25 29.25Die Funktion hist() kann nur metrische Daten verarbeiten. Daher nehmen wir die Variable “alter” (und nicht “alterk”) und stellen die Abstände auf 5 (Jahre).
# Histogram geht mit R-base hist() nur bei metrischen Daten!!
# Die Werteklassen können per "breaks"-Parameter angegeben werden.
hist(df$alter)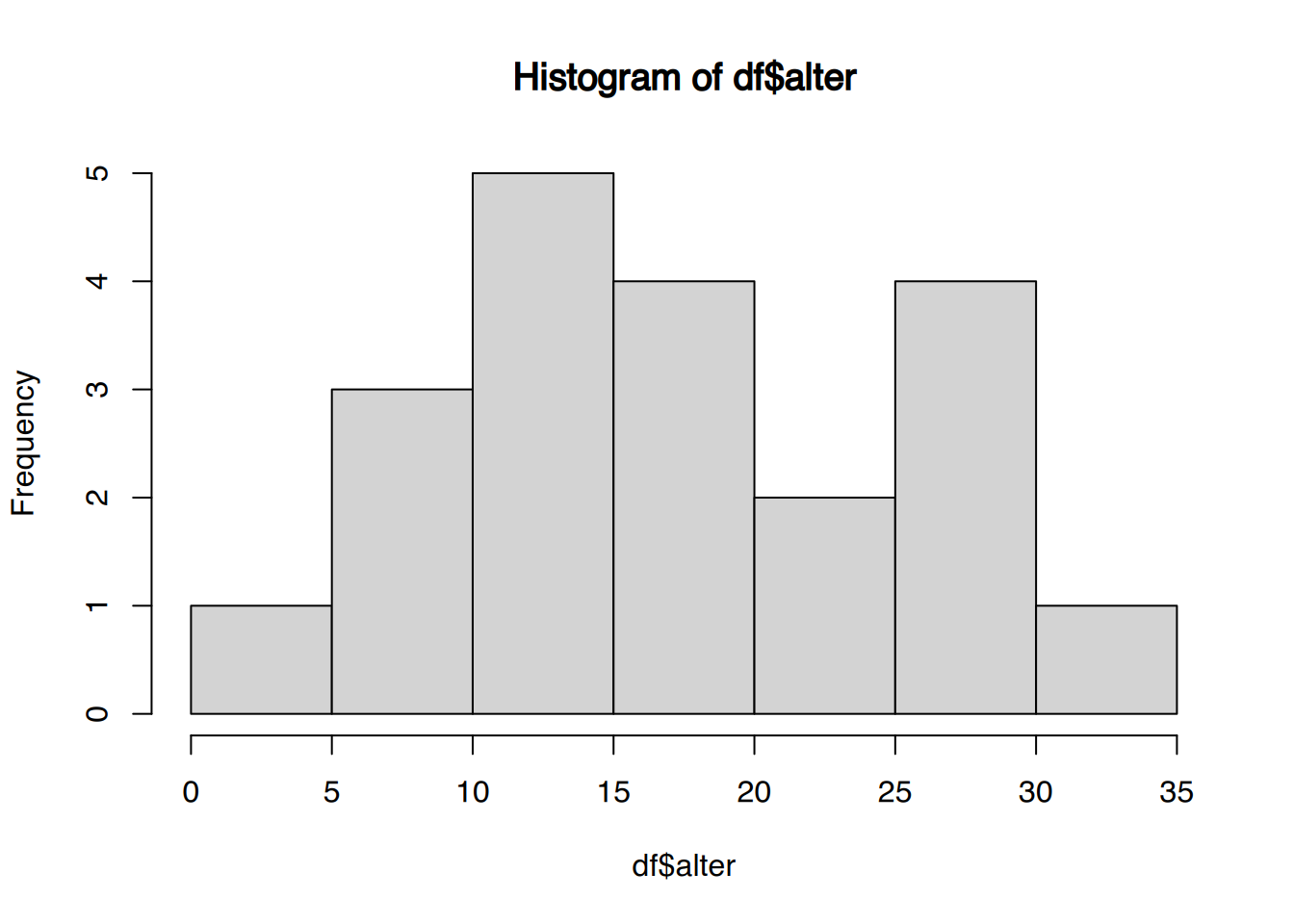
# etwas hübscher
hist(df$alter, breaks = 5, col="cyan",
main="Histogram",
xlab="Alter",
ylab="Häufigkeit") # 5er-Schritte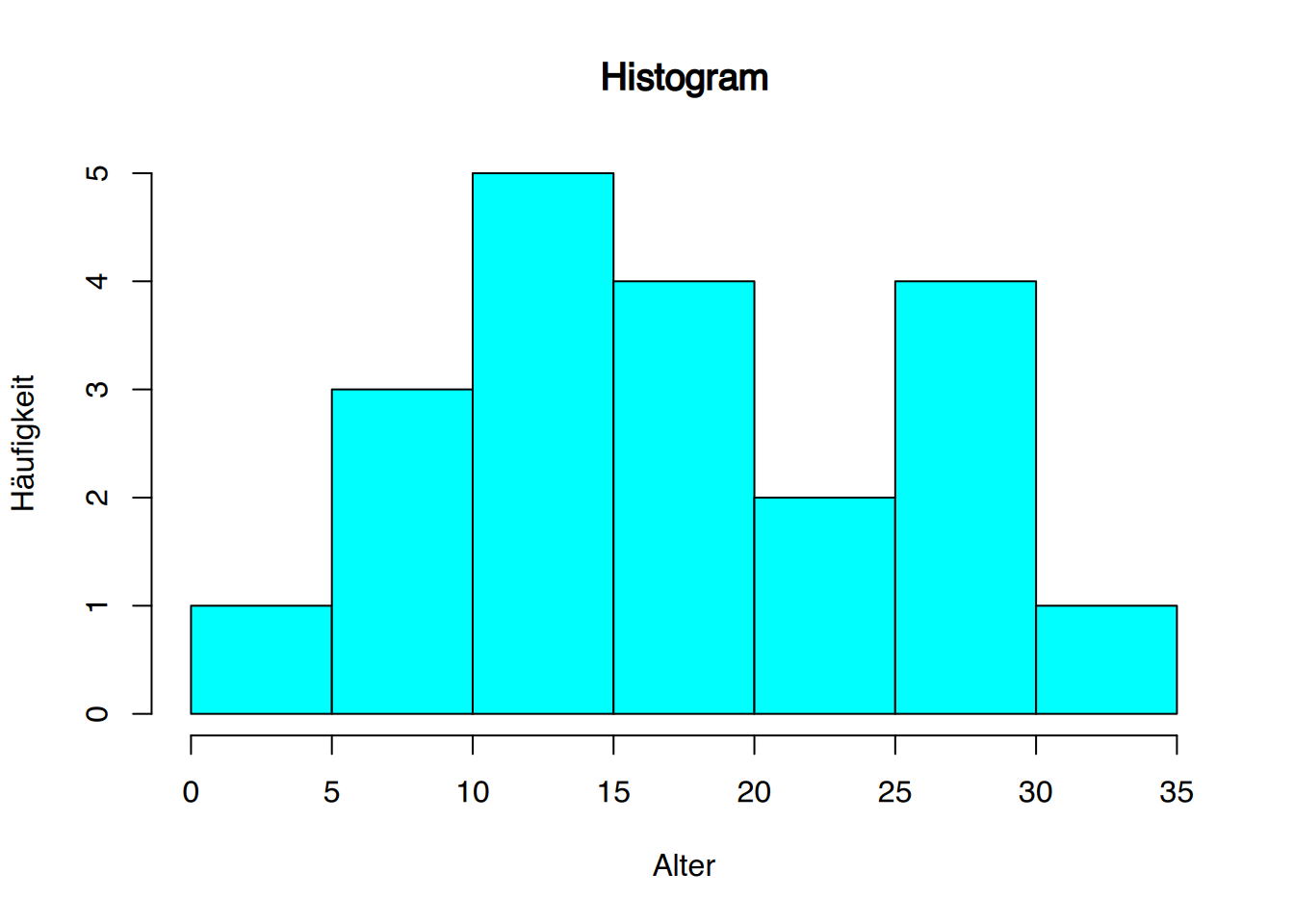
Für das Balkendiagramm nutzen wir die Funktion table() auf die Variable “alterk”.
# Häufigkeitstabelle von "alterk"
table(df$alterk)
0-5 6-10 11-15 16-20 21-25 26-30 31-35
1 3 5 4 2 4 1 # Balkendiagramm
barplot(table(df$alterk), horiz = TRUE)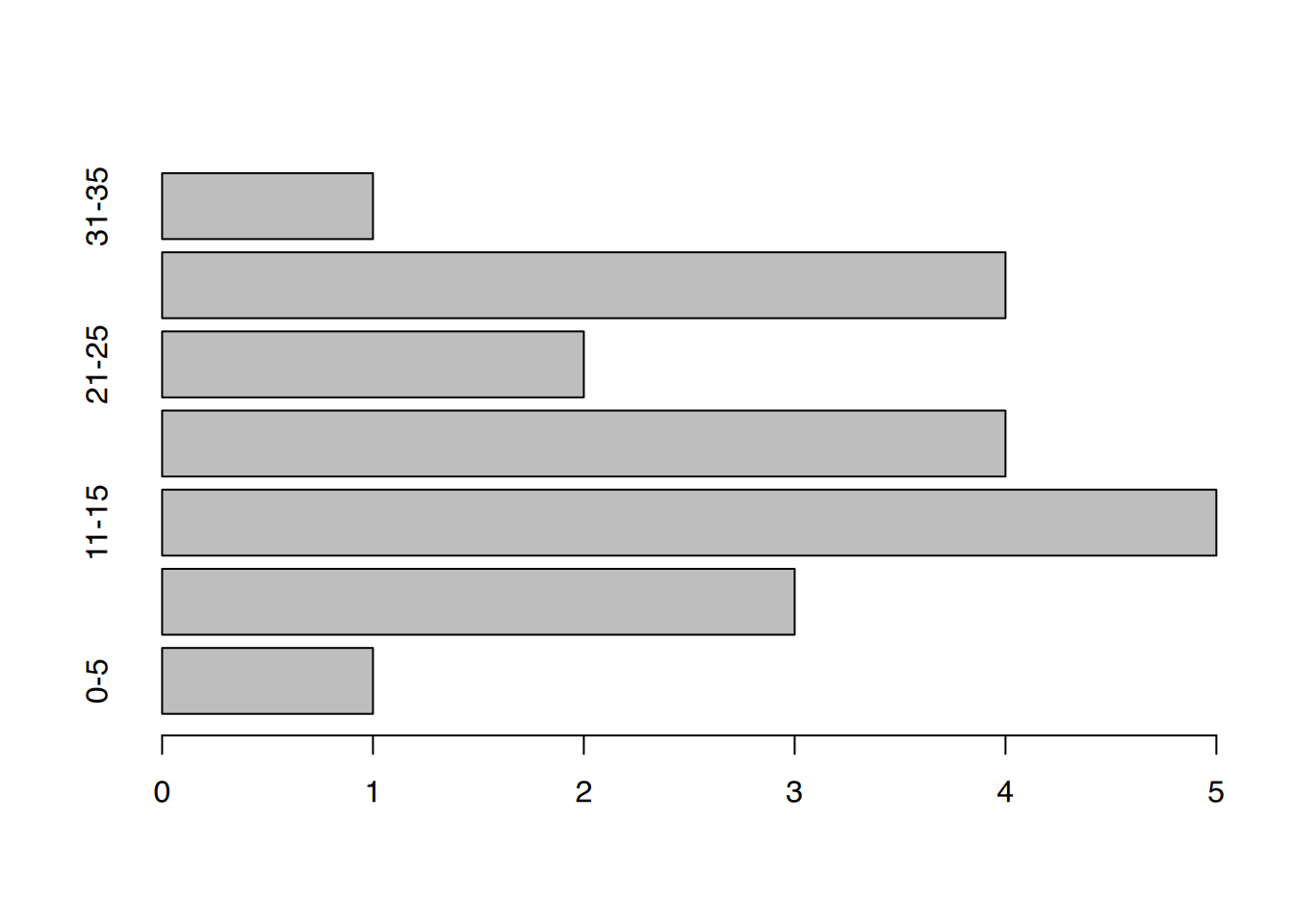
# etwas hübscher
barplot(table(df$alterk), horiz = TRUE,
col=rainbow(7),
main="Häufigkeiten der Altersklassen")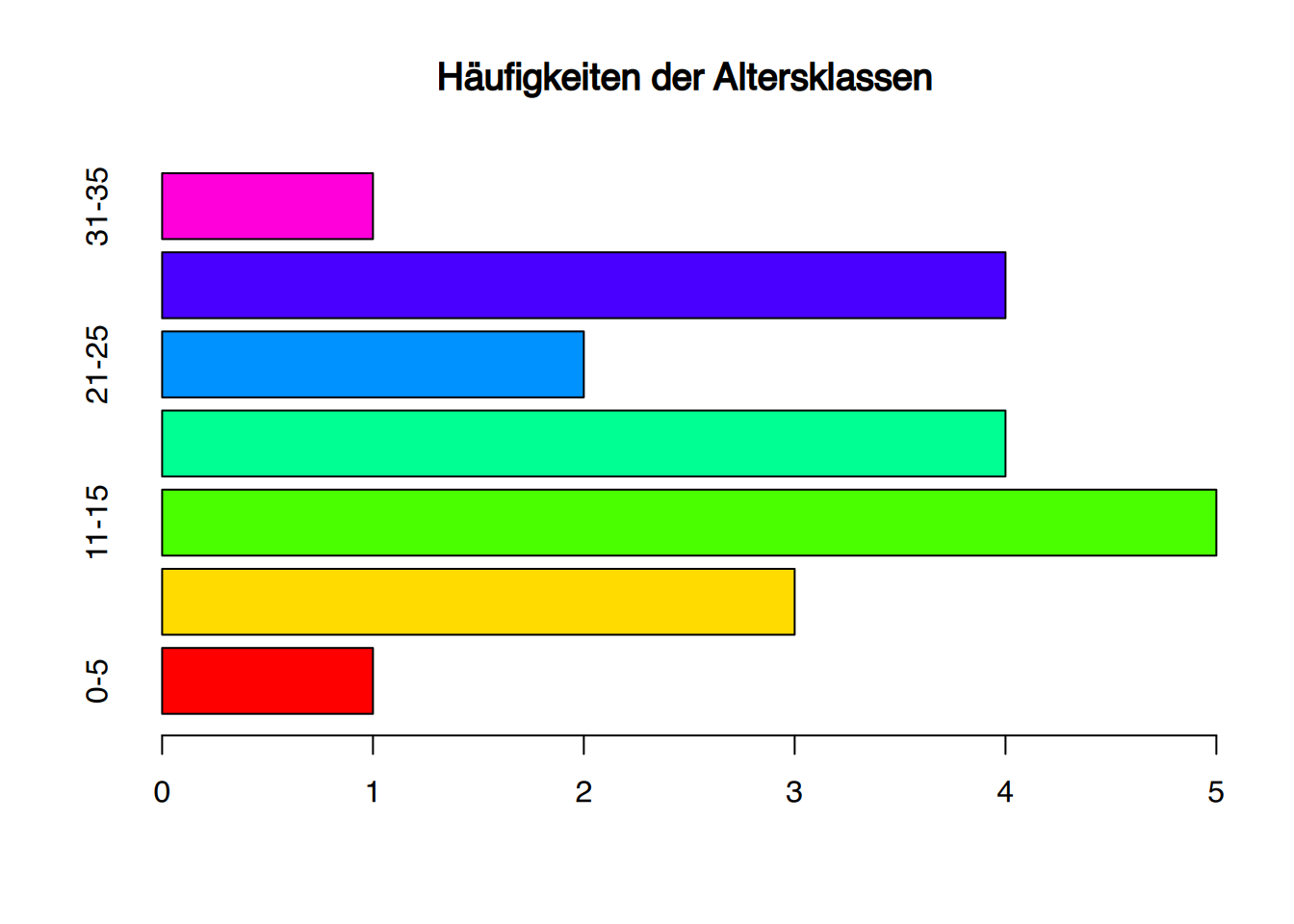
Altersgruppe und Geschlecht.
# Kontingenztafel
# entweder mit table()
table(df$alterk, df$geschlecht)
divers männlich weiblich
0-5 0 1 0
6-10 0 0 3
11-15 0 2 3
16-20 0 2 2
21-25 0 1 1
26-30 1 1 2
31-35 1 0 0# oder mit xtabs()
xtabs(~df$alterk+df$geschlecht) df$geschlecht
df$alterk divers männlich weiblich
0-5 0 1 0
6-10 0 0 3
11-15 0 2 3
16-20 0 2 2
21-25 0 1 1
26-30 1 1 2
31-35 1 0 0# in Dezimal-Prozent
prop.table(table(df$alterk, df$geschlecht))
divers männlich weiblich
0-5 0.00 0.05 0.00
6-10 0.00 0.00 0.15
11-15 0.00 0.10 0.15
16-20 0.00 0.10 0.10
21-25 0.00 0.05 0.05
26-30 0.05 0.05 0.10
31-35 0.05 0.00 0.00# in Prozent
prop.table(table(df$alterk, df$geschlecht))*100
divers männlich weiblich
0-5 0 5 0
6-10 0 0 15
11-15 0 10 15
16-20 0 10 10
21-25 0 5 5
26-30 5 5 10
31-35 5 0 0Altersgruppe und Geschlecht in einer geeigneten Graphik dar.
Geeignet ist ein geschichtetes Barplot.
# Barplot
barplot(table(df$geschlecht, df$alterk))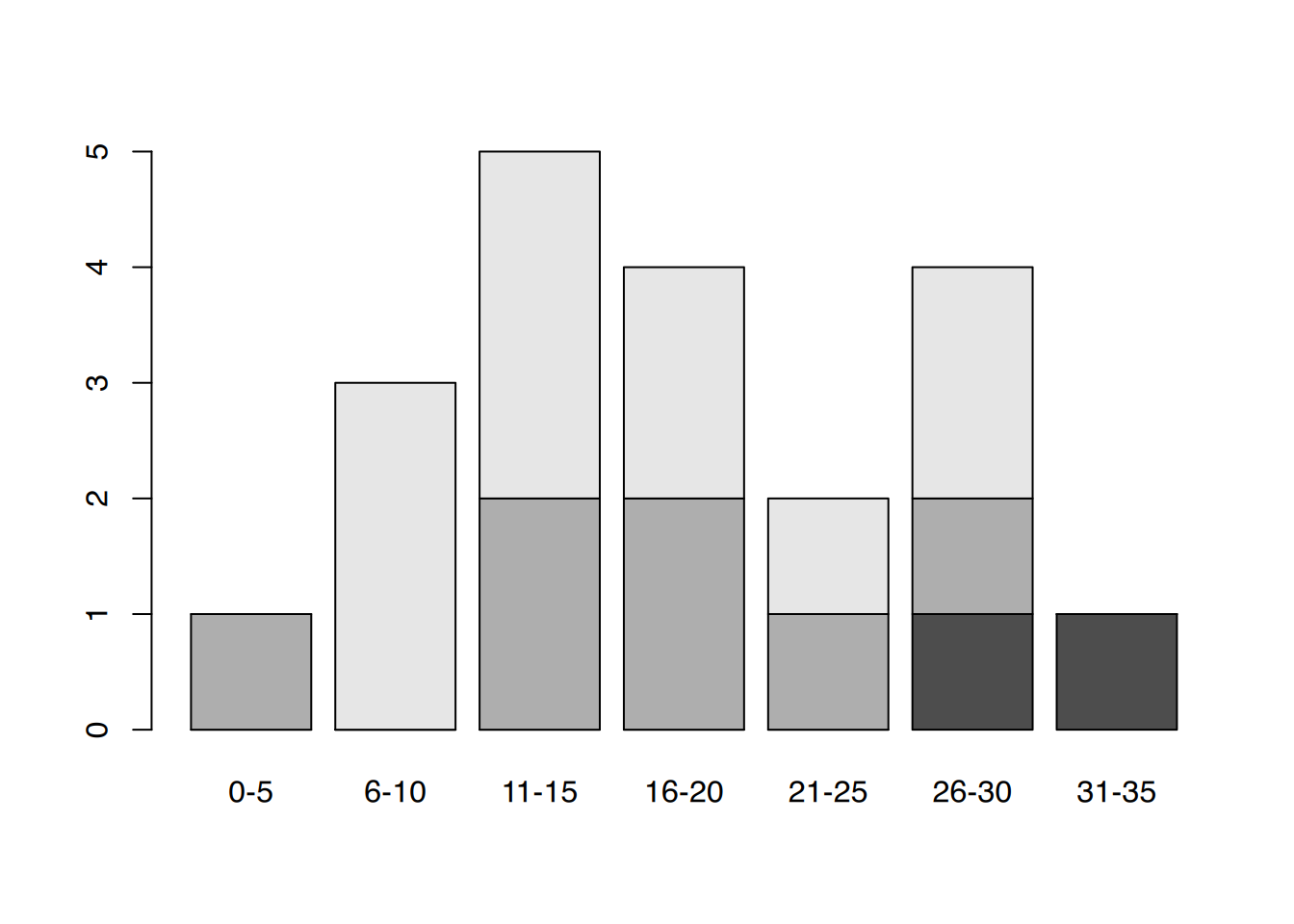
# mit Legendenbox
barplot(table(df$geschlecht, df$alterk), legend.text = levels(df$geschlecht))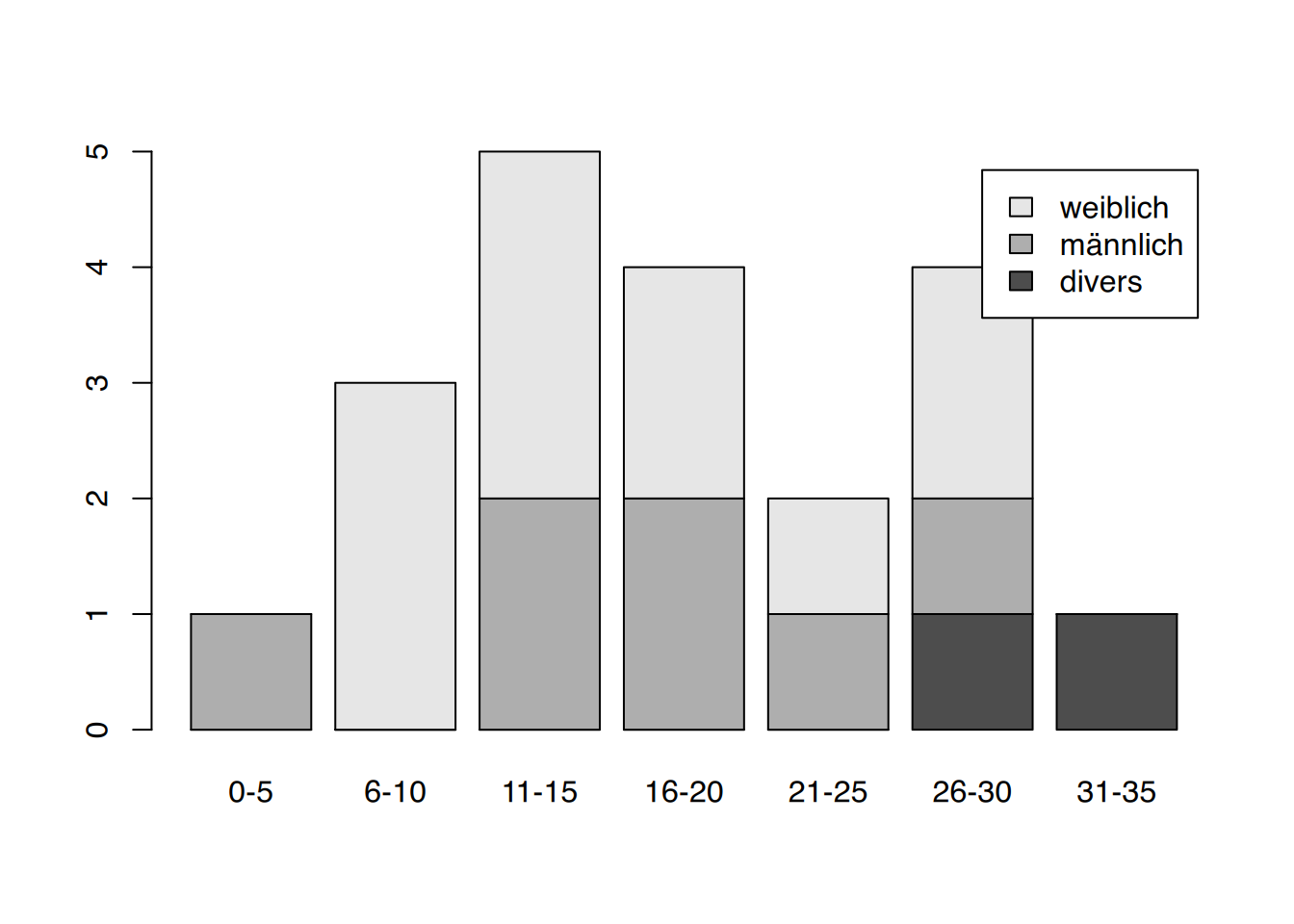
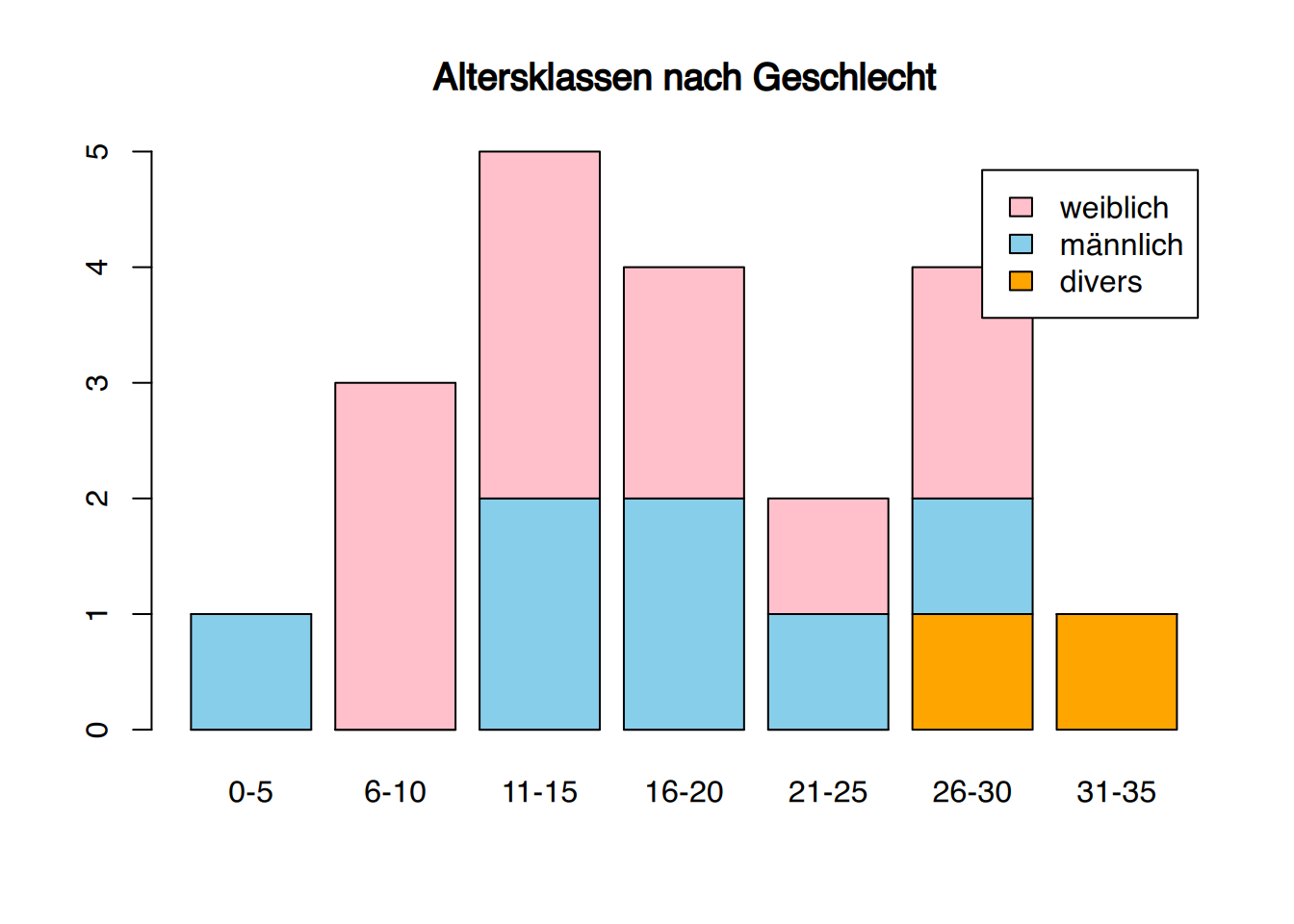
# hübscher
barplot(table(df$geschlecht, df$alterk),
legend.text = levels(df$geschlecht),
main="Altersklassen nach Geschlecht",
col=c("orange", "skyblue", "pink")
)
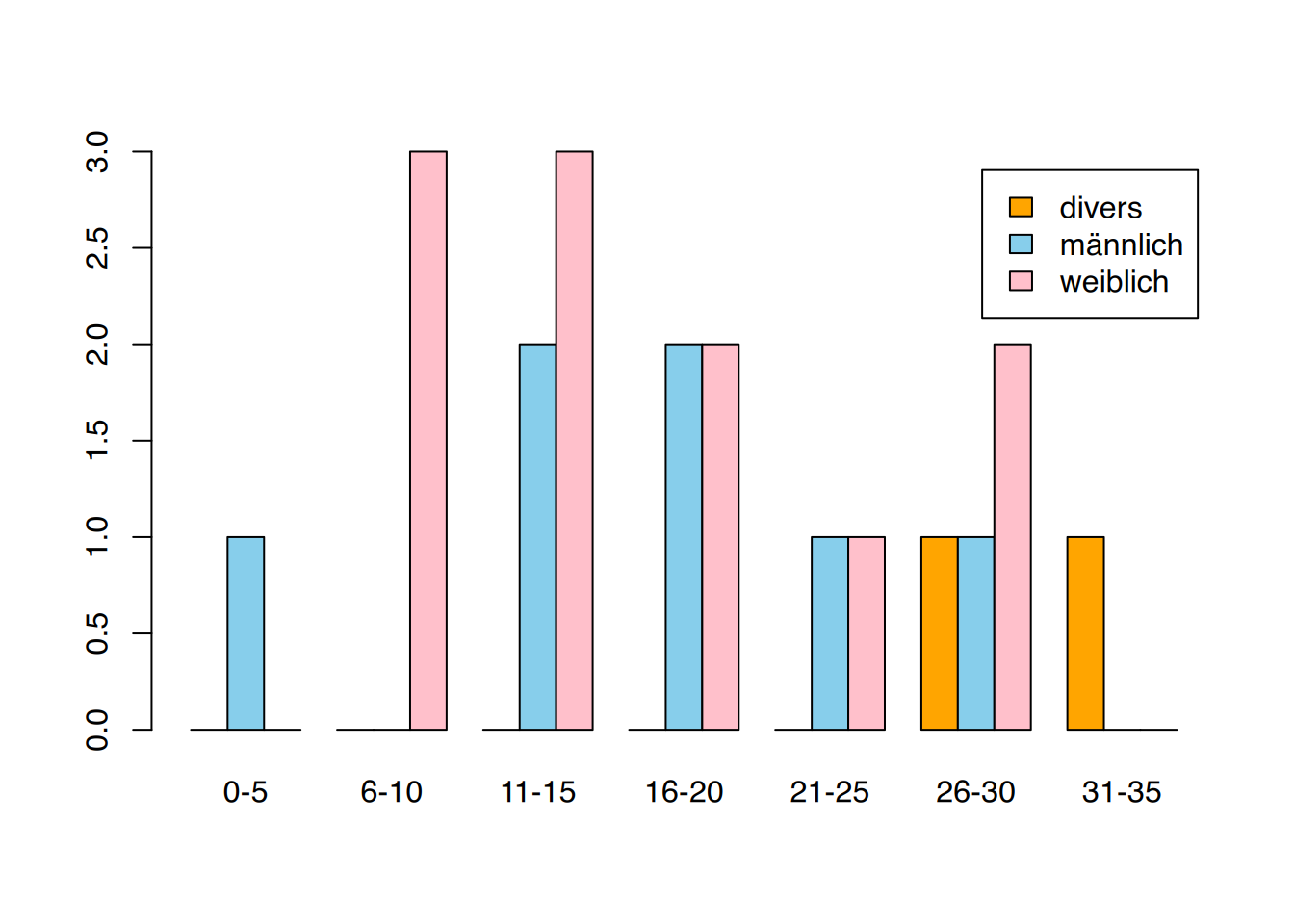
# variante "beside=TRUE"
barplot(table(df$geschlecht, df$alterk),
legend.text = levels(df$geschlecht),
beside = TRUE,
col=c("orange", "skyblue", "pink")
)
anscombe in Ihre R-Session.
# Lade Datensatz
data("anscombe")
# anschauen
str(anscombe)'data.frame': 11 obs. of 8 variables:
$ x1: num 10 8 13 9 11 14 6 4 12 7 ...
$ x2: num 10 8 13 9 11 14 6 4 12 7 ...
$ x3: num 10 8 13 9 11 14 6 4 12 7 ...
$ x4: num 8 8 8 8 8 8 8 19 8 8 ...
$ y1: num 8.04 6.95 7.58 8.81 8.33 ...
$ y2: num 9.14 8.14 8.74 8.77 9.26 8.1 6.13 3.1 9.13 7.26 ...
$ y3: num 7.46 6.77 12.74 7.11 7.81 ...
$ y4: num 6.58 5.76 7.71 8.84 8.47 7.04 5.25 12.5 5.56 7.91 ...x1 bis x4 und y1 bis y4) in 4 neue Datenframes mit den Namen Anscombe1 bis Anscombe4. Die enthaltenen Spalten sollten jeweils x und y heissen.
Anscombe1 <- data.frame(x=anscombe$x1, y=anscombe$y1)
Anscombe2 <- data.frame(x=anscombe$x2, y=anscombe$y2)
Anscombe3 <- data.frame(x=anscombe$x3, y=anscombe$y3)
Anscombe4 <- data.frame(x=anscombe$x4, y=anscombe$y4)### Datensatz Anscombe1
# Mittelwert für x, gerundet auf 2 Stellen
round(mean(Anscombe1$x), 2)[1] 9# Varianz für x
round(var(Anscombe1$x), 2)[1] 11# Mittelwert für y
round(mean(Anscombe1$y), 2)[1] 7.5# Varianz für y
round(var(Anscombe1$y), 2)[1] 4.13# Korrelationskoeffizient
round(cor(Anscombe1$x, Anscombe1$y), 2)[1] 0.82# Regression
fit <- lm(Anscombe1$y ~ Anscombe1$x)
round(fit$coefficients, 2)(Intercept) Anscombe1$x
3.0 0.5 ### Datensatz Anscombe2
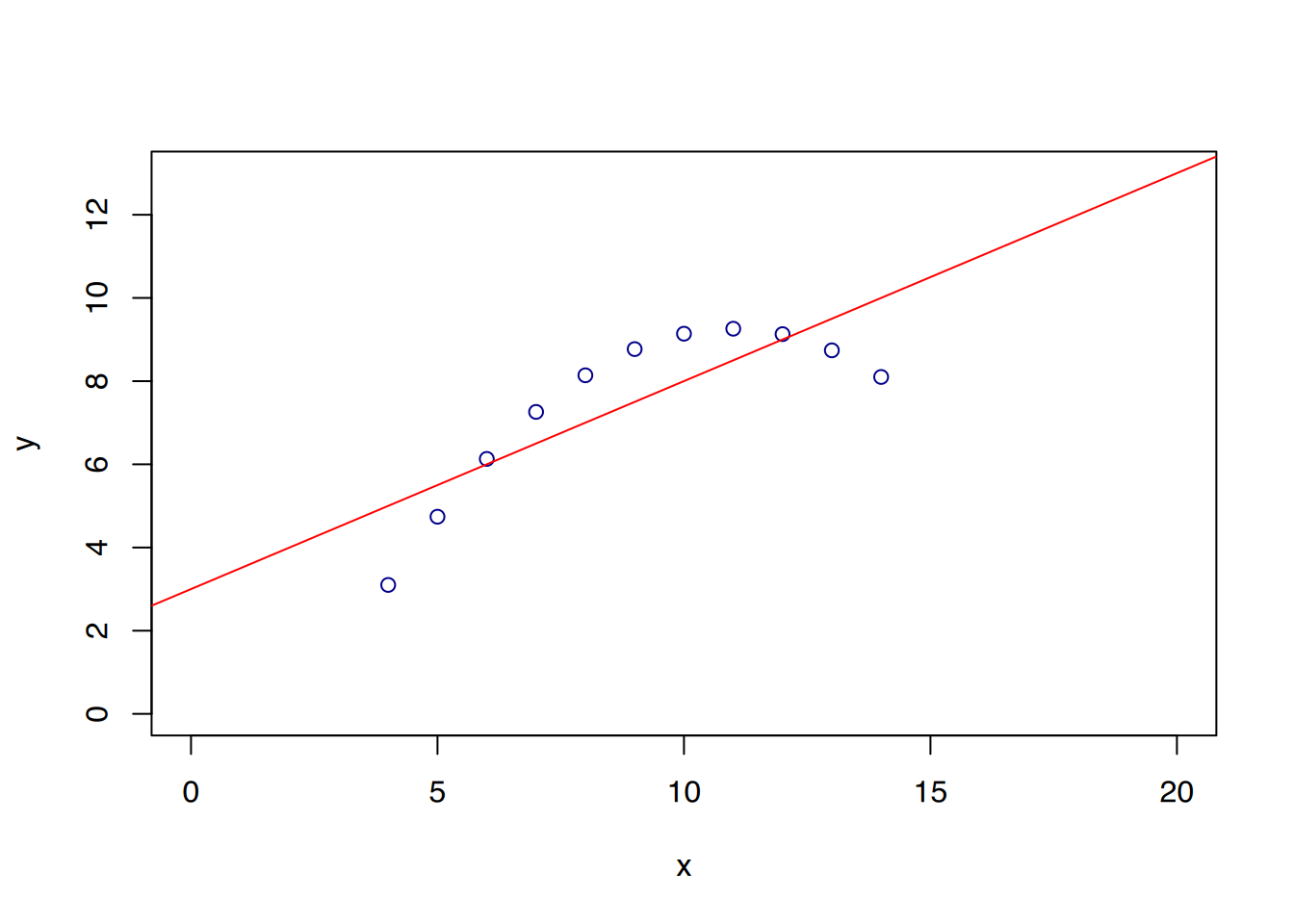
# Mittelwert für x, gerundet auf 2 Stellen
round(mean(Anscombe2$x), 2)[1] 9# Varianz für x
round(var(Anscombe2$x), 2)[1] 11# Mittelwert für y
round(mean(Anscombe2$y), 2)[1] 7.5# Varianz für y
round(var(Anscombe2$y), 2)[1] 4.13# Korrelationskoeffizient
round(cor(Anscombe2$x, Anscombe2$y), 2)[1] 0.82# Regression
fit <- lm(Anscombe2$y ~ Anscombe2$x)
round(fit$coefficients, 2)(Intercept) Anscombe2$x
3.0 0.5 ### Datensatz Anscombe3
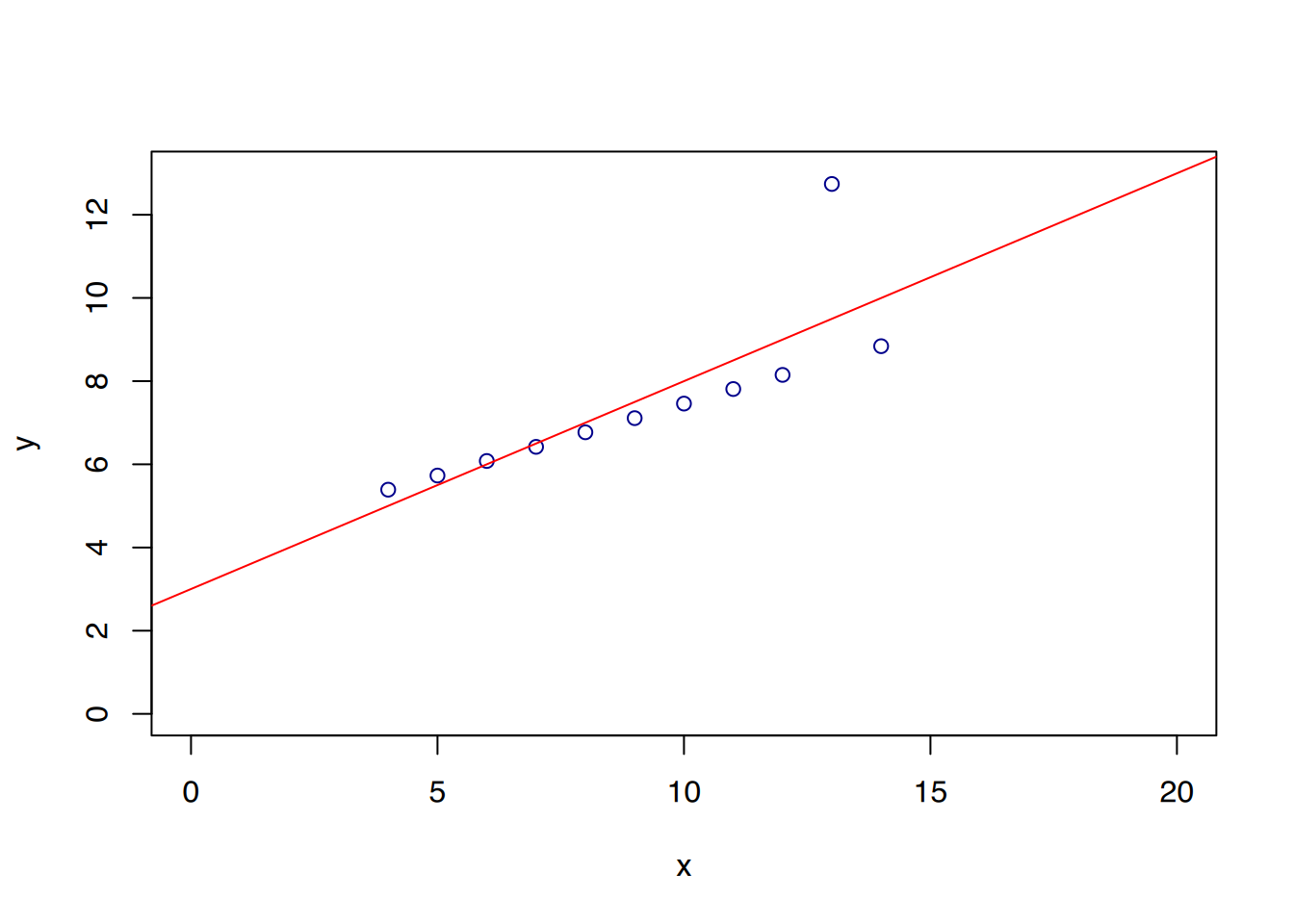
# Mittelwert für x, gerundet auf 2 Stellen
round(mean(Anscombe3$x), 2)[1] 9# Varianz für x
round(var(Anscombe3$x), 2)[1] 11# Mittelwert für y
round(mean(Anscombe3$y), 2)[1] 7.5# Varianz für y
round(var(Anscombe3$y), 2)[1] 4.12# Korrelationskoeffizient
round(cor(Anscombe3$x, Anscombe3$y), 2)[1] 0.82# Regression
fit <- lm(Anscombe3$y ~ Anscombe3$x)
round(fit$coefficients, 2)(Intercept) Anscombe3$x
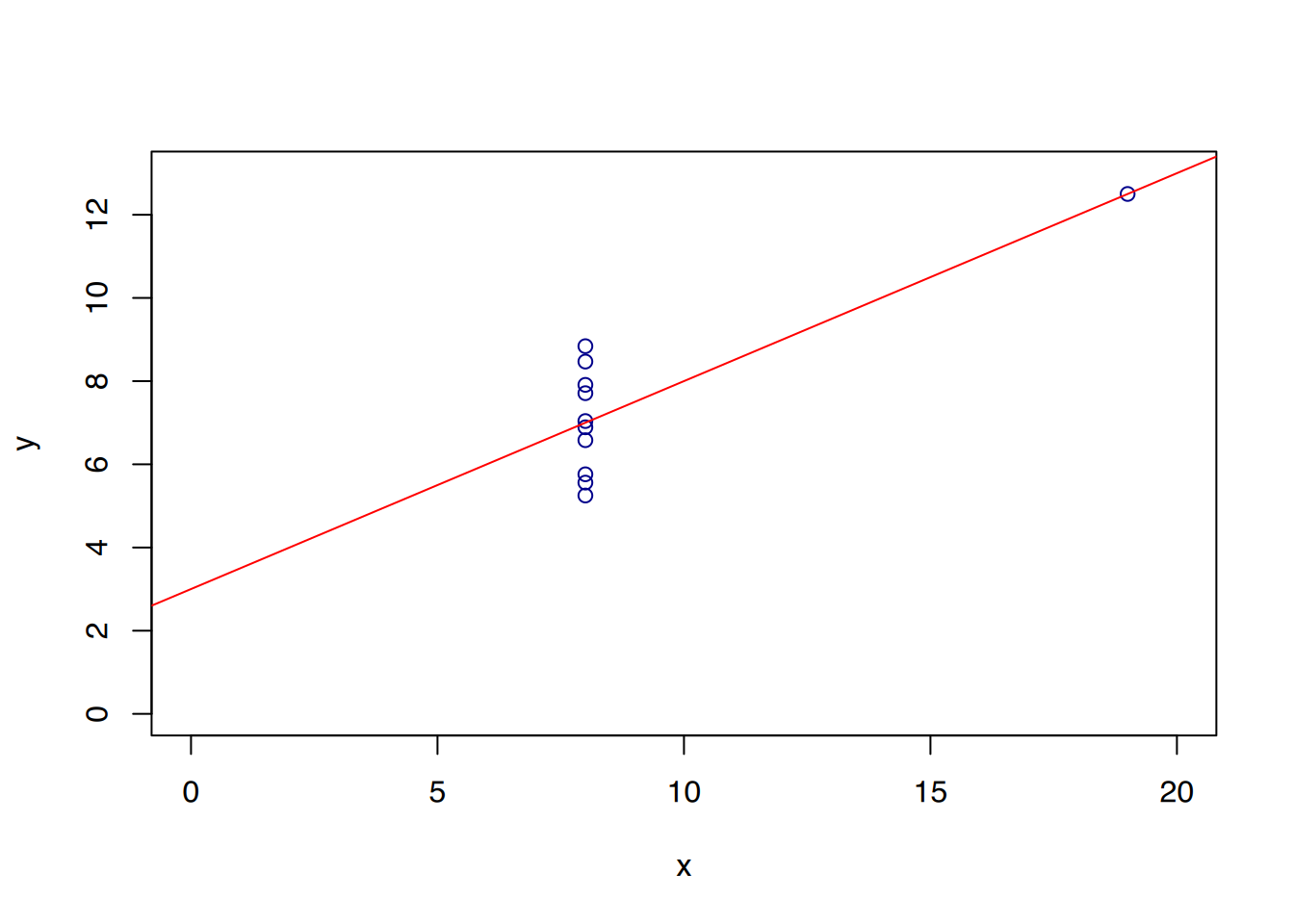
3.0 0.5 ### Datensatz Anscombe4
# Mittelwert für x, gerundet auf 2 Stellen
round(mean(Anscombe4$x), 2)[1] 9# Varianz für x
round(var(Anscombe4$x), 2)[1] 11# Mittelwert für y
round(mean(Anscombe4$y), 2)[1] 7.5# Varianz für y
round(var(Anscombe4$y), 2)[1] 4.12# Korrelationskoeffizient
round(cor(Anscombe4$x, Anscombe4$y), 2)[1] 0.82# Regression
fit <- lm(Anscombe4$y ~ Anscombe4$x)
round(fit$coefficients, 2)(Intercept) Anscombe4$x
3.0 0.5 plot()-Funktion, und hübschen Sie die Plots mit etwas Farbe auf.
# Datensatz Anscombe1
plot(Anscombe1$x, Anscombe1$y,
xlim = c(0,20), xlab="x",
ylim = c(0,13), ylab="y",
col="darkblue")
abline(lm(Anscombe1$y ~ Anscombe1$x), col="red")
# Datensatz Anscombe2
plot(Anscombe2$x, Anscombe2$y,
xlim = c(0,20), xlab="x",
ylim = c(0,13), ylab="y",
col="darkblue")
abline(lm(Anscombe2$y ~ Anscombe2$x), col="red")
# Datensatz Anscombe3
plot(Anscombe3$x, Anscombe3$y,
xlim = c(0,20), xlab="x",
ylim = c(0,13), ylab="y",
col="darkblue")
abline(lm(Anscombe3$y ~ Anscombe3$x), col="red")
# Datensatz Anscombe4
plot(Anscombe4$x, Anscombe4$y,
xlim = c(0,20), xlab="x",
ylim = c(0,13), ylab="y",
col="darkblue")
abline(lm(Anscombe4$y ~ Anscombe4$x), col="red")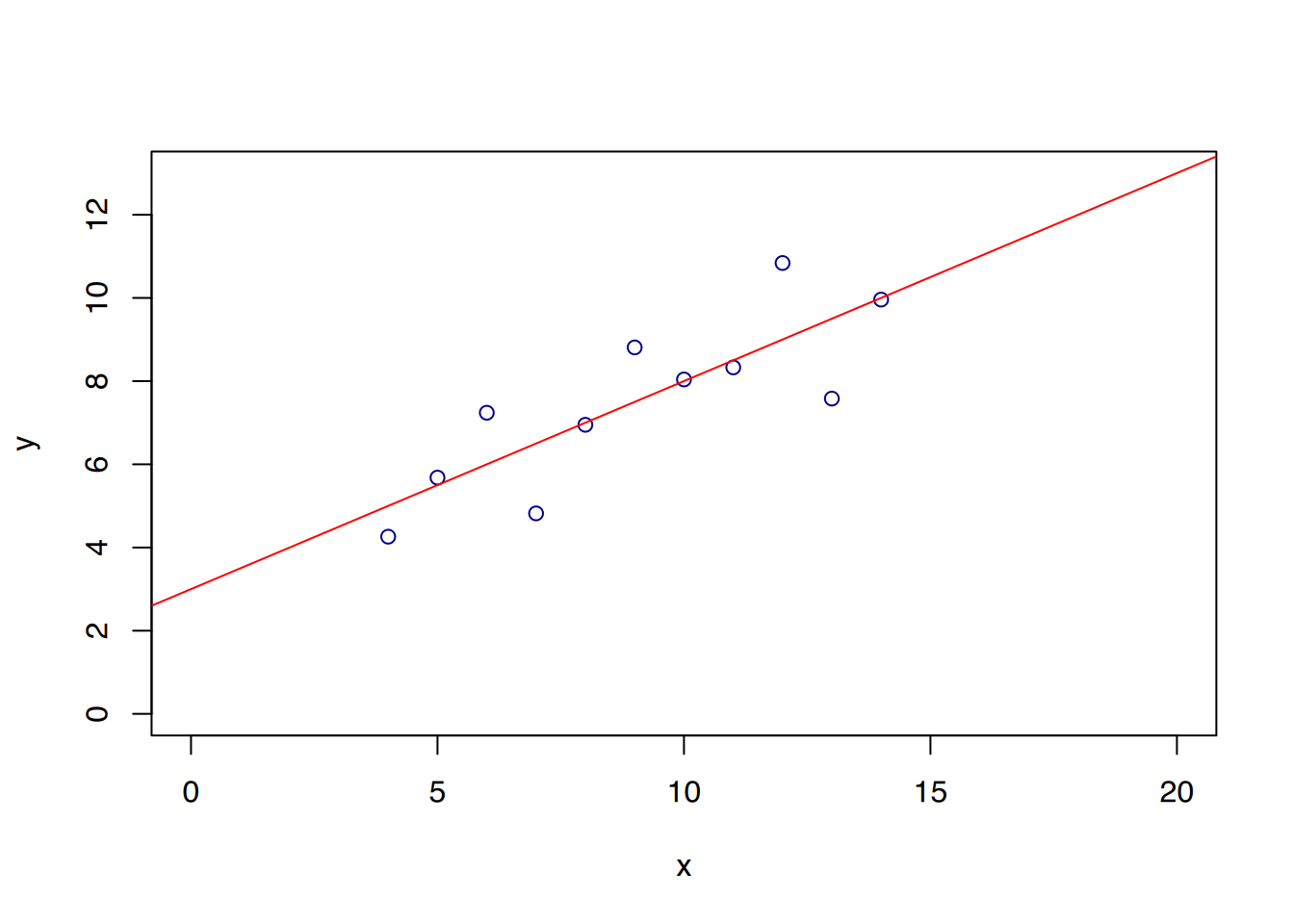
ggplot(), wobei alle 4 Diagramme mit einem Plotaufruf erzeugt werden sollen. Dies geht am einfachsten, wenn der Datensatz im Tidy-Data-Format (long table) vorliegt.
## Tidy-Longtable erzeugen
# Gruppen separieren
Anscombe1 <- data.frame(x=anscombe$x1, y=anscombe$y1, Gruppe="Anscombe1")
Anscombe2 <- data.frame(x=anscombe$x2, y=anscombe$y2, Gruppe="Anscombe2")
Anscombe3 <- data.frame(x=anscombe$x3, y=anscombe$y3, Gruppe="Anscombe3")
Anscombe4 <- data.frame(x=anscombe$x4, y=anscombe$y4, Gruppe="Anscombe4")
# alles zusammenfügen
df <- rbind(Anscombe1, Anscombe2, Anscombe3, Anscombe4)
# anschauen
str(df)'data.frame': 44 obs. of 3 variables:
$ x : num 10 8 13 9 11 14 6 4 12 7 ...
$ y : num 8.04 6.95 7.58 8.81 8.33 ...
$ Gruppe: chr "Anscombe1" "Anscombe1" "Anscombe1" "Anscombe1" ...# plotten
library(ggplot2)
ggplot(df) +
aes(x=x, y=y) +
xlim(0,20) +
ylim(0,13) +
geom_point(color="darkblue")+
geom_smooth(method="lm", color="red", se=FALSE) +
facet_wrap(~ Gruppe)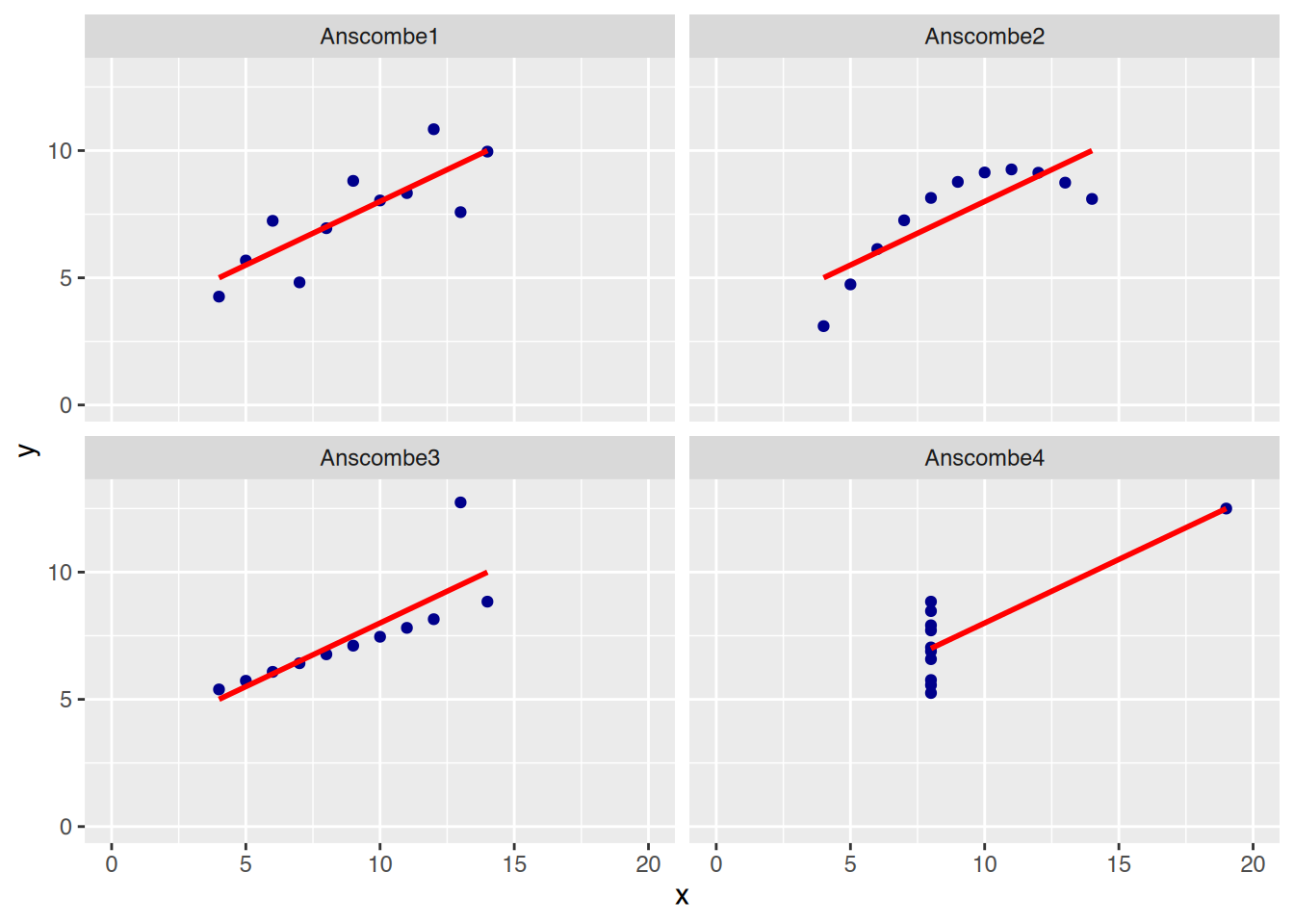
r
df <- data.frame(ehepaar = c(1:5),
kinder = c(0, 2, 3, 0, 1),
raeume = c(1, 4, 3, 2, 3))
# Korreltation nach Pearson
cor(df$kinder, df$raeume)[1] 0.7399401# regressionsmodelle immer in variable speichern
fit <- lm(raeume~kinder, data=df)
# Modellübersicht
summary(fit)
Call:
lm(formula = raeume ~ kinder, data = df)
Residuals:
1 2 3 4 5
-0.8235 0.8824 -0.7647 0.1765 0.5294
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.8235 0.5683 3.209 0.049 *
kinder 0.6471 0.3396 1.905 0.153
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.8856 on 3 degrees of freedom
Multiple R-squared: 0.5475, Adjusted R-squared: 0.3967
F-statistic: 3.63 on 1 and 3 DF, p-value: 0.1528# regressionsmodell plotten
plot(raeume~kinder, data=df) # Punktwolke
abline(fit) # Regressionsgerade hinzufügen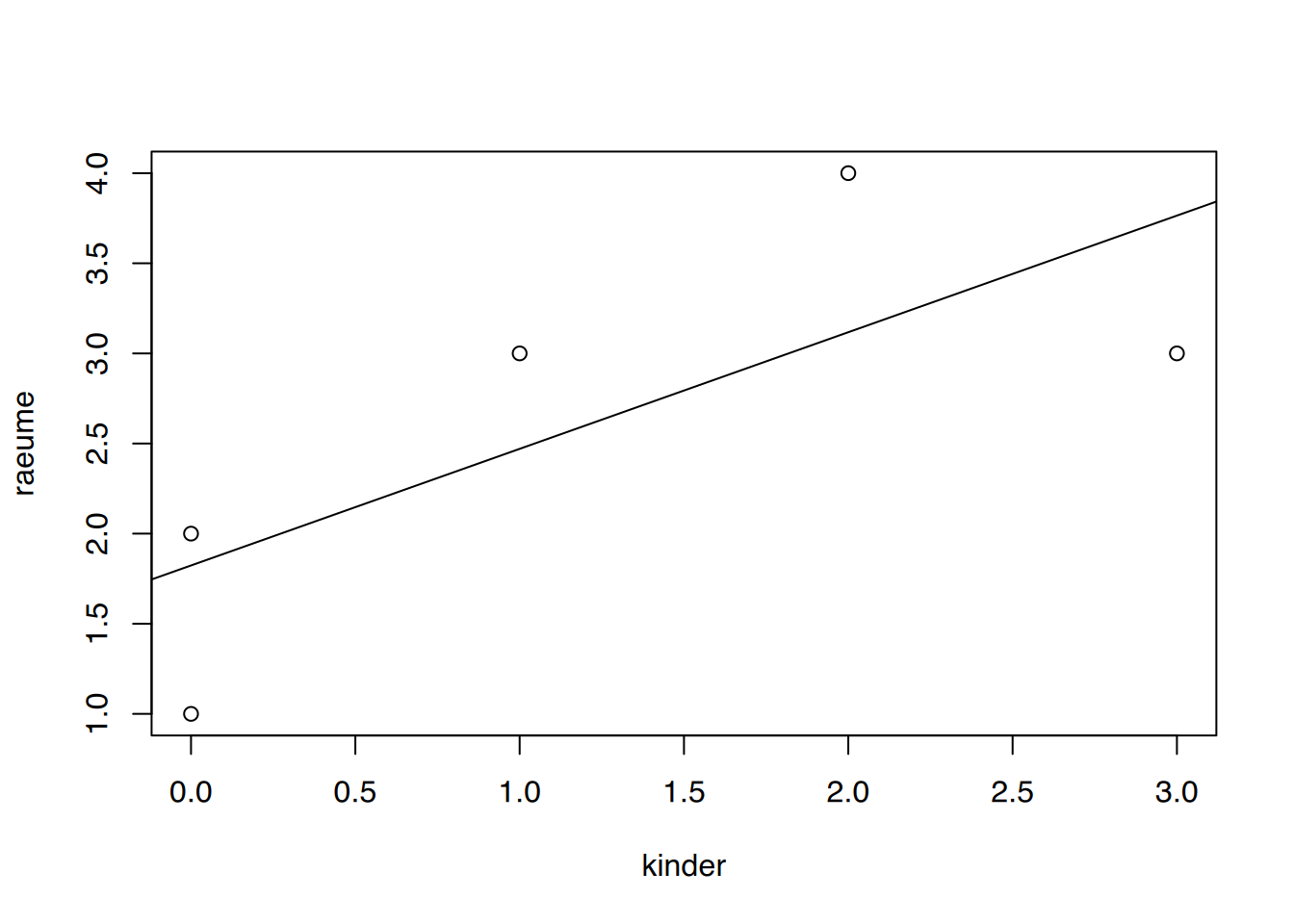
# etwas hübscher
plot(raeume~kinder, data=df,
col="skyblue",
pch=16,
main="Regressionsgerade",
xlab="Anzahl Kinder",
ylab="Anzahl Räume")
abline(fit, col="red")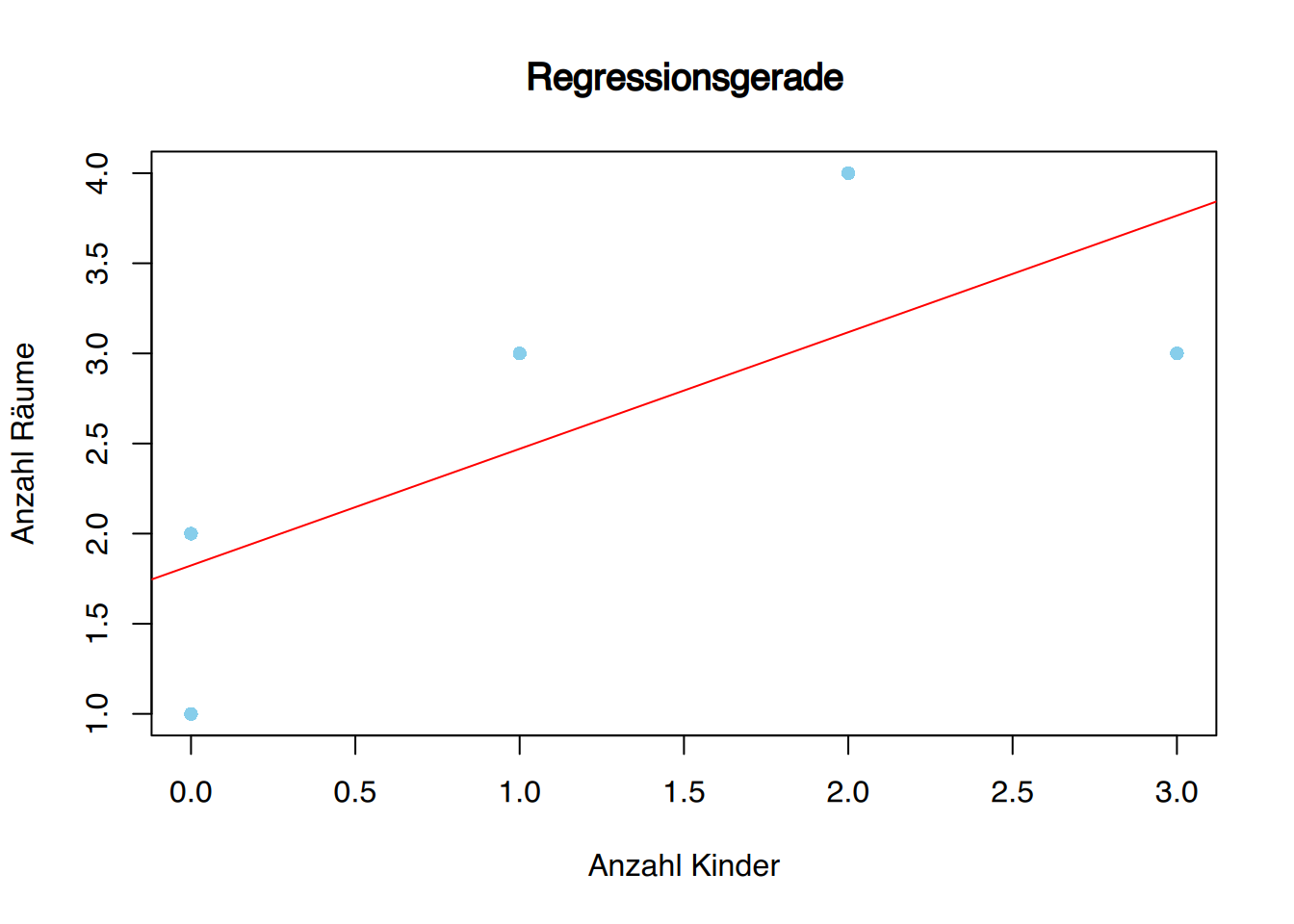
r
df <- data.frame(person = c(1:5),
kinder = c(1, 0, 3, 2, 1),
geschwister = c(0, 1, 4 , 1, 2))
# Korrelation
cor(df$kinder, df$geschwister)[1] 0.6939779# regressionsmodelle immer in variable speichern
fit <- lm(geschwister~kinder, data=df)
# Modellübersicht
summary(fit)
Call:
lm(formula = geschwister ~ kinder, data = df)
Residuals:
1 2 3 4 5
-1.2308 0.6923 0.9231 -1.1538 0.7692
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.3077 0.9577 0.321 0.769
kinder 0.9231 0.5529 1.669 0.194
Residual standard error: 1.261 on 3 degrees of freedom
Multiple R-squared: 0.4816, Adjusted R-squared: 0.3088
F-statistic: 2.787 on 1 and 3 DF, p-value: 0.1936Die Gleichung der Regressionsgeraden lautet \(y = 0,3077\ + 0,9231\cdot x\).
# regressionsmodell plotten
plot(geschwister~kinder, data=df) # Punktwolke
abline(fit) # Regressionsgerade hinzufügen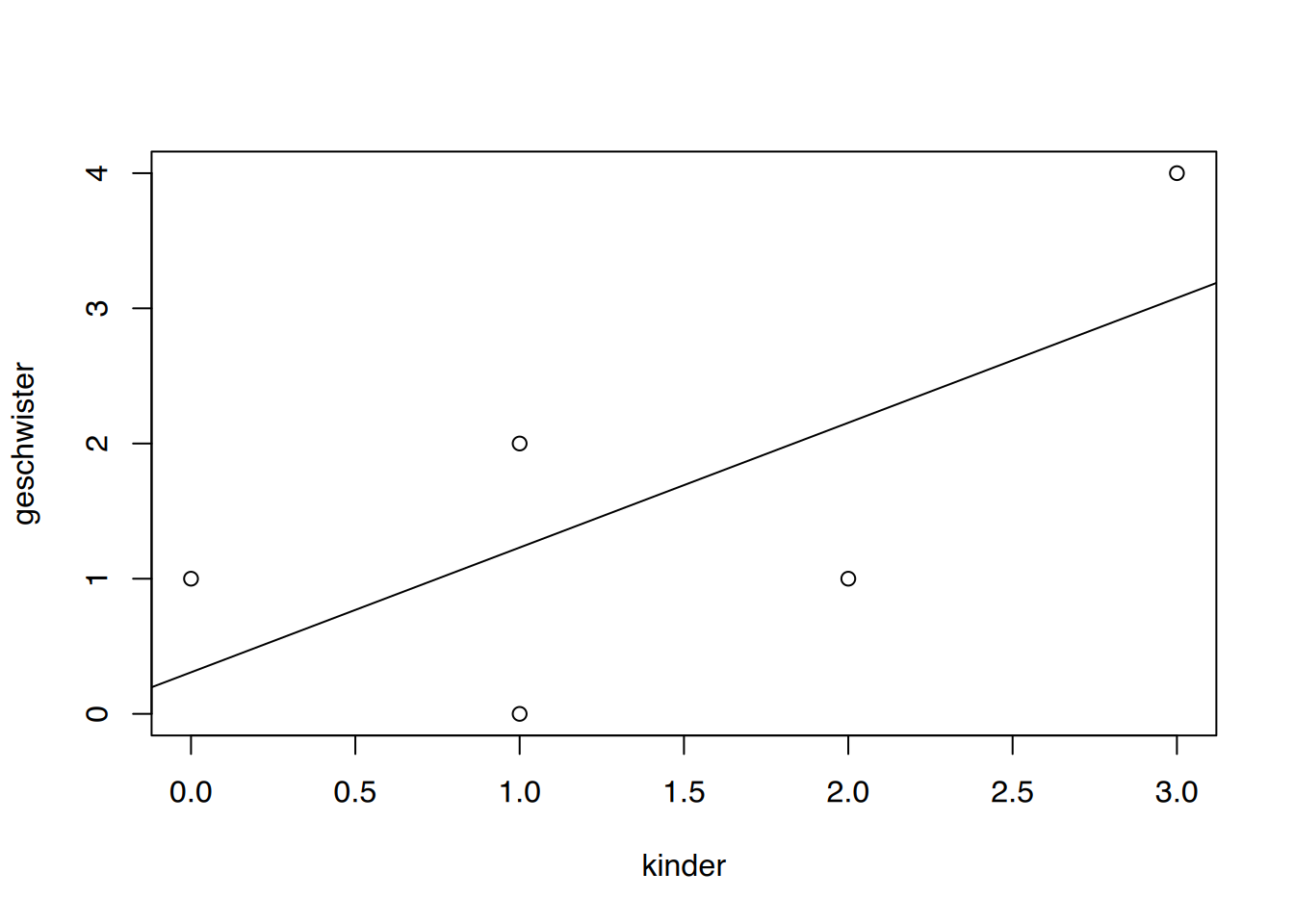
# etwas hübscher
plot(geschwister~kinder, data=df,
col="skyblue",
pch=16,
main="Regressionsgerade",
xlab="Anzahl Kinder",
ylab="Anzahl Geschwister")
abline(fit, col="red")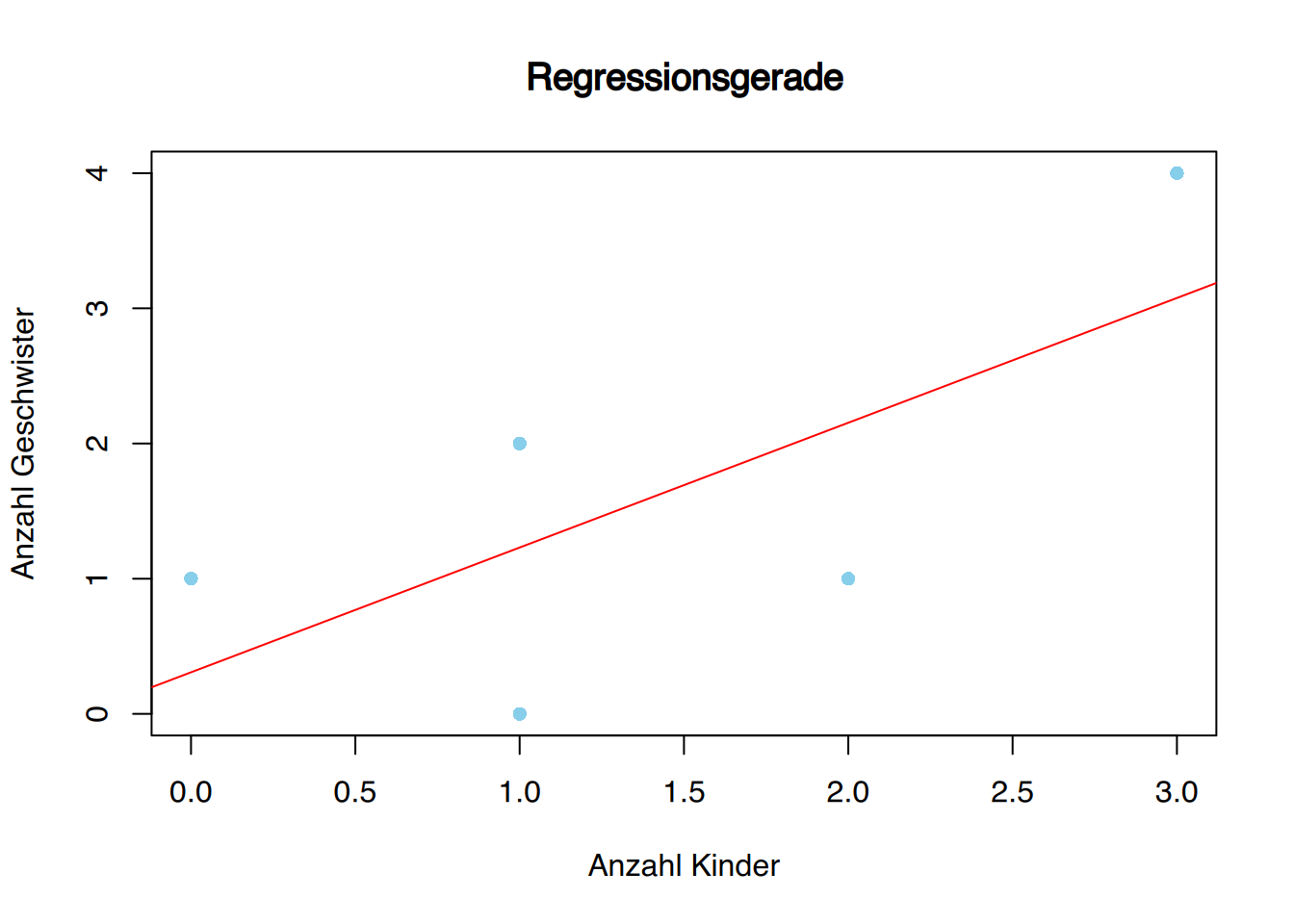
r und mit der Regressionsgeraden, falls Sie die Angaben der 3. Person streichen und dann die Auswertung wiederholen?
# dritte Person streichen
df <- df[-3,]
# Korrelation
cor(df$kinder, df$geschwister)[1] 0# Regression
fit <- lm(geschwister~kinder, data=df)
# Modellübersicht
summary(fit)
Call:
lm(formula = geschwister ~ kinder, data = df)
Residuals:
1 2 4 5
-1.000e+00 0.000e+00 5.551e-17 1.000e+00
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.000e+00 8.660e-01 1.155 0.368
kinder -3.925e-17 7.071e-01 0.000 1.000
Residual standard error: 1 on 2 degrees of freedom
Multiple R-squared: 2.465e-32, Adjusted R-squared: -0.5
F-statistic: 4.93e-32 on 1 and 2 DF, p-value: 1# etwas hübscher
plot(geschwister~kinder, data=df,
col="skyblue",
pch=16,
main="Regressionsgerade",
xlab="Anzahl Kinder",
ylab="Anzahl Geschwister")
abline(fit, col="red")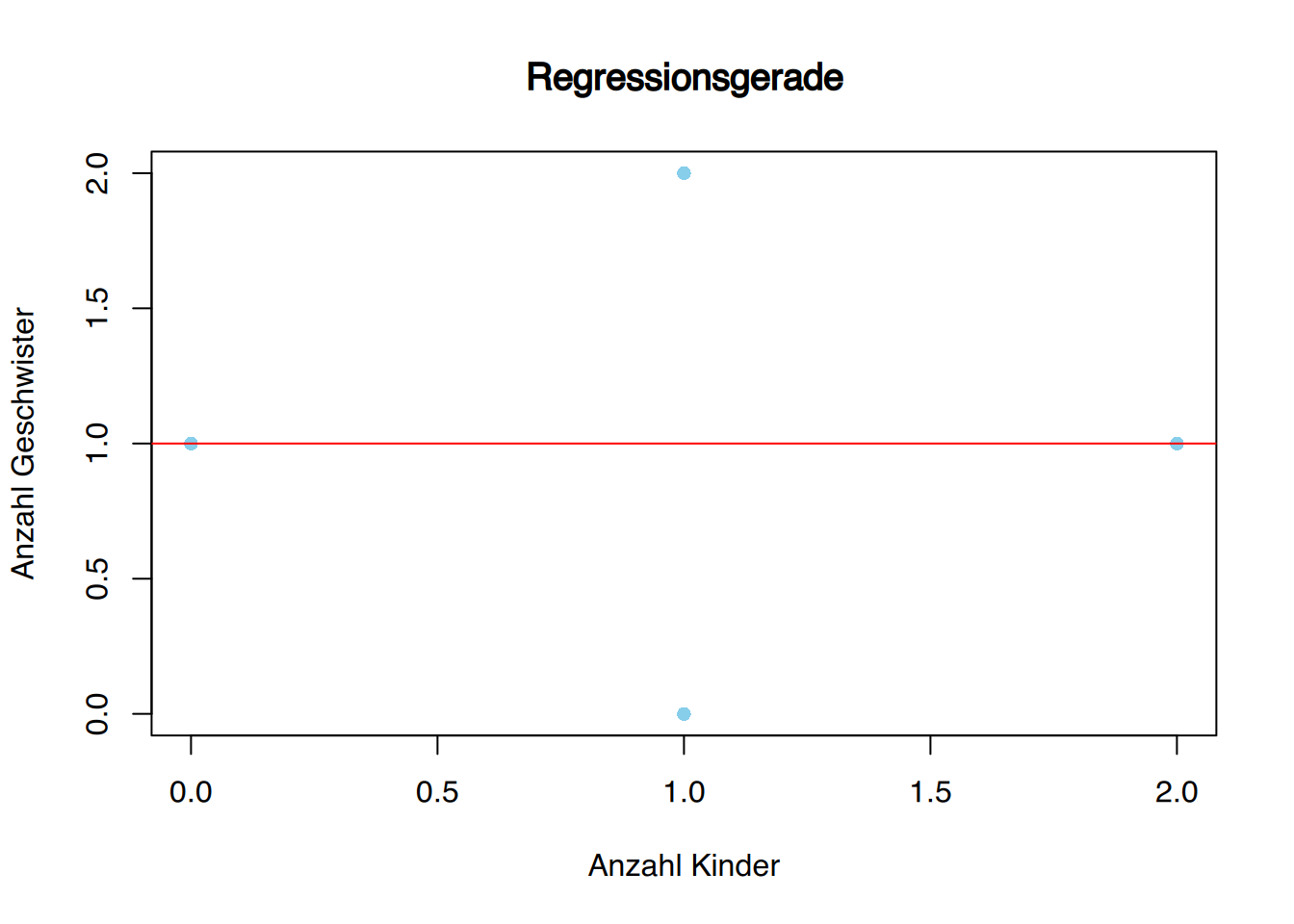
Wenn die 3. Person aus dem Datensatz entfernt wird, kann kein Zusammenhang zwischen geschwister und kinder gezeigt werden (r=0). Die Regressionsgerade verläuft parallel zur X-Achse, so dass Y für jedes X gleich ist.
# erstellen des Datensatzes mittels tribble()
library(tibble)
df <- tribble(
~Vorname, ~Geschlecht, ~Alter, ~Wohnort, ~Groesse, ~Gewicht, ~Rauchen,
"Hannah", "weiblich", 25, "Berlin", 1.75, 65, FALSE,
"Max", "maennlich", 30, "Hamburg", 1.85, 75, TRUE,
"Sophia", "weiblich", 20, "Muenchen", 1.65, 55, FALSE,
"Lukas", "maennlich", 35, "Frankfurt", 1.95, 85, TRUE,
"Emma", "weiblich", 18, "Stuttgart", 1.70, 60, FALSE,
"Jonas", "maennlich", 40, "Duesseldorf", 1.80, 70, TRUE,
"Lea", "weiblich", 22, "Hannover", 1.60, 50, FALSE,
"Jan", "divers", 28, "Nuernberg", 1.90, 80, TRUE,
"Mia", "weiblich", 24, "Bremen", 1.73, 63, FALSE,
"Luca", "maennlich", 33, "Gelsenkirchen", 1.88, 78, TRUE
)mutate() die Variablen Geschlecht und Wohnort in Faktoren um.
library(dplyr)
df <- df %>%
mutate(Geschlecht = factor(Geschlecht),
Wohnort = factor(Wohnort))
# anzeigen
glimpse(df)Rows: 10
Columns: 7
$ Vorname <chr> "Hannah", "Max", "Sophia", "Lukas", "Emma", "Jonas", "Lea",…
$ Geschlecht <fct> weiblich, maennlich, weiblich, maennlich, weiblich, maennli…
$ Alter <dbl> 25, 30, 20, 35, 18, 40, 22, 28, 24, 33
$ Wohnort <fct> Berlin, Hamburg, Muenchen, Frankfurt, Stuttgart, Duesseldor…
$ Groesse <dbl> 1.75, 1.85, 1.65, 1.95, 1.70, 1.80, 1.60, 1.90, 1.73, 1.88
$ Gewicht <dbl> 65, 75, 55, 85, 60, 70, 50, 80, 63, 78
$ Rauchen <lgl> FALSE, TRUE, FALSE, TRUE, FALSE, TRUE, FALSE, TRUE, FALSE, …filter(), um nur die Fälle anzuzeigen, die Raucher sind.
df %>%
filter(Rauchen == TRUE)# A tibble: 5 × 7
Vorname Geschlecht Alter Wohnort Groesse Gewicht Rauchen
<chr> <fct> <dbl> <fct> <dbl> <dbl> <lgl>
1 Max maennlich 30 Hamburg 1.85 75 TRUE
2 Lukas maennlich 35 Frankfurt 1.95 85 TRUE
3 Jonas maennlich 40 Duesseldorf 1.8 70 TRUE
4 Jan divers 28 Nuernberg 1.9 80 TRUE
5 Luca maennlich 33 Gelsenkirchen 1.88 78 TRUE group_by() und summarise(), um Mittelwert, Standardabweichung und Median der Variable Alter für jedes Geschlecht zu berechnen.
df %>%
group_by(Geschlecht) %>%
summarise(MW = mean(Alter),
SD = sd(Alter),
Median = median(Alter))# A tibble: 3 × 4
Geschlecht MW SD Median
<fct> <dbl> <dbl> <dbl>
1 divers 28 NA 28
2 maennlich 34.5 4.20 34
3 weiblich 21.8 2.86 22arrange(), um den Datensatz nach Wohnort in alphabetischer Reihenfolge zu sortieren.
df %>%
arrange(Wohnort)# A tibble: 10 × 7
Vorname Geschlecht Alter Wohnort Groesse Gewicht Rauchen
<chr> <fct> <dbl> <fct> <dbl> <dbl> <lgl>
1 Hannah weiblich 25 Berlin 1.75 65 FALSE
2 Mia weiblich 24 Bremen 1.73 63 FALSE
3 Jonas maennlich 40 Duesseldorf 1.8 70 TRUE
4 Lukas maennlich 35 Frankfurt 1.95 85 TRUE
5 Luca maennlich 33 Gelsenkirchen 1.88 78 TRUE
6 Max maennlich 30 Hamburg 1.85 75 TRUE
7 Lea weiblich 22 Hannover 1.6 50 FALSE
8 Sophia weiblich 20 Muenchen 1.65 55 FALSE
9 Jan divers 28 Nuernberg 1.9 80 TRUE
10 Emma weiblich 18 Stuttgart 1.7 60 FALSE R
# überführe in ordinalen Factor
noten <- factor(c("2", "2", "4+", "2", "2-", "durchgefallen", "2", "2-", "2+",
"2+", "4", "2", "2", "3-", "2", "2", "1-", "2", "2", "4",
"3+", "2-", "2-", "2+", "1+", "1", "2", "2+", "3+", "2-",
"3-", "1-", "3", "1-", "4", "4+", "2", "3+", "3", "2-", "2",
"1-", "3+", "1+", "3", "2", "durchgefallen", "2-", "1-",
"2+", "3", "3+", "2-", "2+", "2+", "durchgefallen", "2-",
"2+", "2+", "2+", "1", "1-", "2", "4", "1-", "1+", "3", "2+",
"2-", "2+", "2-", "2-", "2-", "4", "3+", "1", "2-",
"durchgefallen", "1", "2+", "2-", "2"),
ordered=TRUE,
levels = c("1+", "1", "1-", "2+", "2", "2-",
"3+", "3", "3-", "4+", "4",
"durchgefallen")
)freqTable() zu verwenden.
# überführe in Datenframe
df <- as.data.frame(table(noten))
# kumuliere
df$Hkum <- cumsum(df$Freq)
# relative Häufigkeiten
df$rel <- as.numeric(prop.table(table(noten)))
# kumulierte relative
df$relkum <- cumsum(df$rel)
# umbenennen
colnames(df) <- c("Note", "Haeufig", "Hkum", "Relativ", "Rkum")
# anzeigen
df Note Haeufig Hkum Relativ Rkum
1 1+ 3 3 0.03658537 0.03658537
2 1 4 7 0.04878049 0.08536585
3 1- 7 14 0.08536585 0.17073171
4 2+ 13 27 0.15853659 0.32926829
5 2 16 43 0.19512195 0.52439024
6 2- 15 58 0.18292683 0.70731707
7 3+ 6 64 0.07317073 0.78048780
8 3 5 69 0.06097561 0.84146341
9 3- 2 71 0.02439024 0.86585366
10 4+ 2 73 0.02439024 0.89024390
11 4 5 78 0.06097561 0.95121951
12 durchgefallen 4 82 0.04878049 1.00000000# Rbase
barplot(prop.table(table(noten)),
col="steelblue",
xlab="Abschlussnote",
ylab="relative Häufigkeit")
# ggplot()
library(ggplot2)
ggplot(df, aes(x=Note, y=Relativ)) +
geom_bar(stat="identity", fill="steelblue", color="black") +
xlab("Abschlussnote") +
ylab("relative Häufigkeit")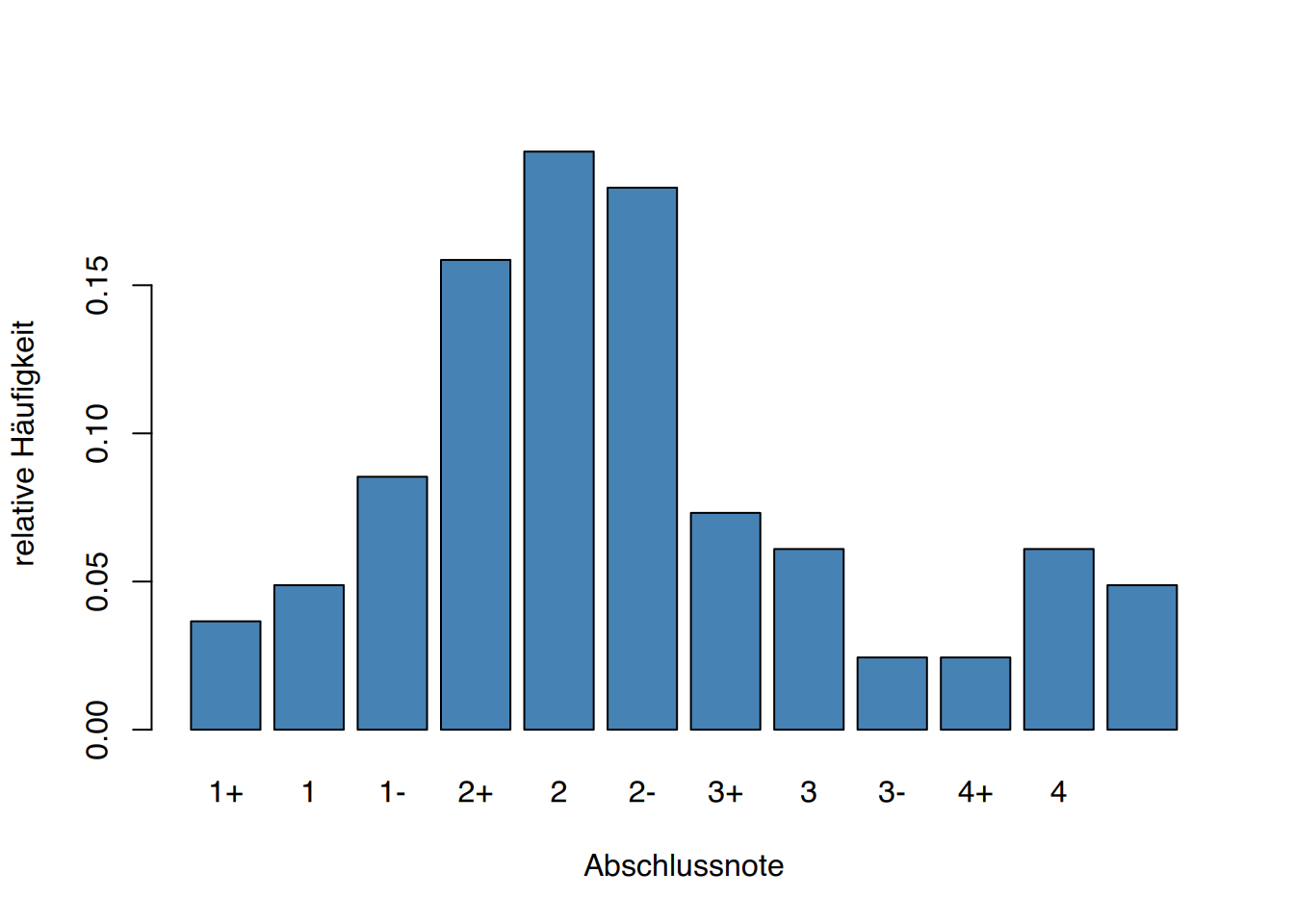
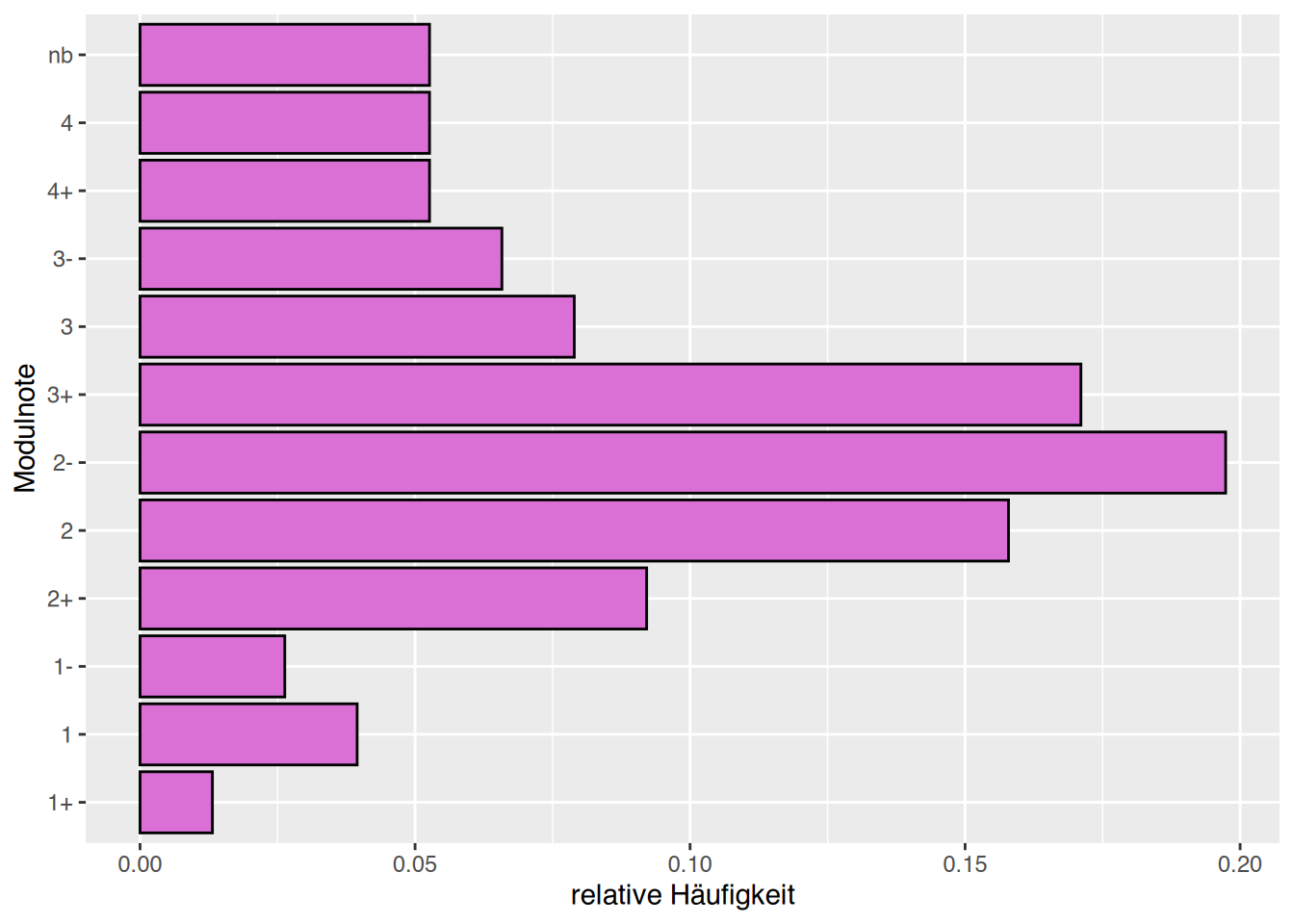
rep()-Funktion in R
# überführe mittels rep()
noten <- c( rep("1+",1), rep("1", 3), rep("1-", 2),
rep("2+",7), rep("2", 12), rep("2-", 15),
rep("3+",13), rep("3", 6), rep("3-", 5),
rep("4+",4), rep("4", 4), rep("nb", 4))
# überführe in ordinalen Factor
noten <- factor(noten, ordered=TRUE,
levels = c("1+", "1", "1-", "2+", "2", "2-",
"3+", "3", "3-", "4+", "4", "nb")
)jgsbook::freqTable() zu verwenden.
# überführe in Datenframe
df <- as.data.frame(table(noten))
# kumuliere
df$Hkum <- cumsum(df$Freq)
# relative Häufigkeiten
df$rel <- as.numeric(prop.table(table(noten)))
# kumulierte relative
df$relkum <- cumsum(df$rel)
# umbenennen
colnames(df) <- c("Note", "Haeufig", "Hkum", "Relativ", "Rkum")
# anzeigen
df Note Haeufig Hkum Relativ Rkum
1 1+ 1 1 0.01315789 0.01315789
2 1 3 4 0.03947368 0.05263158
3 1- 2 6 0.02631579 0.07894737
4 2+ 7 13 0.09210526 0.17105263
5 2 12 25 0.15789474 0.32894737
6 2- 15 40 0.19736842 0.52631579
7 3+ 13 53 0.17105263 0.69736842
8 3 6 59 0.07894737 0.77631579
9 3- 5 64 0.06578947 0.84210526
10 4+ 4 68 0.05263158 0.89473684
11 4 4 72 0.05263158 0.94736842
12 nb 4 76 0.05263158 1.00000000# Rbase
barplot(prop.table(table(noten)),
col="orchid",
# schalte um auf "Balkendiagramm"
horiz=TRUE,
ylab="Modulnote",
xlab="relative Häufigkeit")
# ggplot()
library(ggplot2)
ggplot(df, aes(y=Note, x=Relativ)) +
geom_bar(stat="identity", fill="orchid", color="black") +
ylab("Modulnote") +
xlab("relative Häufigkeit")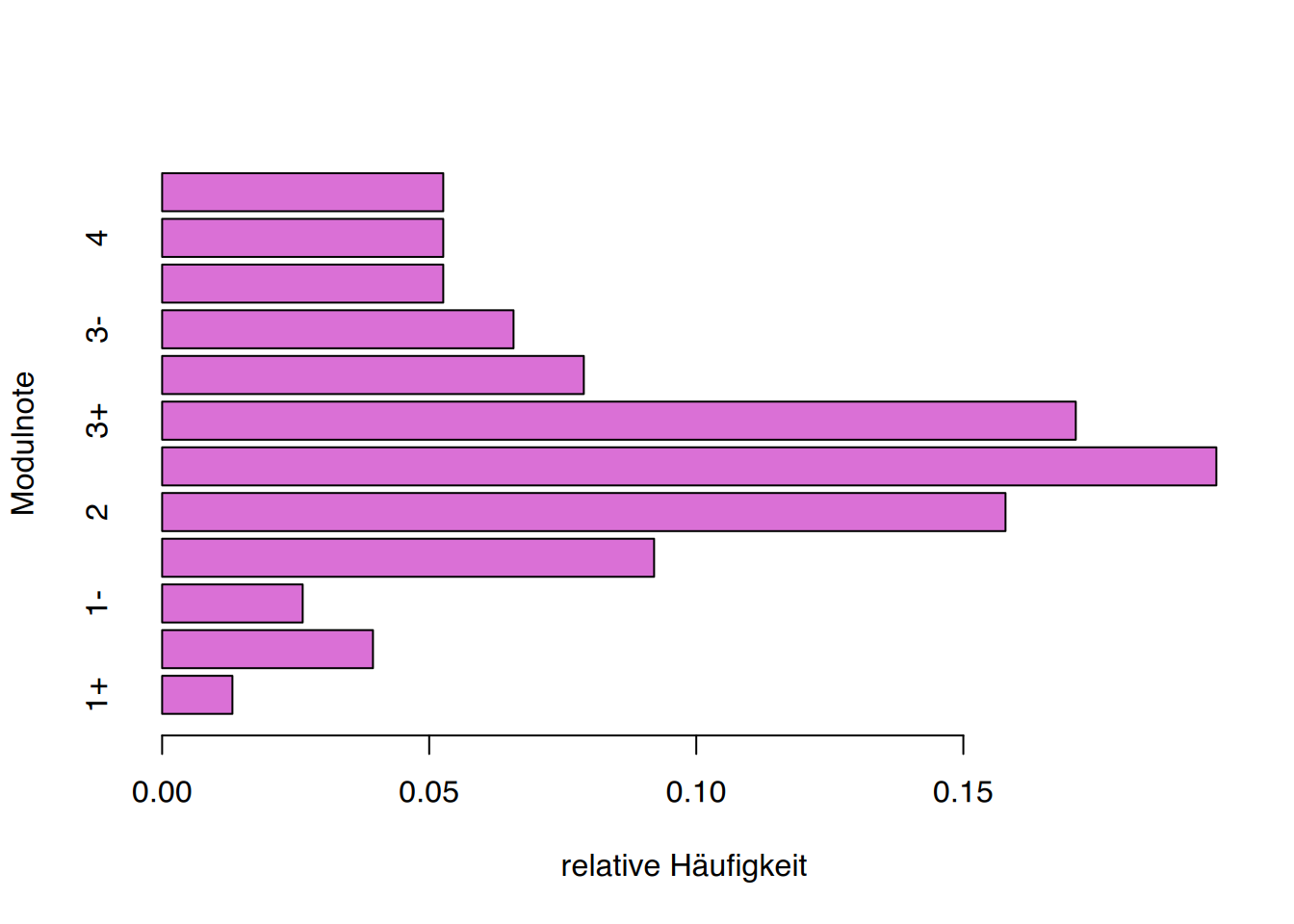
df <- data.frame(Punkte = c(0, 0, 1, 85, 95, 63, 89, 98, 88, 75, 90, 41, 89, 99,
97, 68, 49, 59, 96, 57, 65, 94, 48, 71,96, 72, 98, 88, 66, 58, 43, 66, 76,
98, 44, 74, 99, 86, 87, 97, 99, 86, 61, 41, 77, 73, 71,40, 63, 71, 78, 72,
58, 52, 68, 81, 75, 80, 70, 65, 86, 63, 97, 45, 58, 96, 48, 64, 67, 100,
49, 90, 63, 69, 93, 90, 85, 78, 62, 84, 100, 67, 88, 71, 42, 72, 44, 89,
73, 42, 71, 88, 74,60, 81, 58, 56, 94, 90, 69, 44, 42, 69, 100, 100))Note, indem Sie die erreichten Punkte mit Hilfe der cut()-Funktion in “echte” Noten umwandeln.
Die Schwierigkeit besteht darin, die absoluten Ränder (0 und 100 Punkte) korrekt zu klassieren. Ein möglicher Trick besteht darin, den Parameter right=FALSE zu setzen, und dann die oberste Klassengrenze nicht bei 100, sondern bei 101 aufhören zu lassen. Auf diese Weise werden Studierende, die 100 Punkte erreicht haben, ebenfalls korrekt klassiert.
df$Note <- cut(df$Punkte, breaks=c(0, seq(50, 95, 5), 101),
ordered=TRUE, right=FALSE,
labels = c("5.0", "4.0", "3.7",
"3.3", "3.0", "2.7",
"2.3", "2.0", "1.7",
"1.3", "1.0"))
# anschauen
head(df) Punkte Note
1 0 5.0
2 0 5.0
3 1 5.0
4 85 1.7
5 95 1.0
6 63 3.3jgsbook::freqTable(df$Note) Wert Haeufig Hkum Relativ Rkum
1 5.0 18 18 17.14 17.14
2 4.0 1 19 0.95 18.09
3 3.7 7 26 6.67 24.76
4 3.3 8 34 7.62 32.38
5 3.0 11 45 10.48 42.86
6 2.7 13 58 12.38 55.24
7 2.3 6 64 5.71 60.95
8 2.0 4 68 3.81 64.76
9 1.7 13 81 12.38 77.14
10 1.3 7 88 6.67 83.81
11 1.0 17 105 16.19 100.00hist() die “Originaldaten” erwartet und die Klassierung selbst (nach Ihren Angaben) vornimmt.
Geben Sie dem Plot eine Überschrift und beschriften Sie die Achsen.
hist(df$Punkte, col="orchid",
breaks=c(0, seq(50, 100, 5)),
main="Notenhäufigkeiten", freq = TRUE,
xlab="Note", ylab="Häufigkeiten")
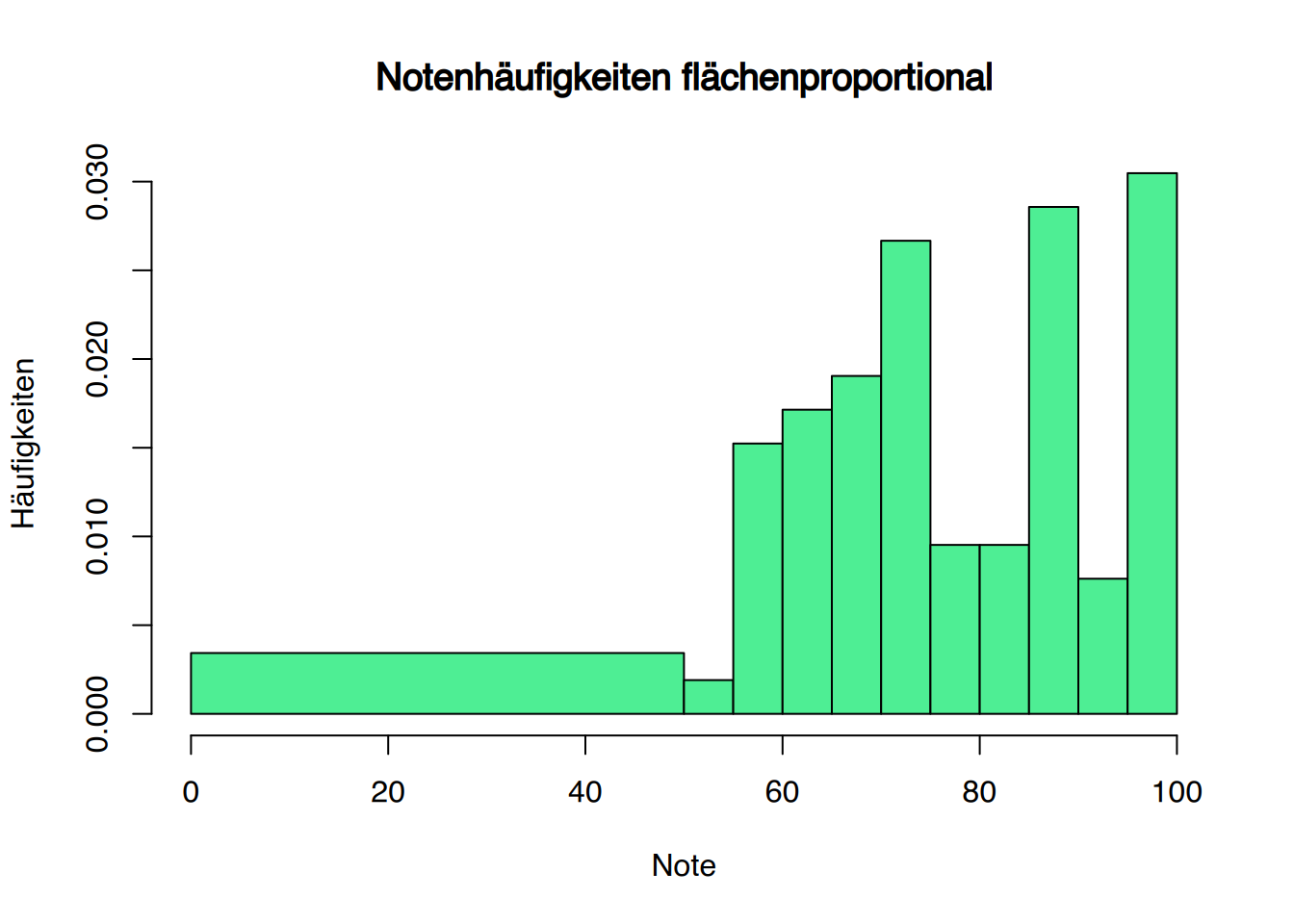
# flächenproportional
hist(df$Punkte, col="seagreen2",
breaks=c(0, seq(50, 100, 5)),
main="Notenhäufigkeiten flächenproportional",
xlab="Note", ylab="Häufigkeiten")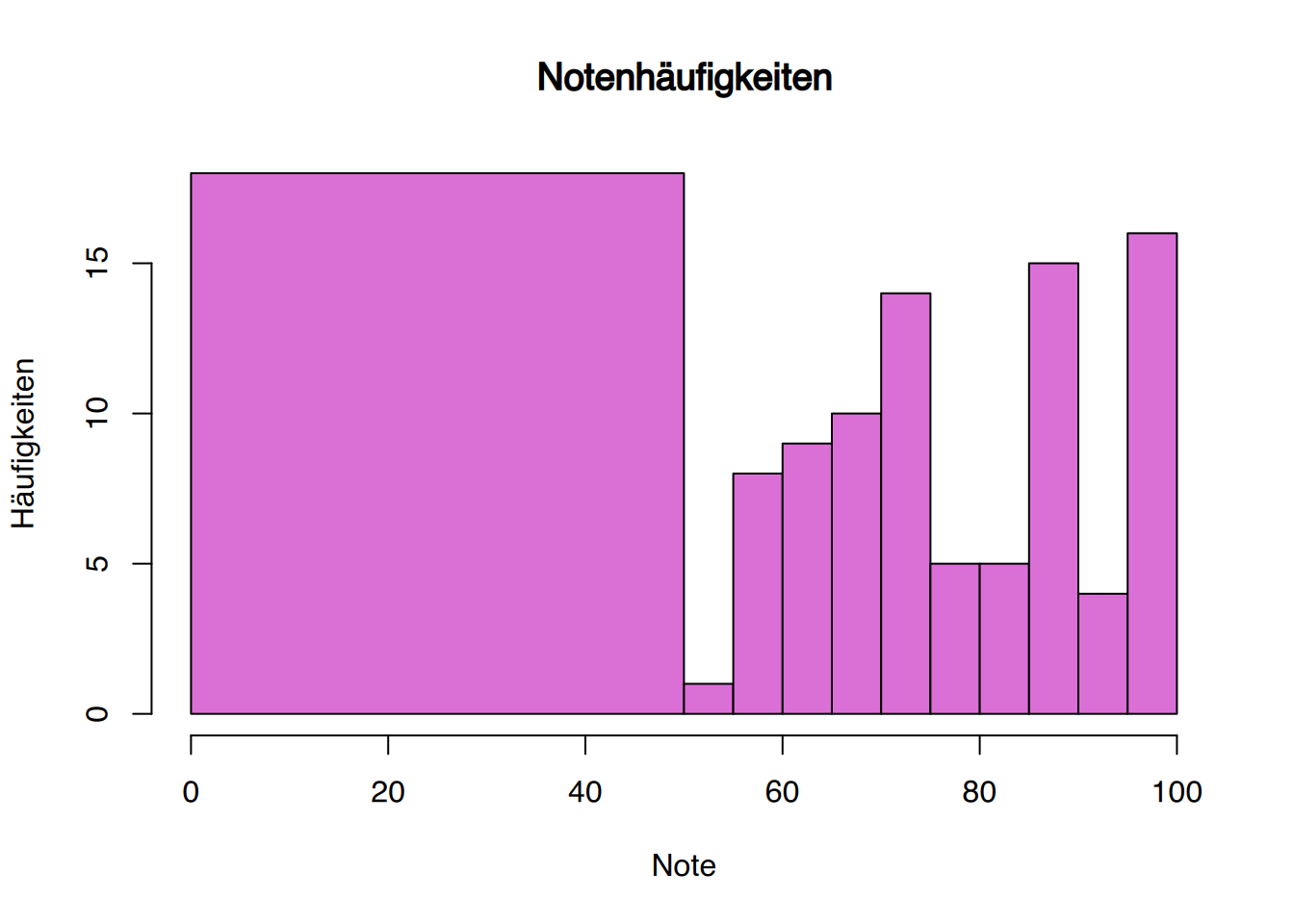
qnorm(0.77, 80, 8)[1] 85.91077qnorm(0.95, 80, 8)[1] 93.15883Bei normalverteilten Werten liegen Median und Mittelwert im selben Punkt. Somit liegt der Median bei 80 Punkten.
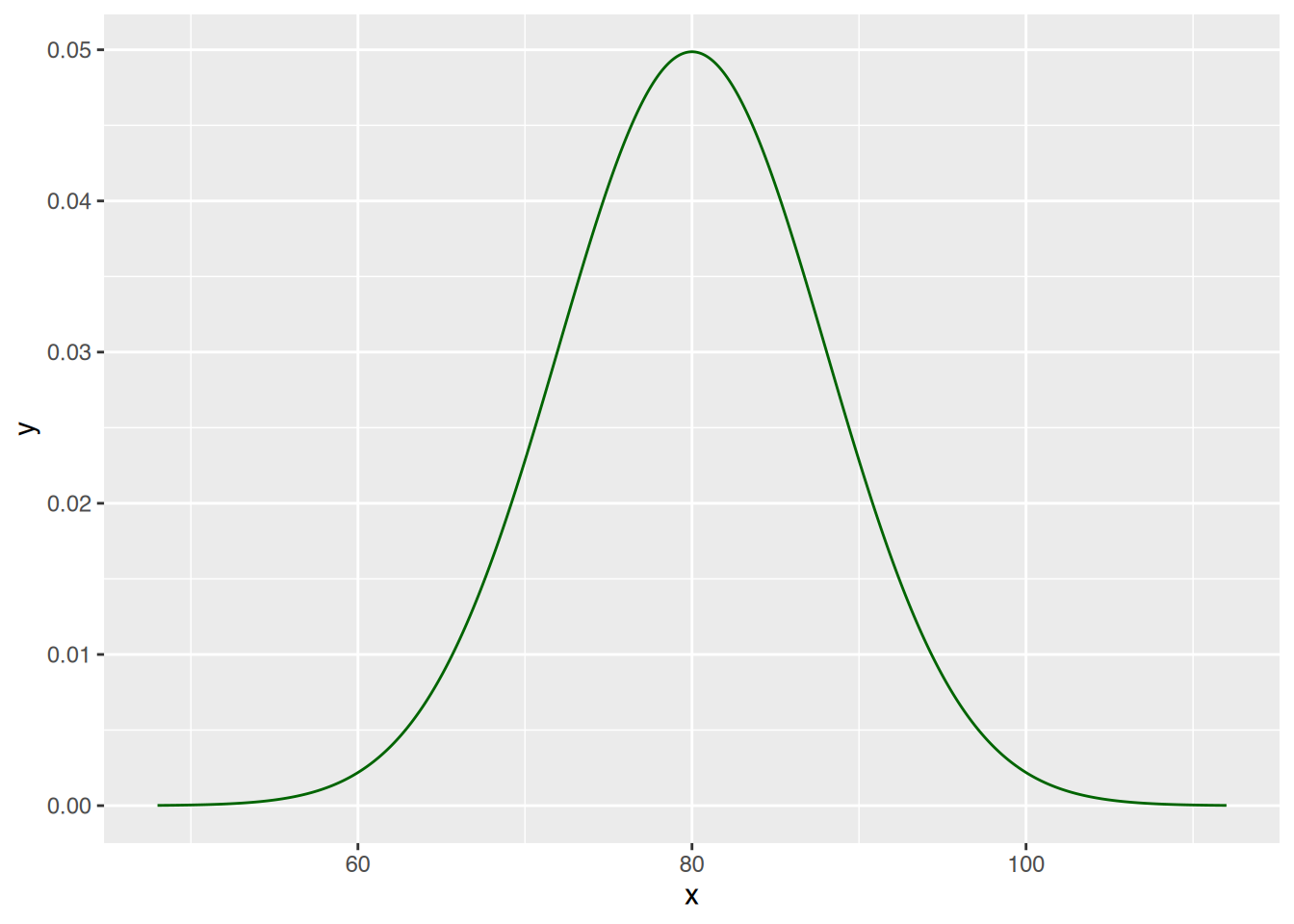
qnorm(0.75, 80, 8)[1] 85.39592# helper
mean <- 80
sd <- 8
# x-Werte erzeugen
x <- seq(mean - 4*sd, mean + 4*sd, by=0.1)
# y-Werte
y <- dnorm(x, mean, sd)
# mit R-base
plot(x, y, type="l", col="darkgreen")
# mit ggplot
library(ggplot2)
df <- data.frame(x, y)
ggplot(df, aes(x=x, y=y)) +
geom_line(color="darkgreen")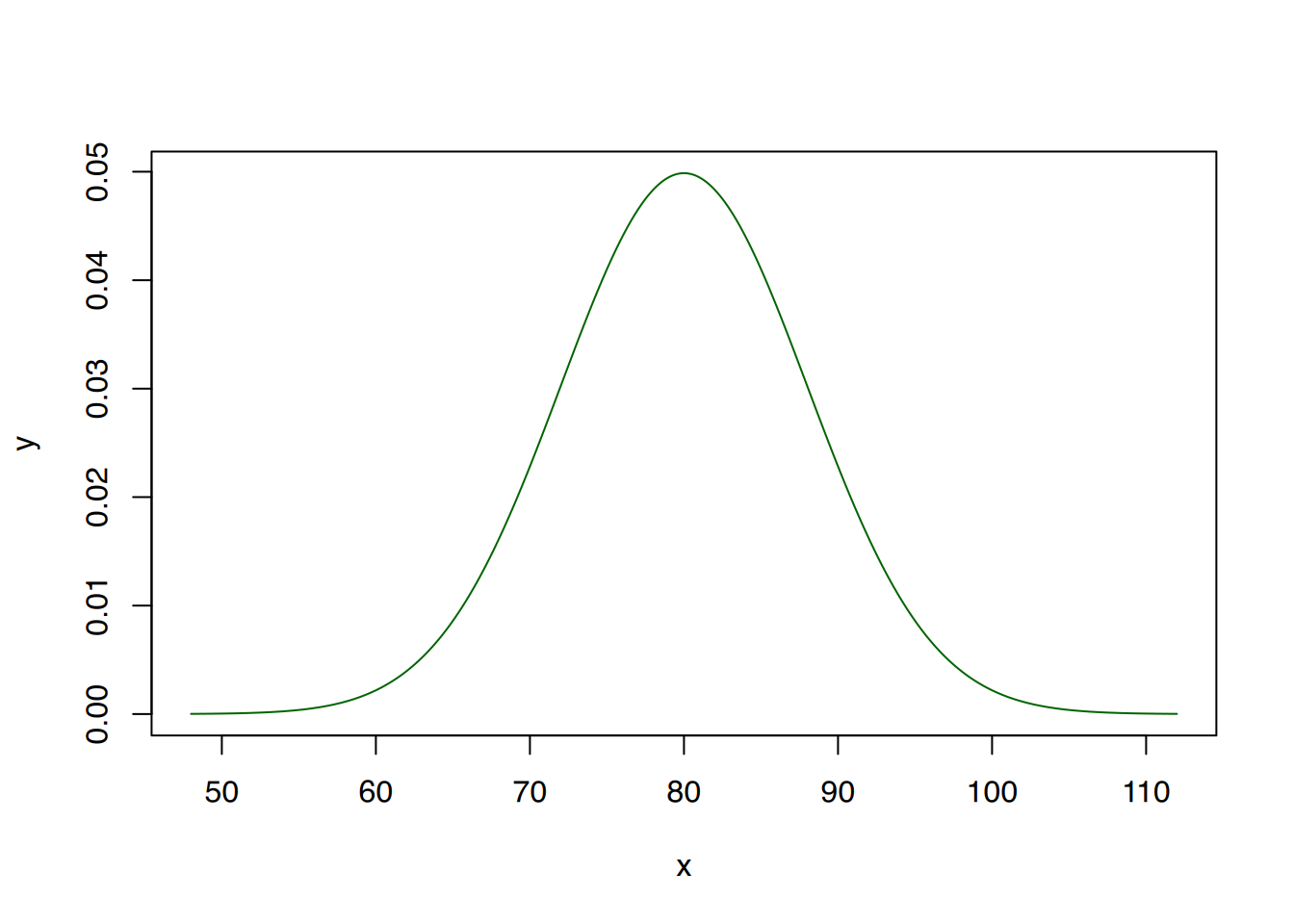
# z-Wert von Tina
as.numeric(scale(105, center=110, scale=17))[1] -0.2941176Tinas z-Wert lautet -0,2941.
# schneller bedeutet weniger Sekunden
pnorm(105, mean=110, s=17)[1] 0.384334Es sind 38,43% der Kolleginnen schneller als Tina.
1 - pnorm(105, mean=110, s=17)[1] 0.615666Es sind 61,57% der Kolleginnen schneller als Tina
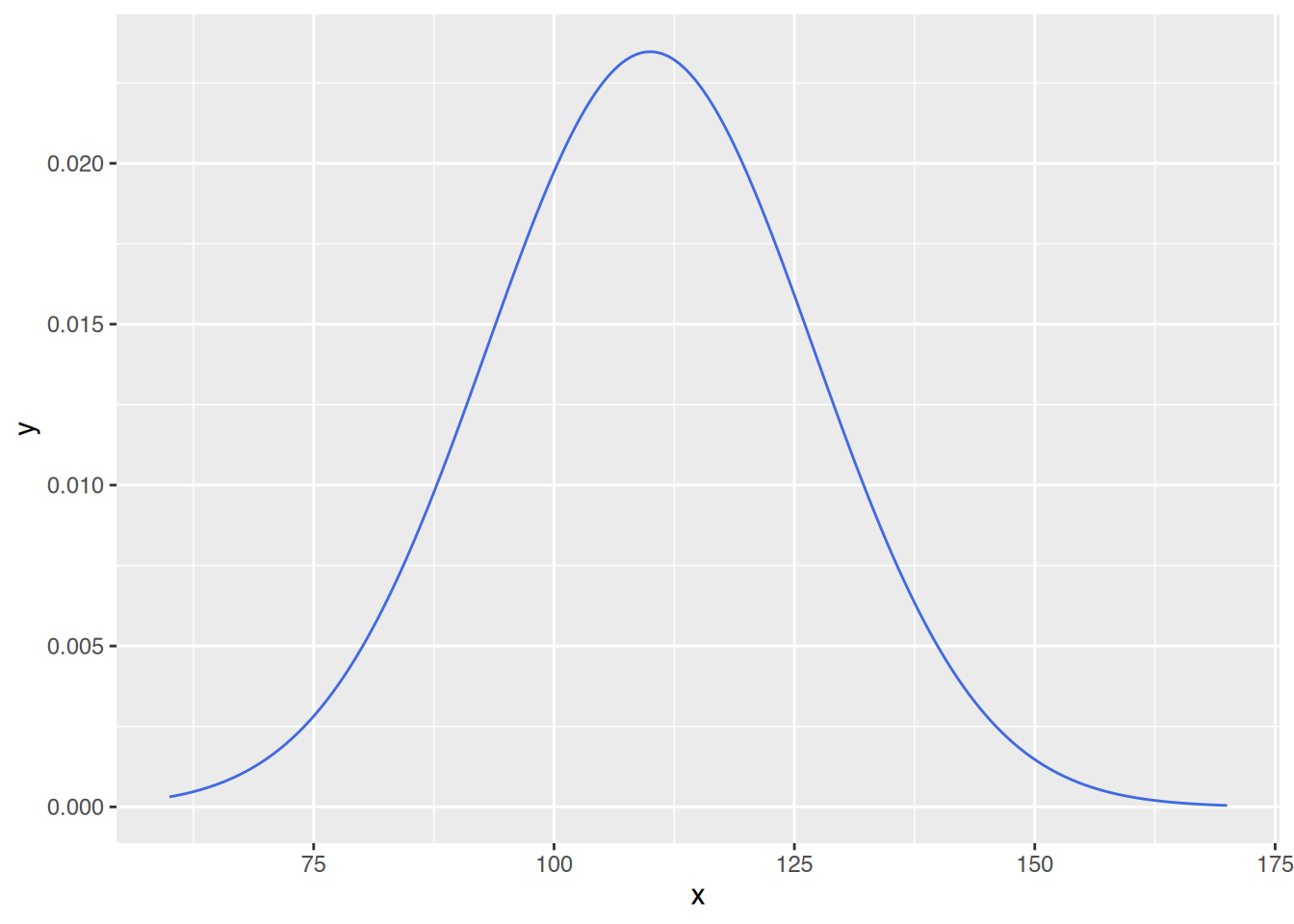
# x-Werte von 60 bis 170 erzeugen
x <- seq(60, 170, by=0.1)
# y-Werte mittels dnorm erzeugen
y <- dnorm(x, mean=110, sd=17)
# plotten mit Rbase
plot(x, y, col="royalblue")
# plotten mit ggplot
df <- data.frame(x, y)
ggplot(df, aes(x=x, y=y)) +
geom_line(color="royalblue")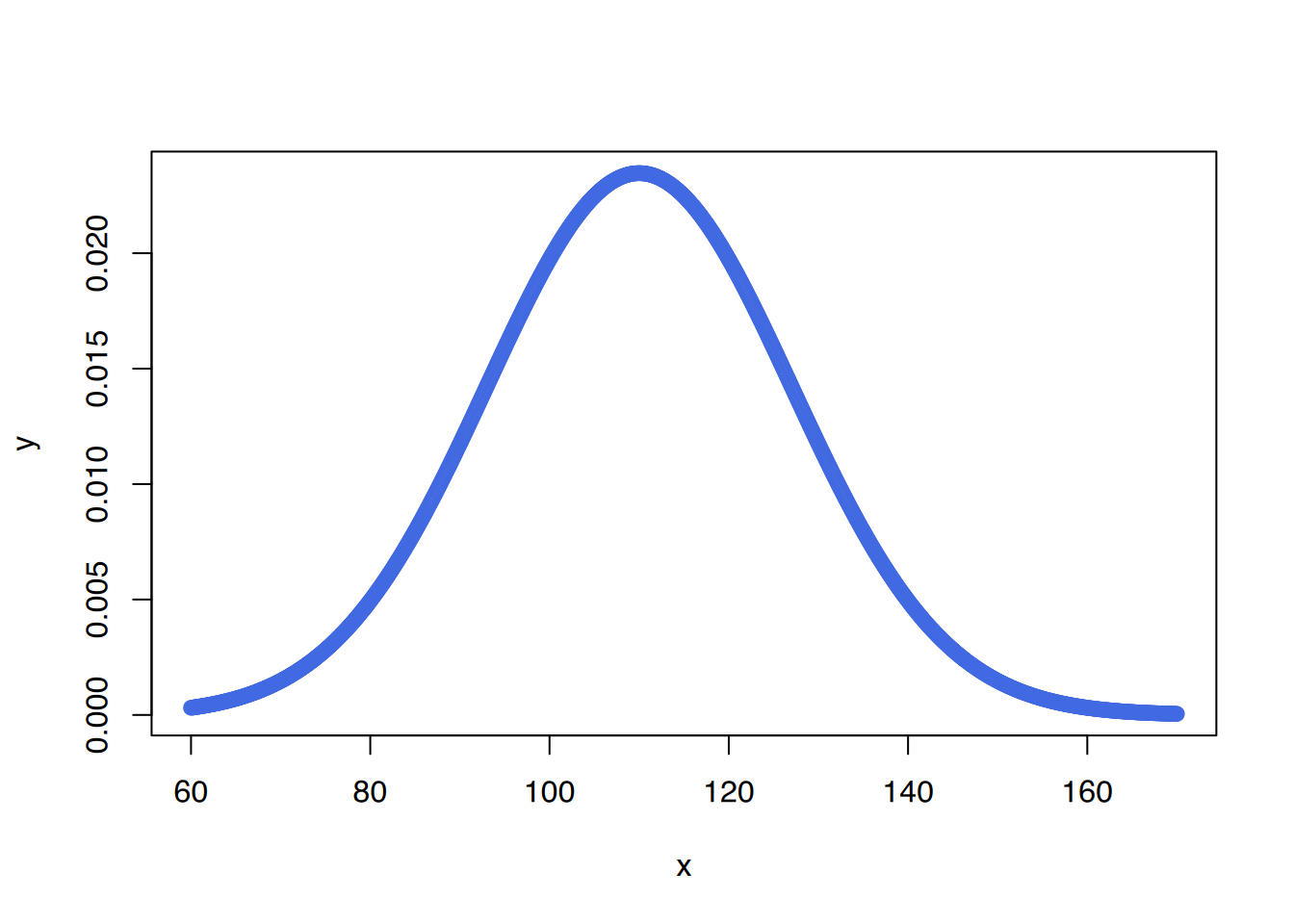
R und passen Sie wo notwendig das Skalenniveau an.
library(tidyverse)
# Daten übertragen, z.B. mittels tribble()
df <- tribble(
~Proband, ~Partys, ~bestanden,
1,2,3,
2,3,3,
3,6,1,
4,4,0,
5,0,5,
6,1,3,
7,4,0,
8,0,4,
9,8,1,
10,4,2
)
# Skalenniveau anpassen
df <- mutate(df, Proband=factor(Proband))
# anzeigen
glimpse(df)Rows: 10
Columns: 3
$ Proband <fct> 1, 2, 3, 4, 5, 6, 7, 8, 9, 10
$ Partys <dbl> 2, 3, 6, 4, 0, 1, 4, 0, 8, 4
$ bestanden <dbl> 3, 3, 1, 0, 5, 3, 0, 4, 1, 2cor(df$Partys, df$bestanden)[1] -0.7782703Der Korrelationskoeffizient liegt bei -0,7783. Das spricht für einen starken negativen Zusammenhang.
bestanden erklärt durch Partys durch. Wie stark ist der Zusammenhang?
# Regression durchführen
fit<- lm(bestanden~Partys, data=df)
# Ergebnisse anzeigen
summary(fit)
Call:
lm(formula = bestanden ~ Partys, data = df)
Residuals:
Min 1Q Median 3Q Max
-1.7919 -0.1997 0.1980 0.5805 1.2483
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.8322 0.5856 6.544 0.00018 ***
Partys -0.5101 0.1455 -3.506 0.00801 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.123 on 8 degrees of freedom
Multiple R-squared: 0.6057, Adjusted R-squared: 0.5564
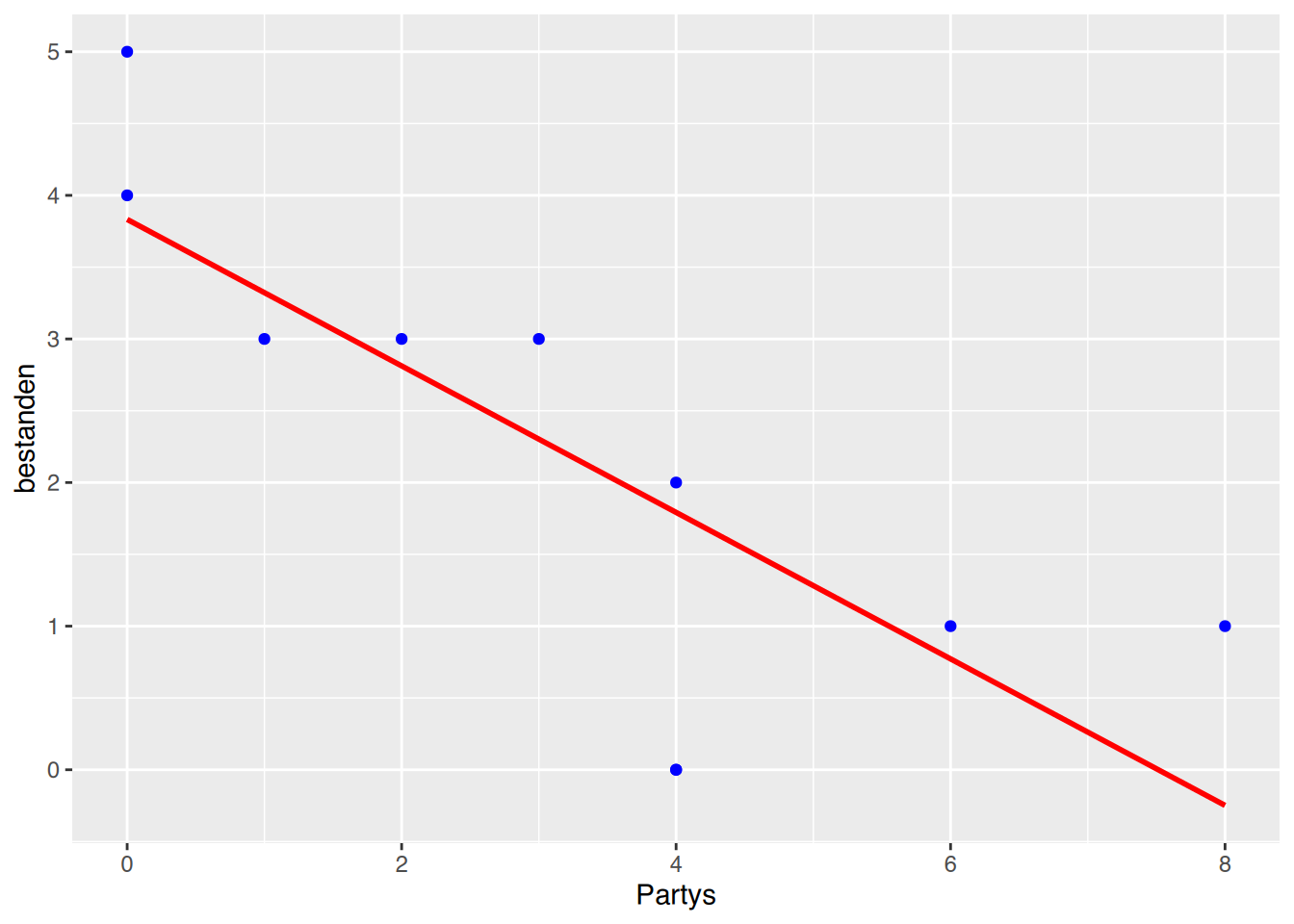
F-statistic: 12.29 on 1 and 8 DF, p-value: 0.008013Der Koeffizient (die Steigung der Geraden) beträgt -0,5101. Mit jeder Party werden 0,5101 Prüfungen weniger bestanden.
# mit Rbase plotten
plot(df$Partys, df$bestanden, col="blue",
xlab="Anzahl besuchter Partys",
ylab="Anzahl bestandener Prüfungen")
# Regressionsgerade
abline(lm(bestanden~Partys, data=df), col="red")
# mit ggplot
ggplot(df, aes(x=Partys, y=bestanden)) +
geom_point(color="blue") +
# Regressionsgerade
geom_smooth(method="lm", se=FALSE, color="red")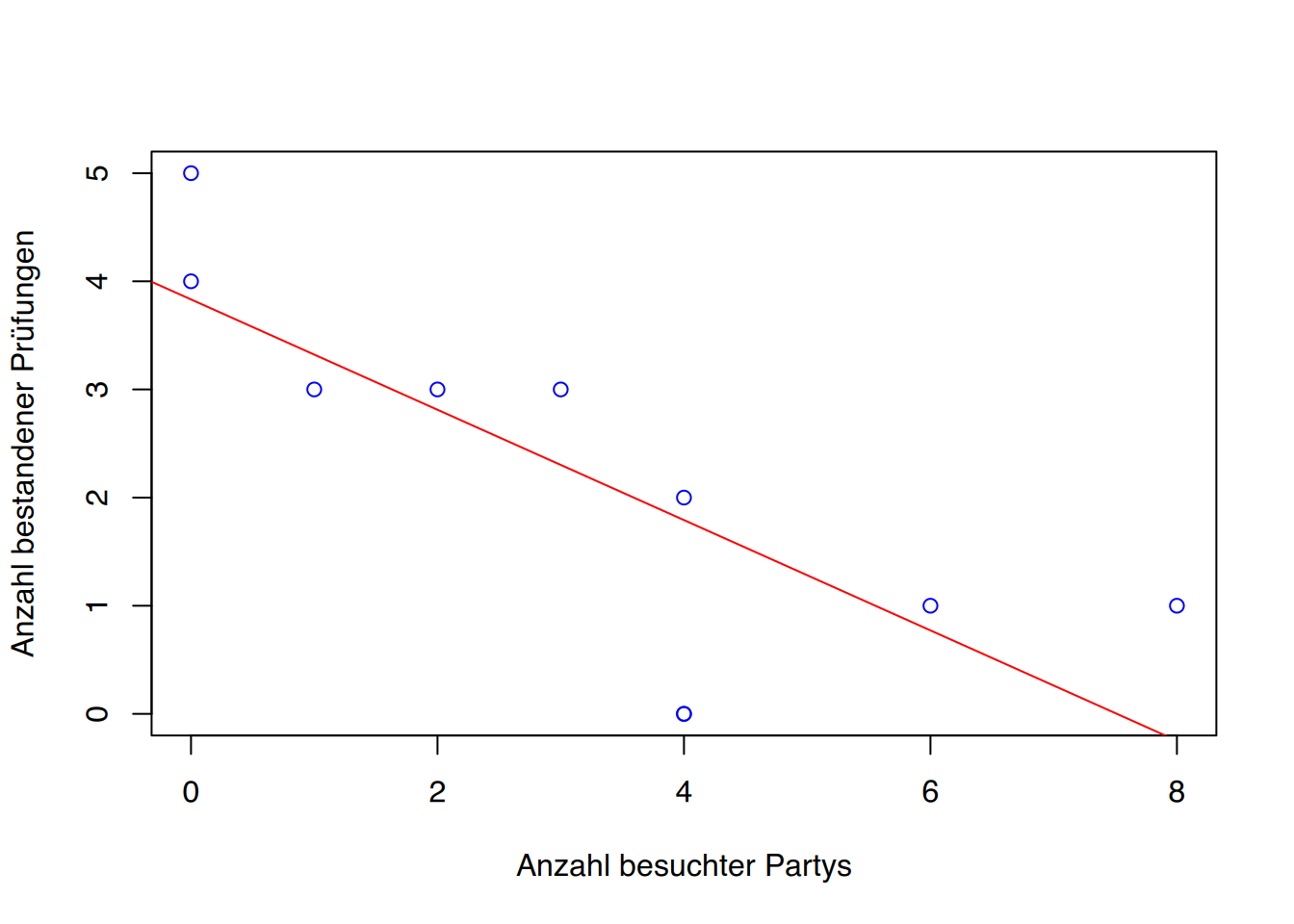
# neuer Proband als eigenes tibble/data.frame
new <- tibble(Proband=factor(11),
Partys=0,
bestanden=0)
# mittels rbind() zusammenführen
df <- rbind(df, new)
# anzeigen
df# A tibble: 11 × 3
Proband Partys bestanden
<fct> <dbl> <dbl>
1 1 2 3
2 2 3 3
3 3 6 1
4 4 4 0
5 5 0 5
6 6 1 3
7 7 4 0
8 8 0 4
9 9 8 1
10 10 4 2
11 11 0 0# Korrelation
cor(df$Partys, df$bestanden)[1] -0.5278522Der Zusammenhang wird schwächer, ist aber immernoch leicht größer als 0,5.
# Regression erneut durchführen
fit<- lm(bestanden~Partys, data=df)
# Ergebnisse anzeigen
summary(fit)
Call:
lm(formula = bestanden ~ Partys, data = df)
Residuals:
Min 1Q Median 3Q Max
-3.0132 -0.7718 0.3800 0.8800 1.9868
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 3.0132 0.7169 4.203 0.0023 **
Partys -0.3483 0.1868 -1.864 0.0951 .
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.551 on 9 degrees of freedom
Multiple R-squared: 0.2786, Adjusted R-squared: 0.1985
F-statistic: 3.476 on 1 and 9 DF, p-value: 0.09513Das Bestimmtheitsmaß R^2 beträgt 0,1985. Das bedeutet, dass nur noch 19,85% des Rauschens erklärt werden können. Ohne den letzten Studenten lag es bei 55,64% (siehe oben unter c)).
plot(df$Partys, df$bestanden, col="blue",
xlab="Anzahl besuchter Partys",
ylab="Anzahl bestandener Prüfungen")
# Regressionsgerade
abline(lm(bestanden~Partys, data=df), col="red")
# mit ggplot
ggplot(df, aes(x=Partys, y=bestanden)) +
geom_point(color="blue") +
# Regressionsgerade
geom_smooth(method="lm", se=FALSE, color="red")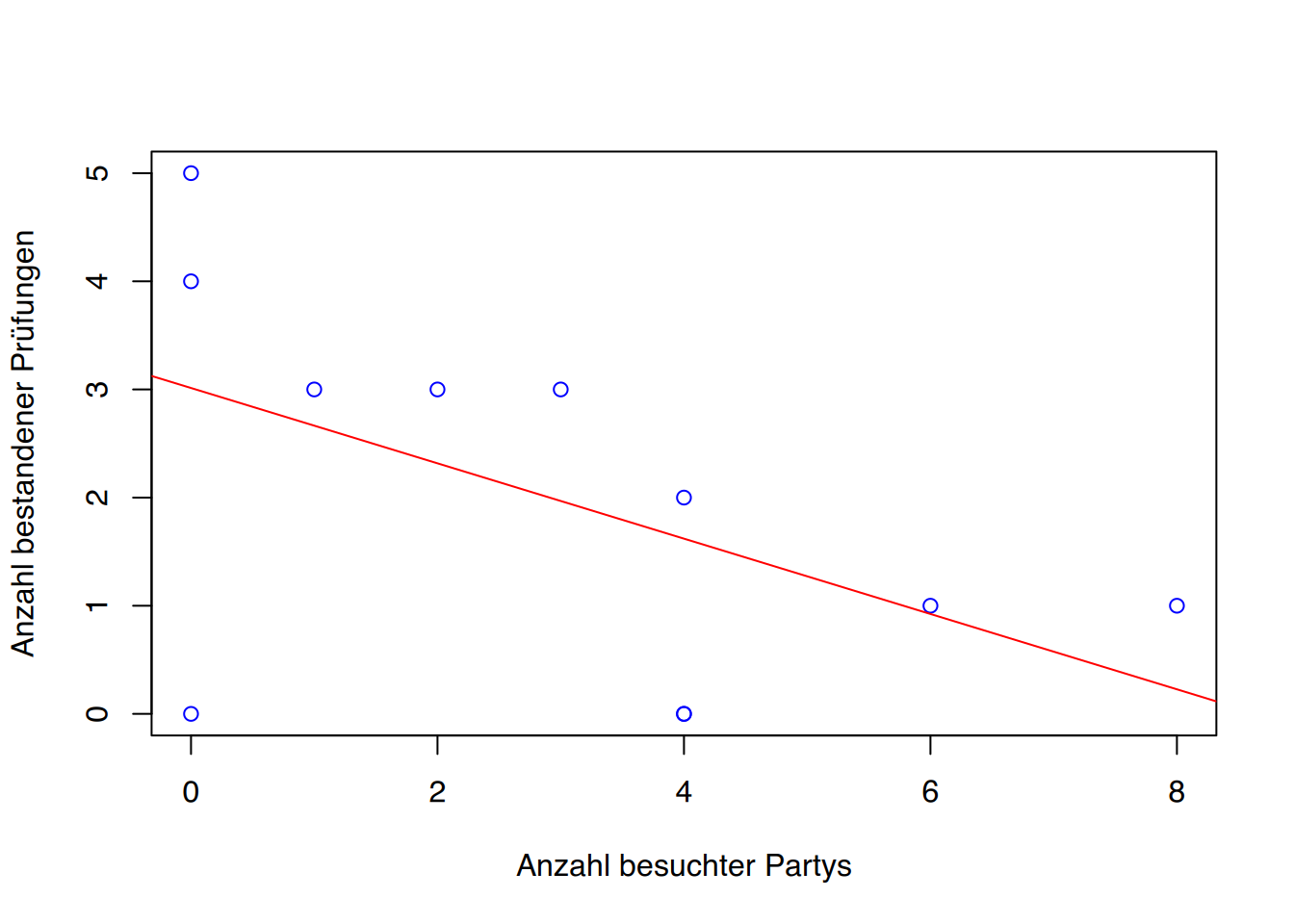
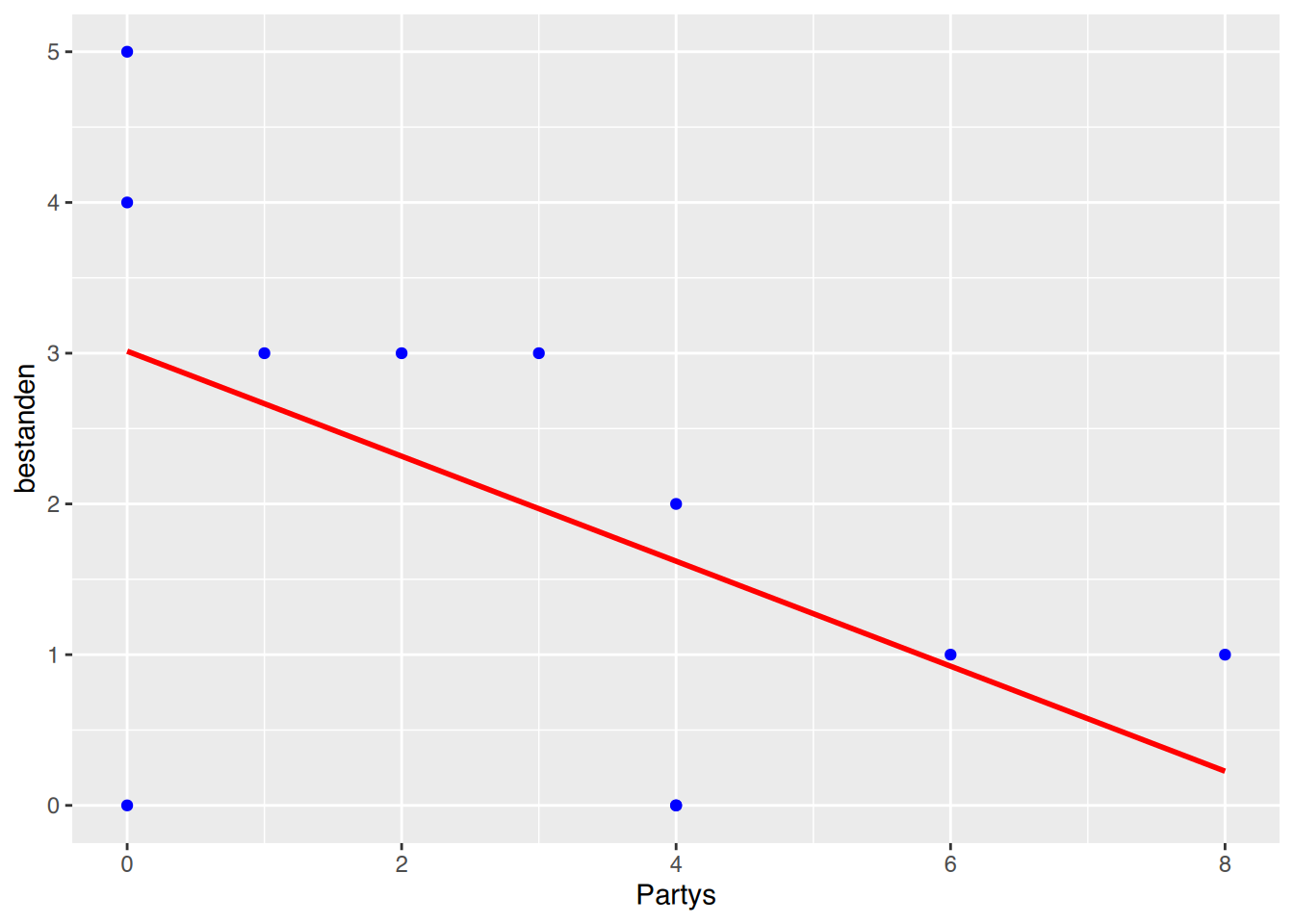
Die Regressionsgerade wird flacher, die Steigung beträgt nur noch -0.3483.