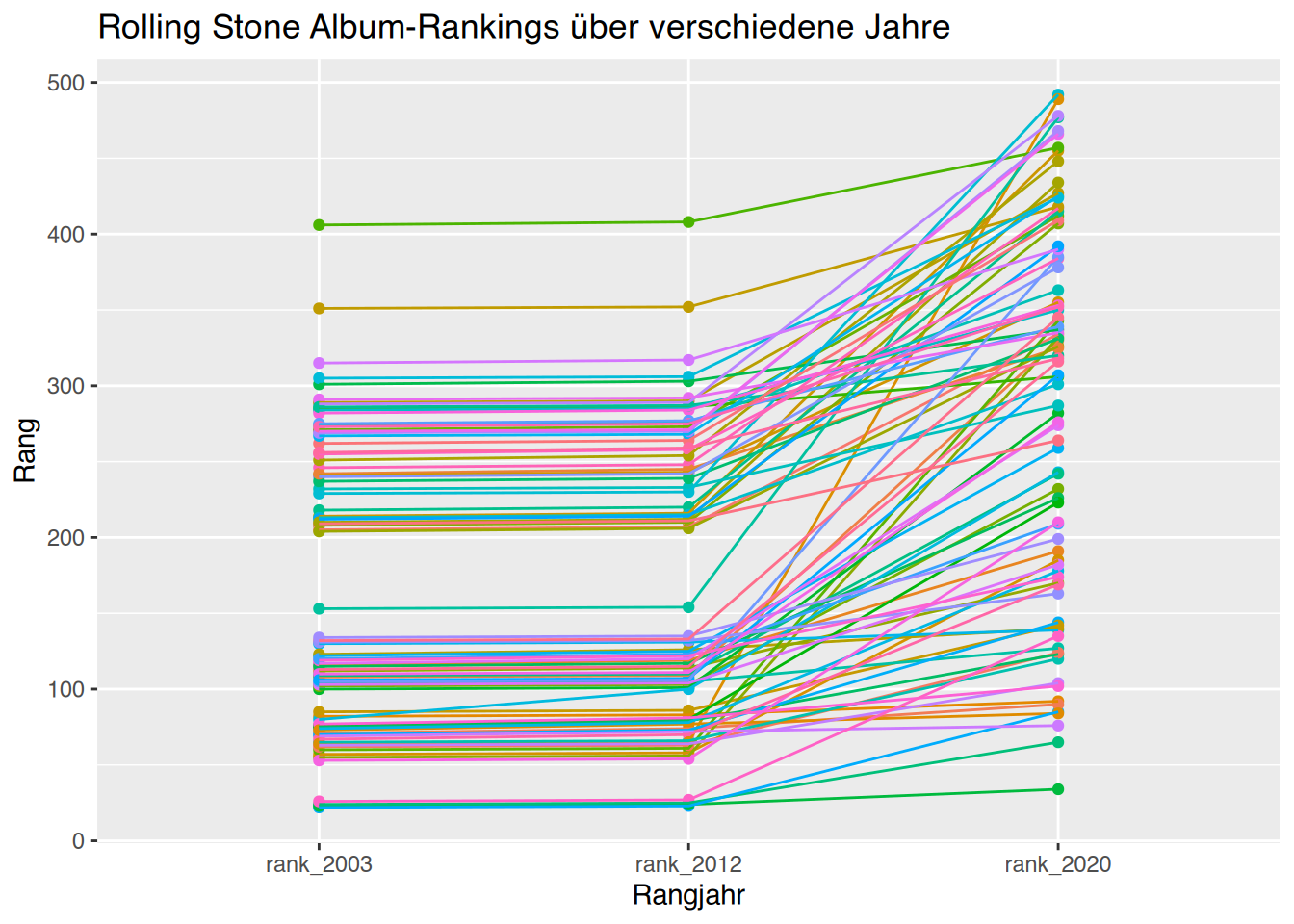
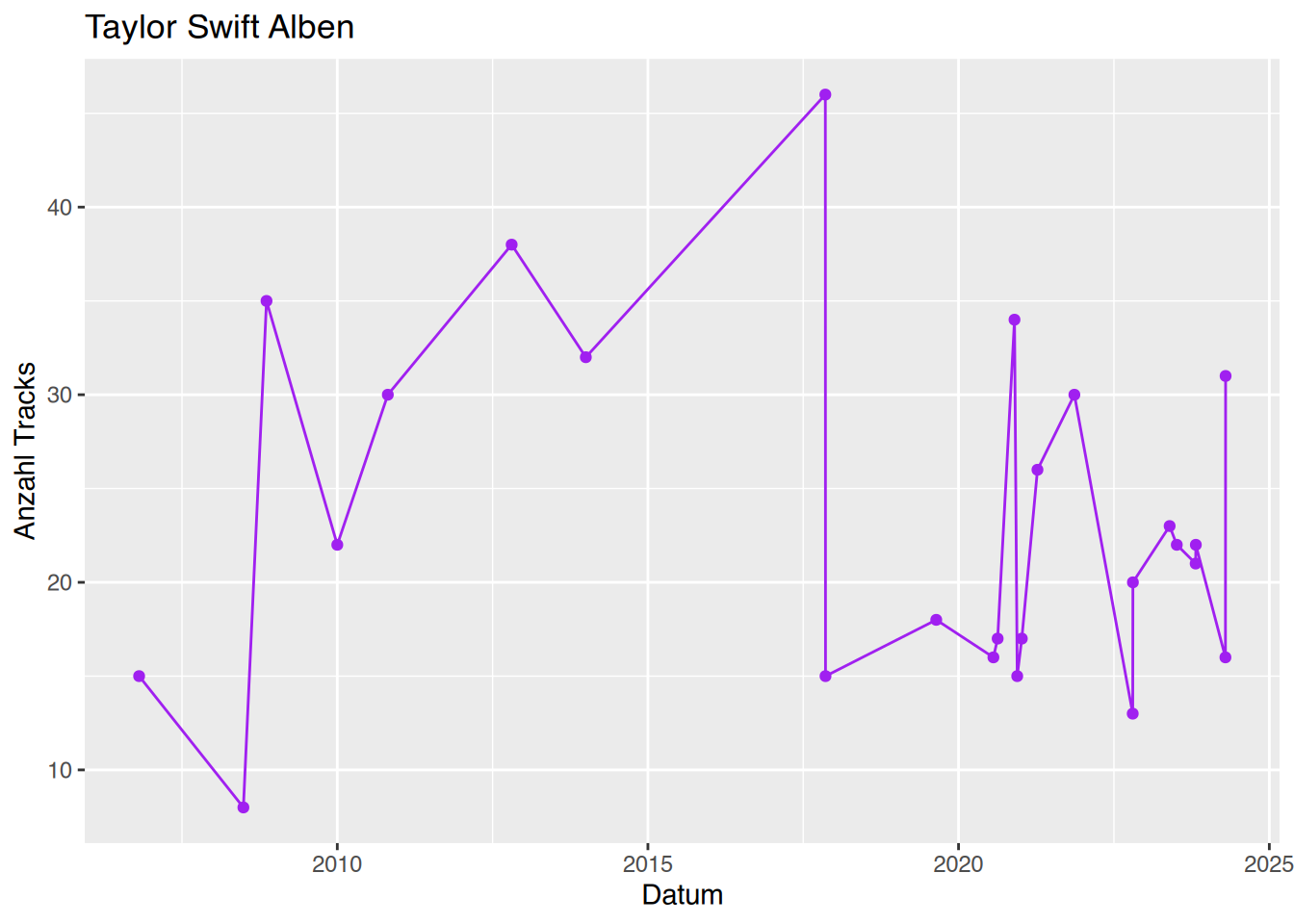
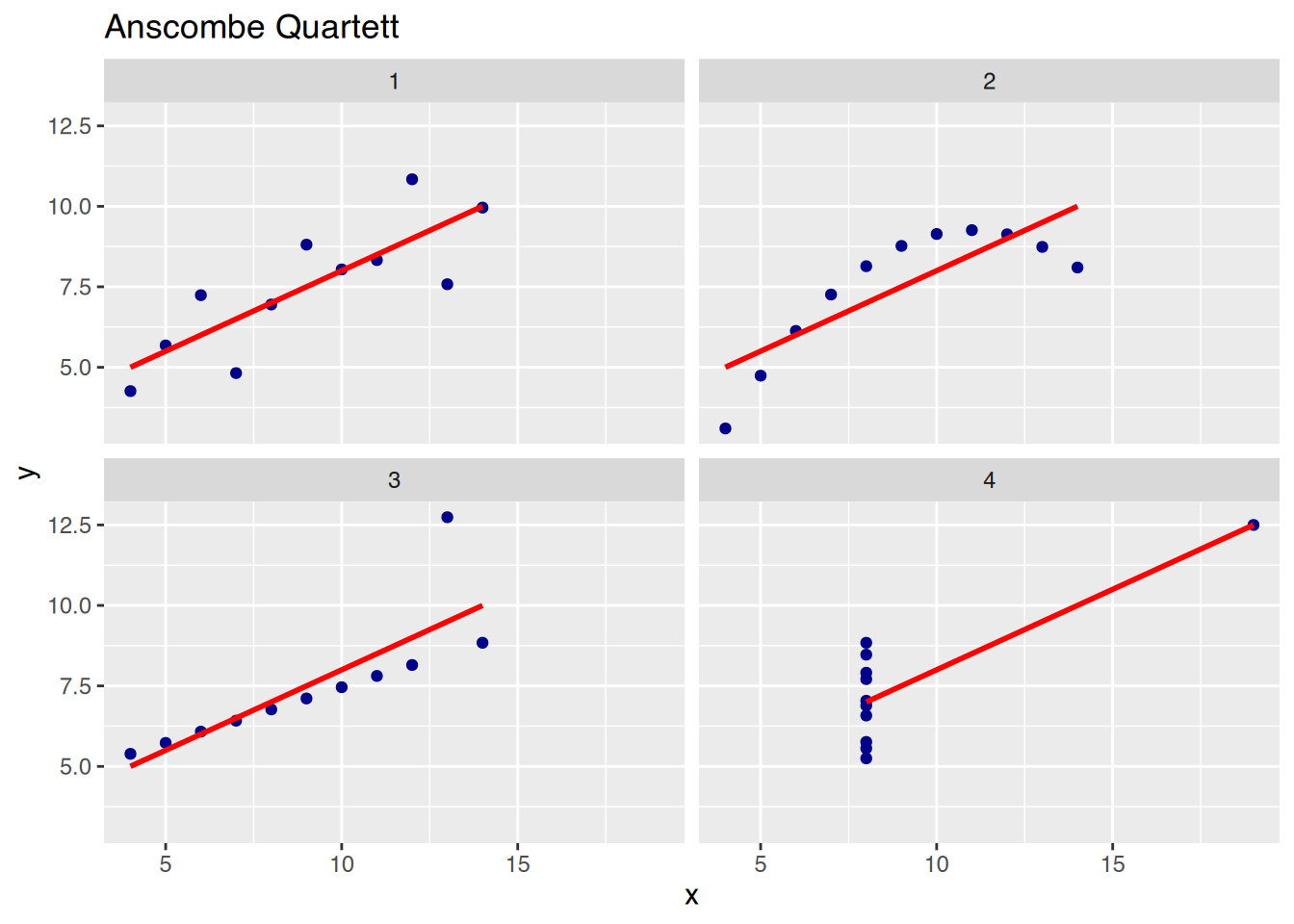
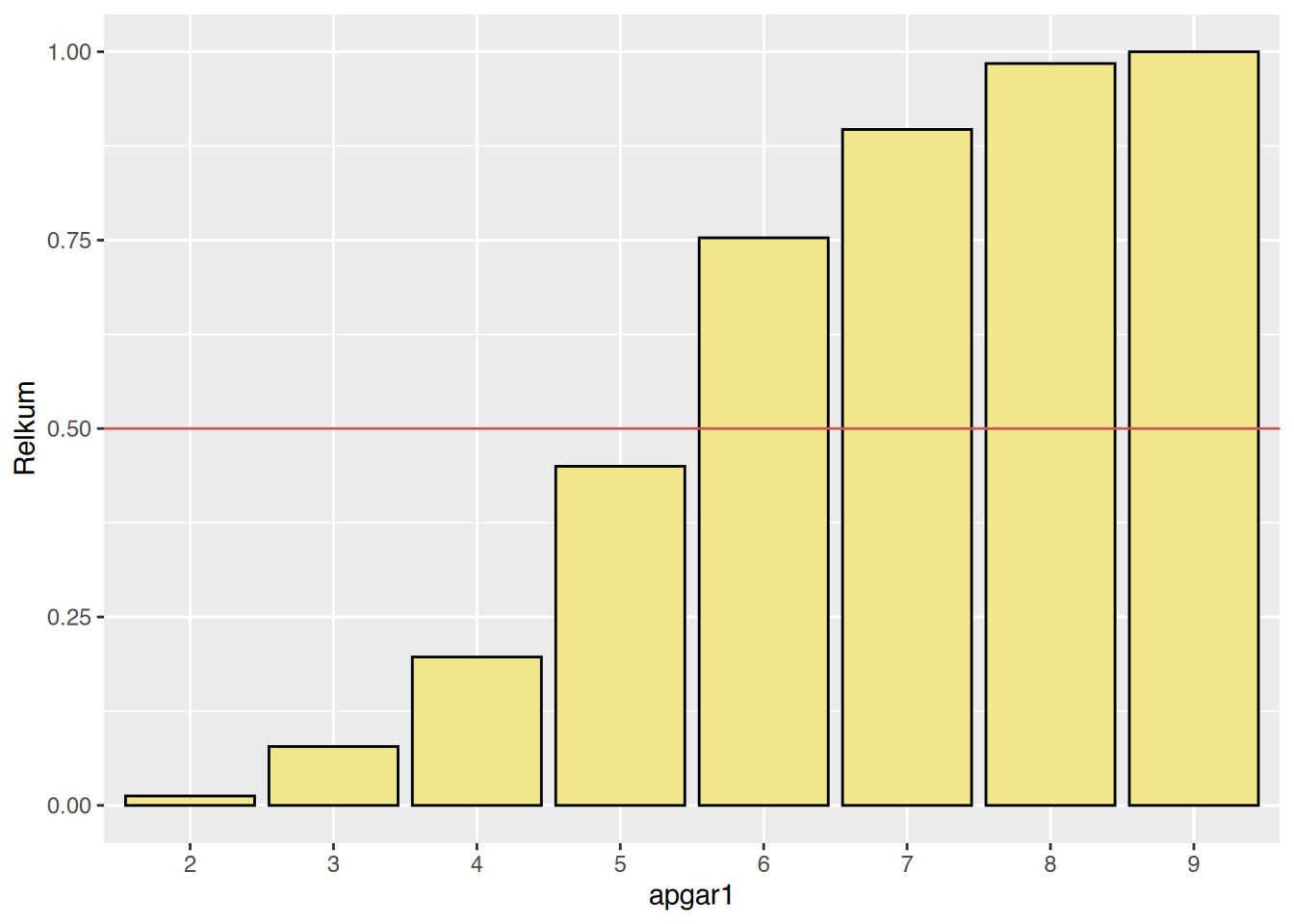
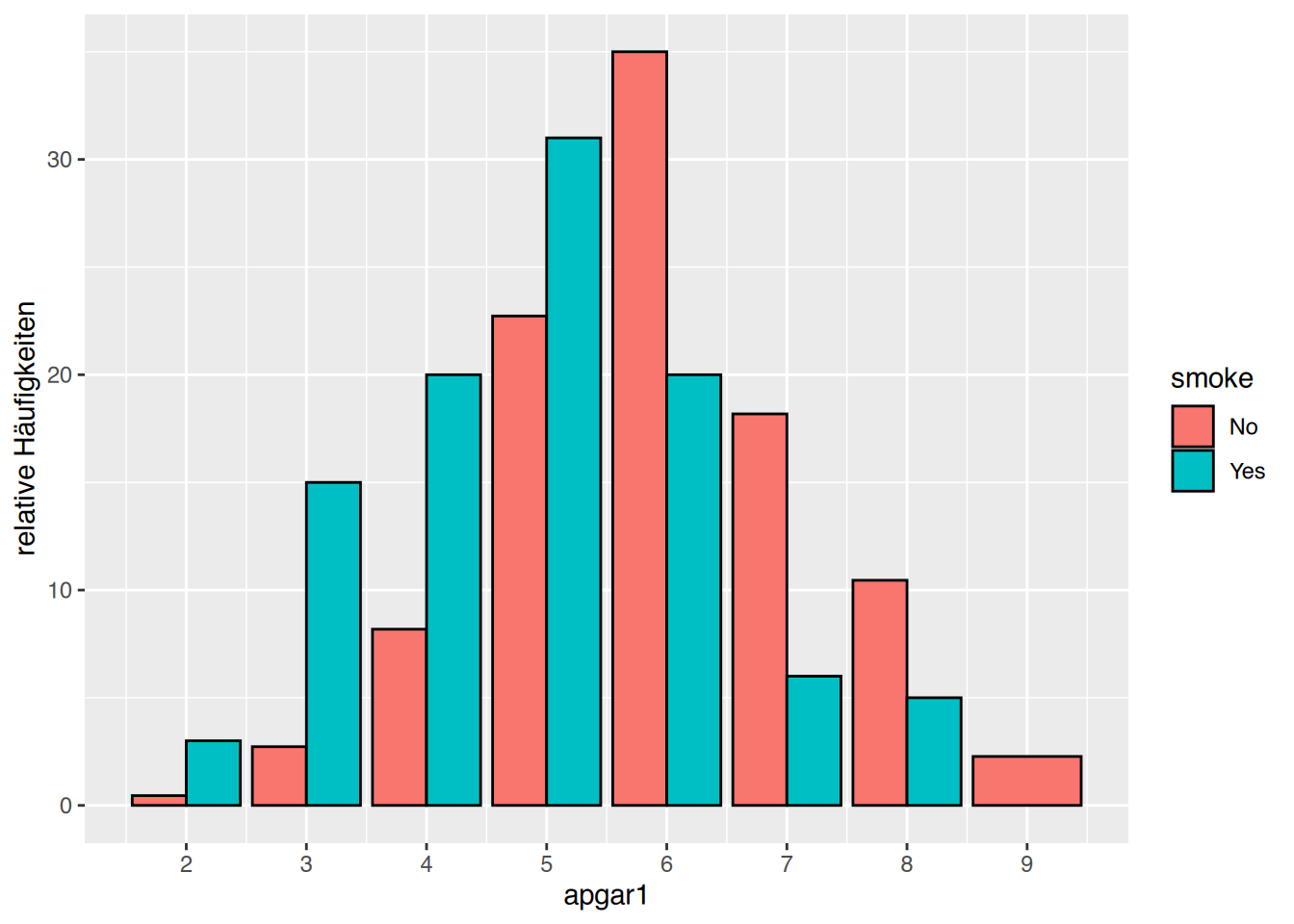
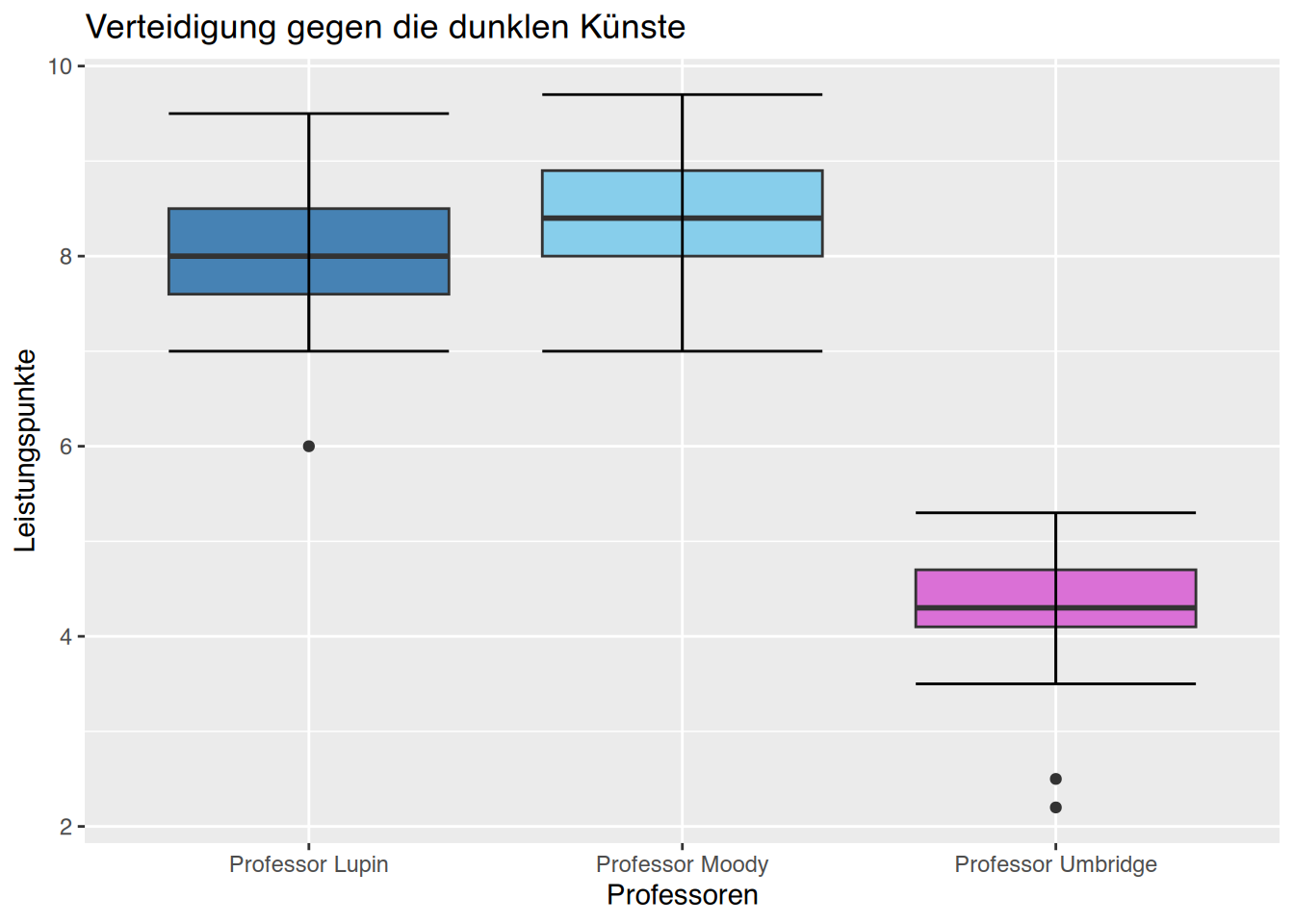
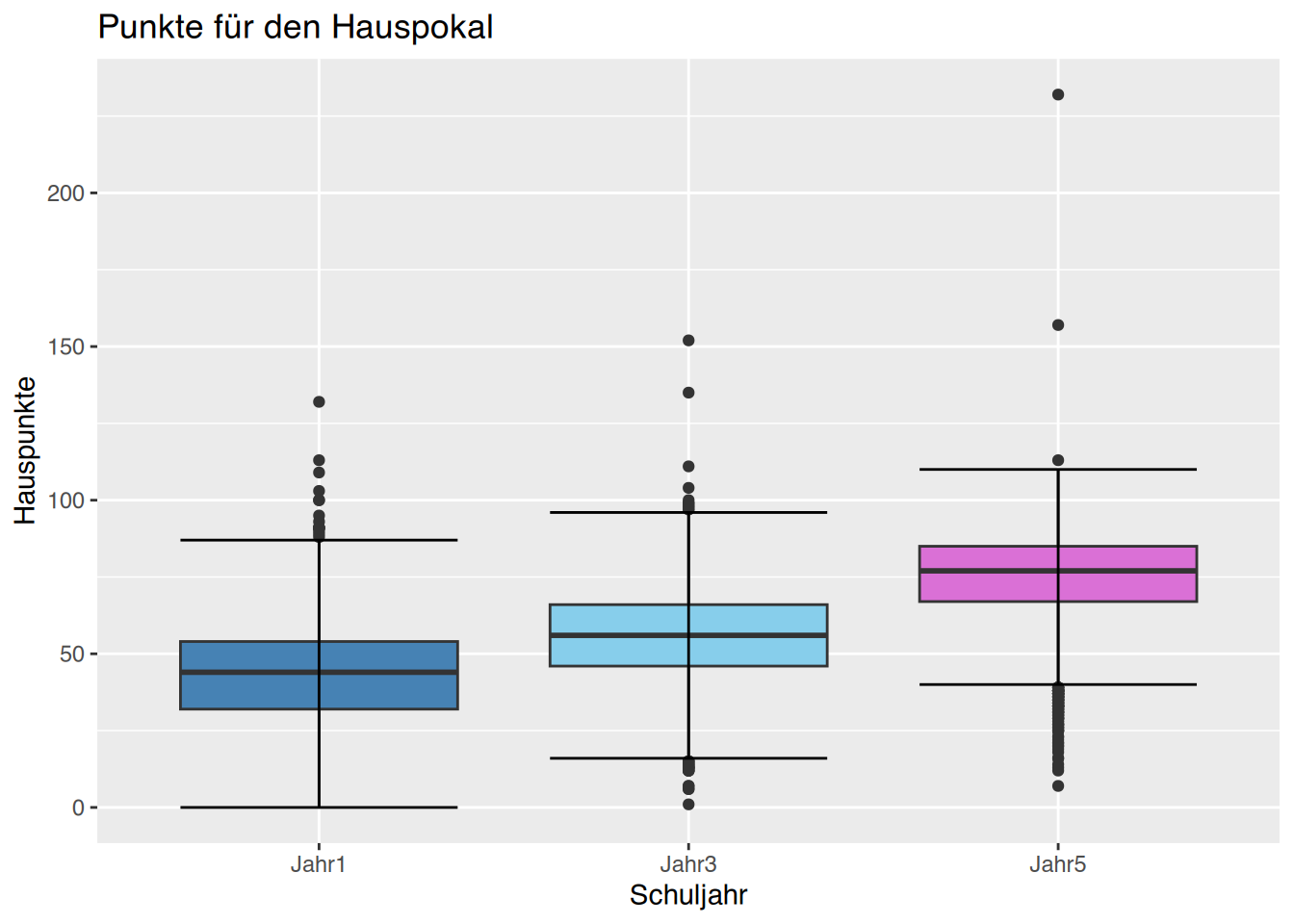
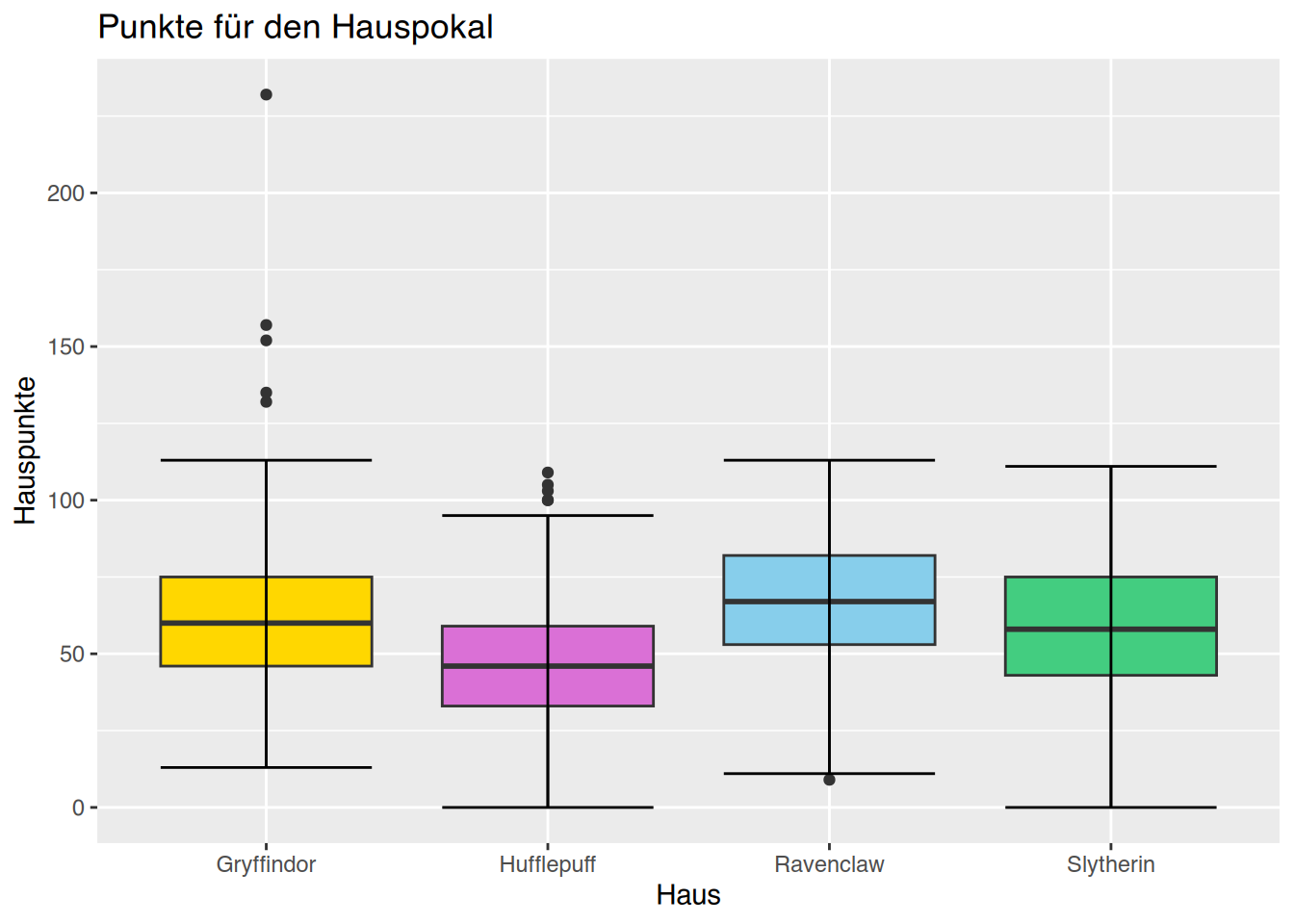
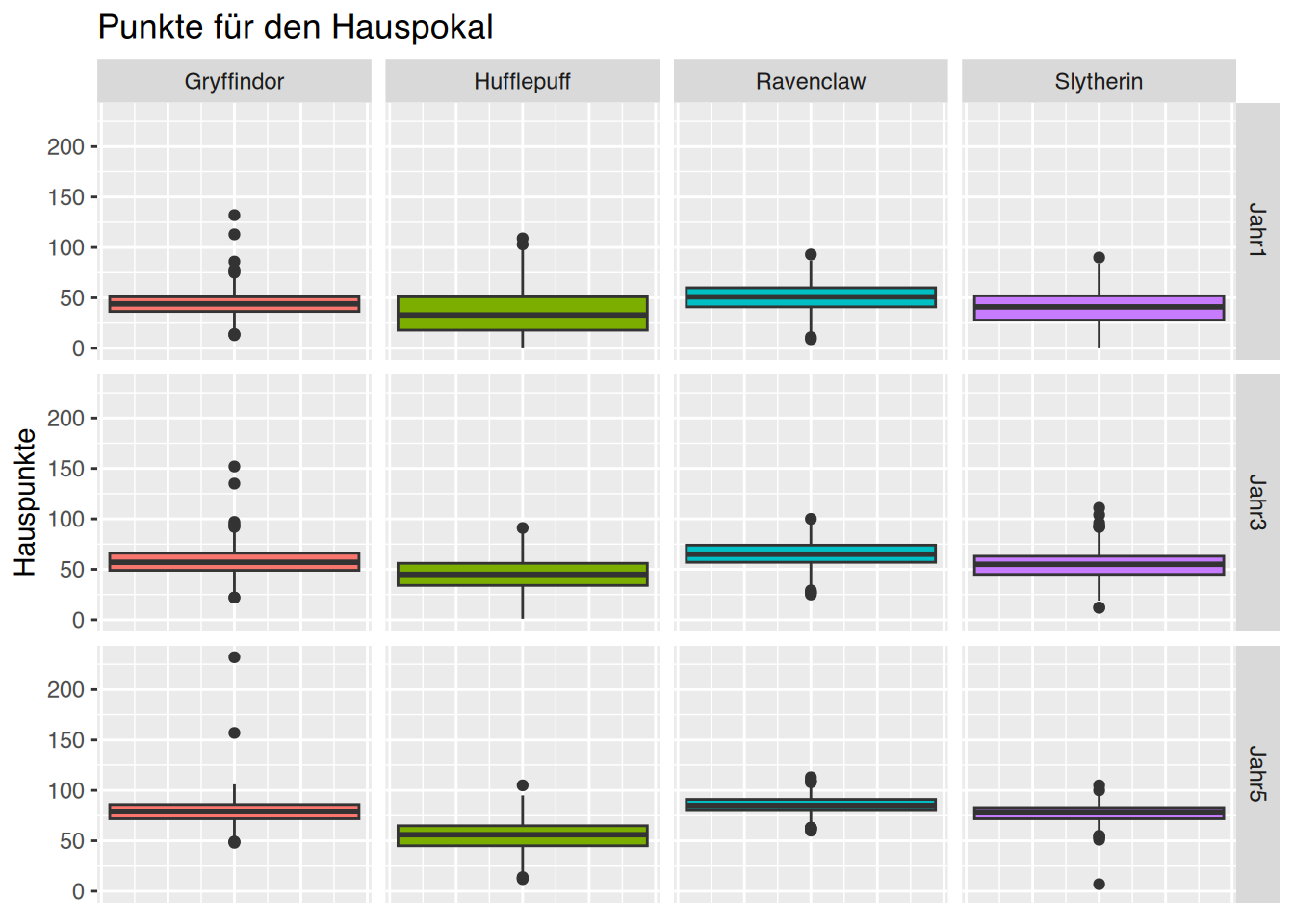
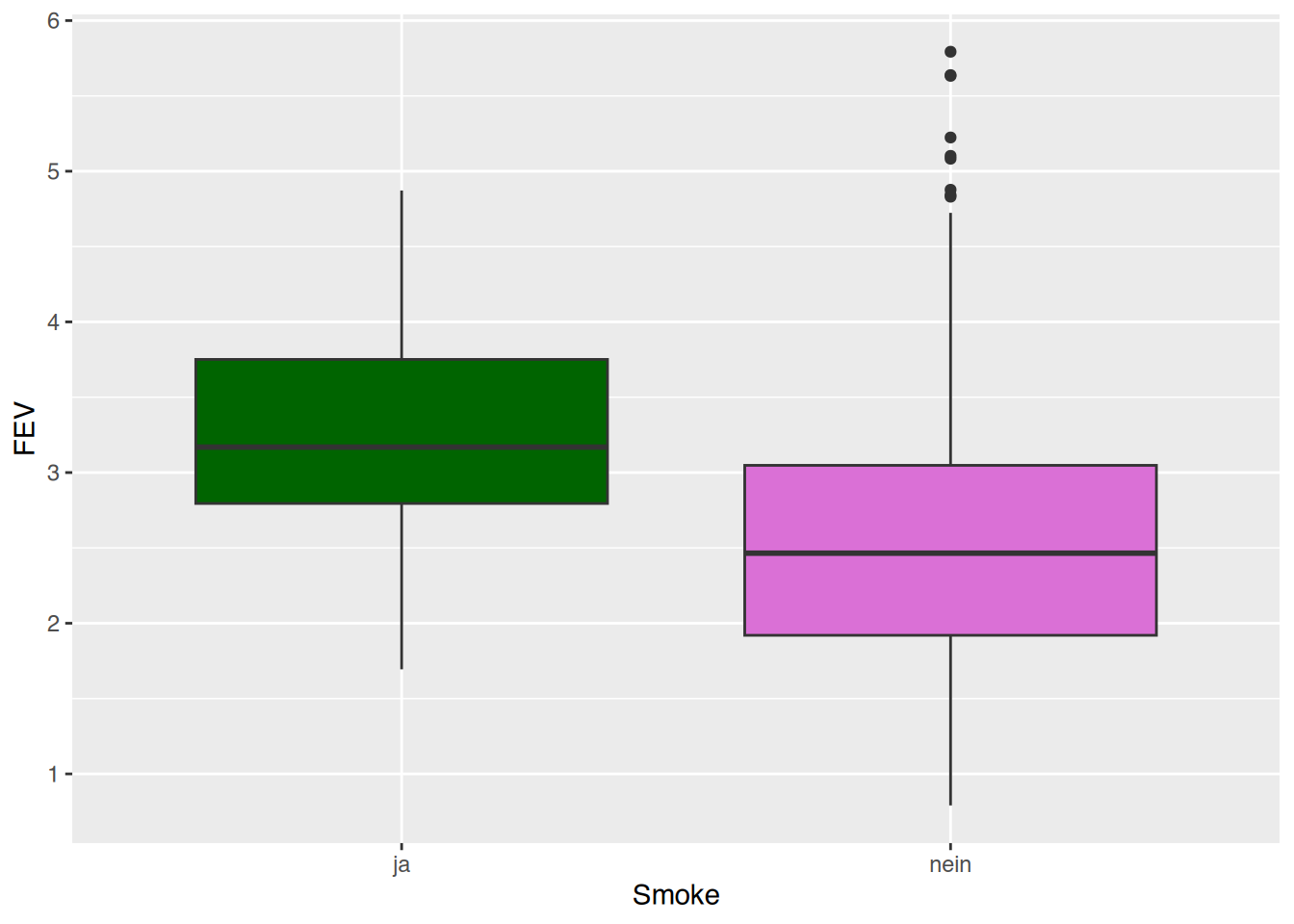
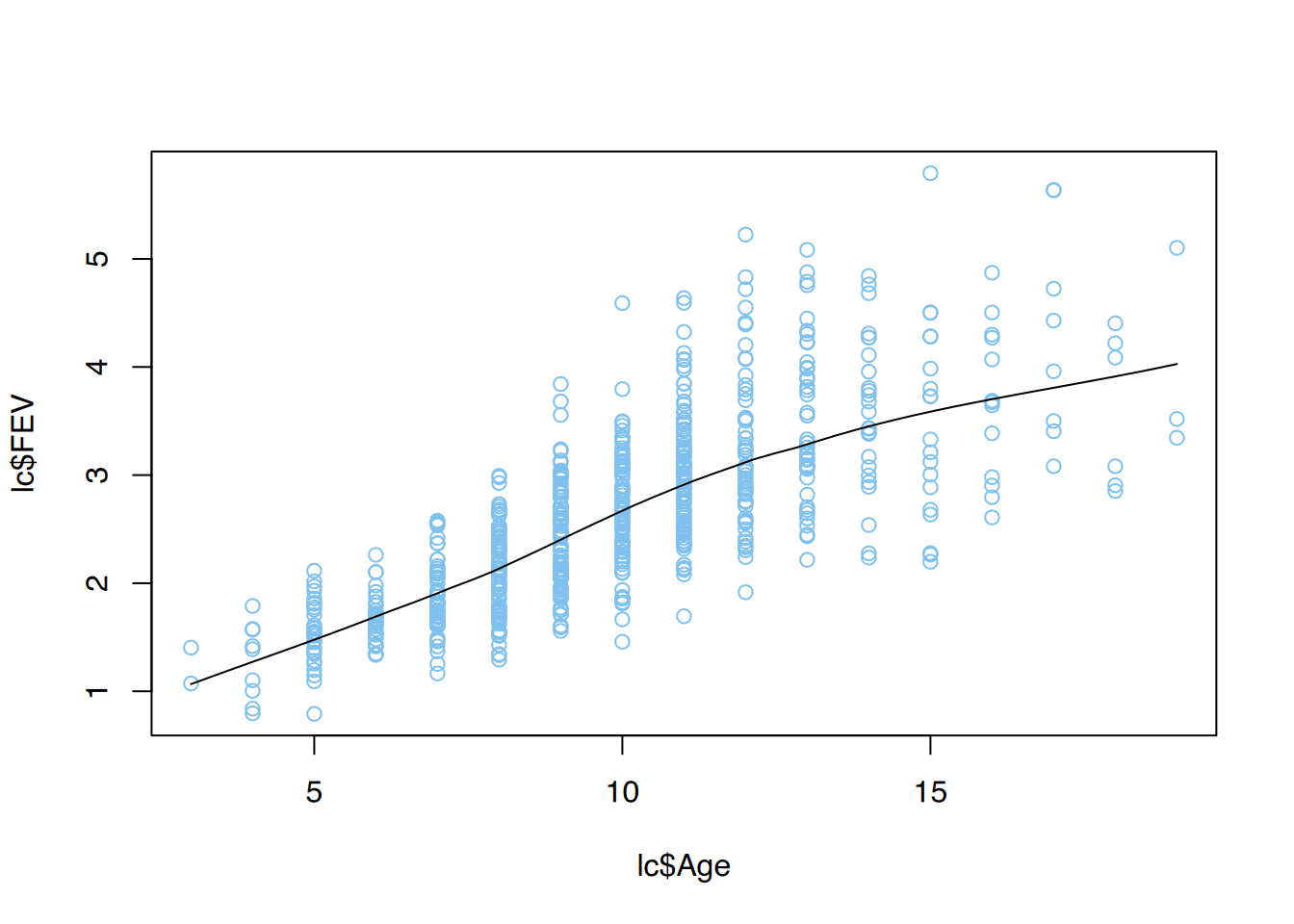
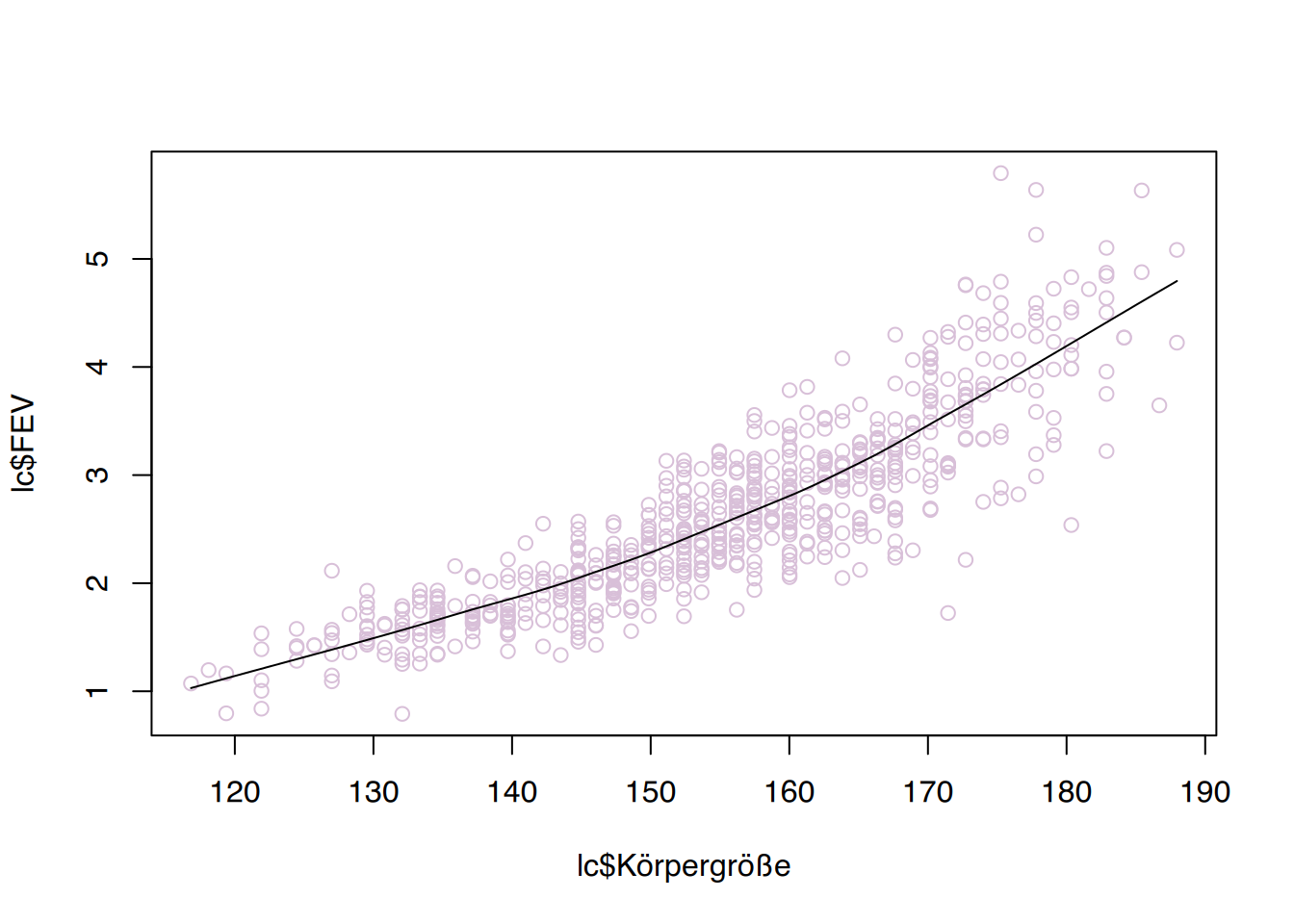
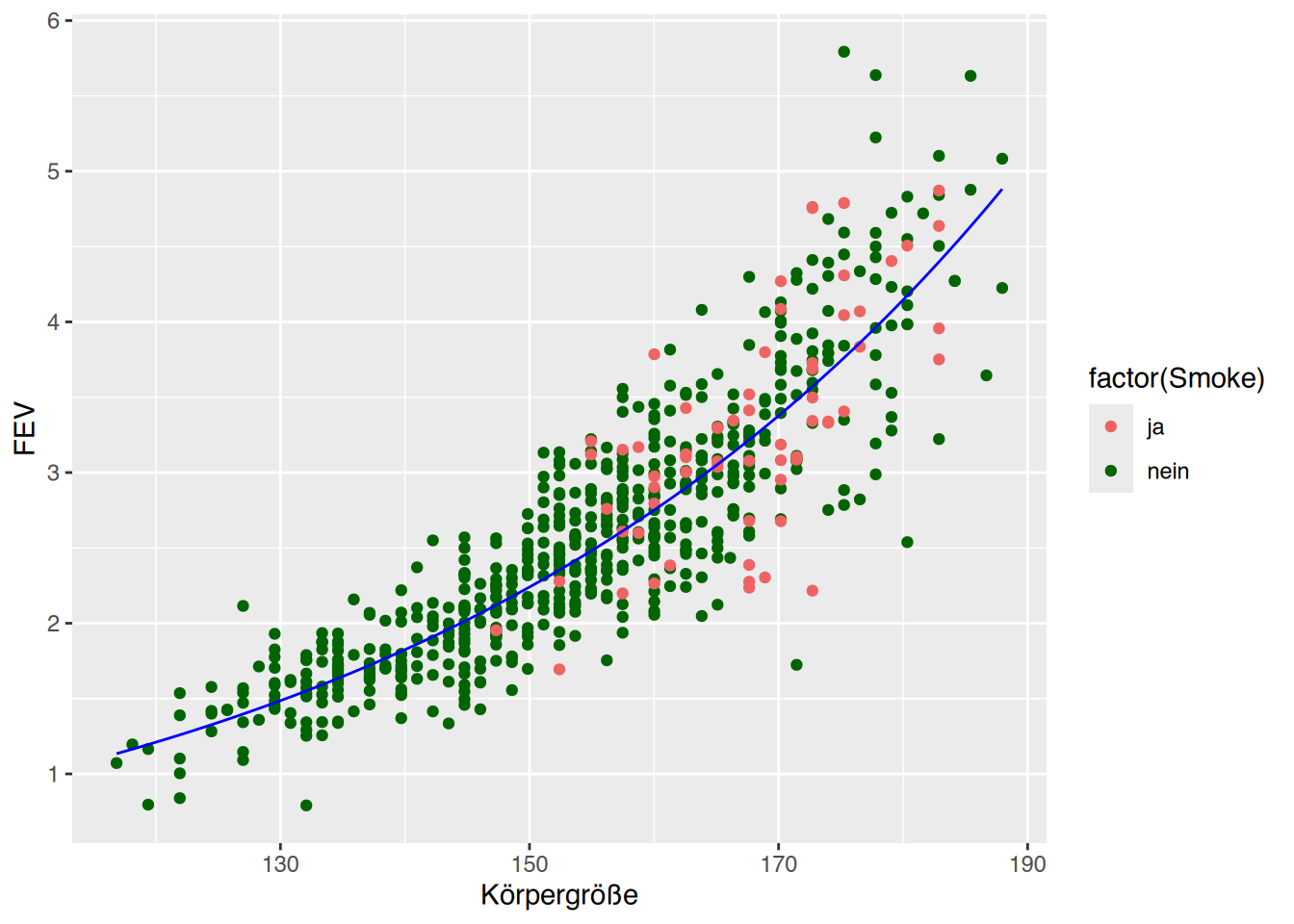
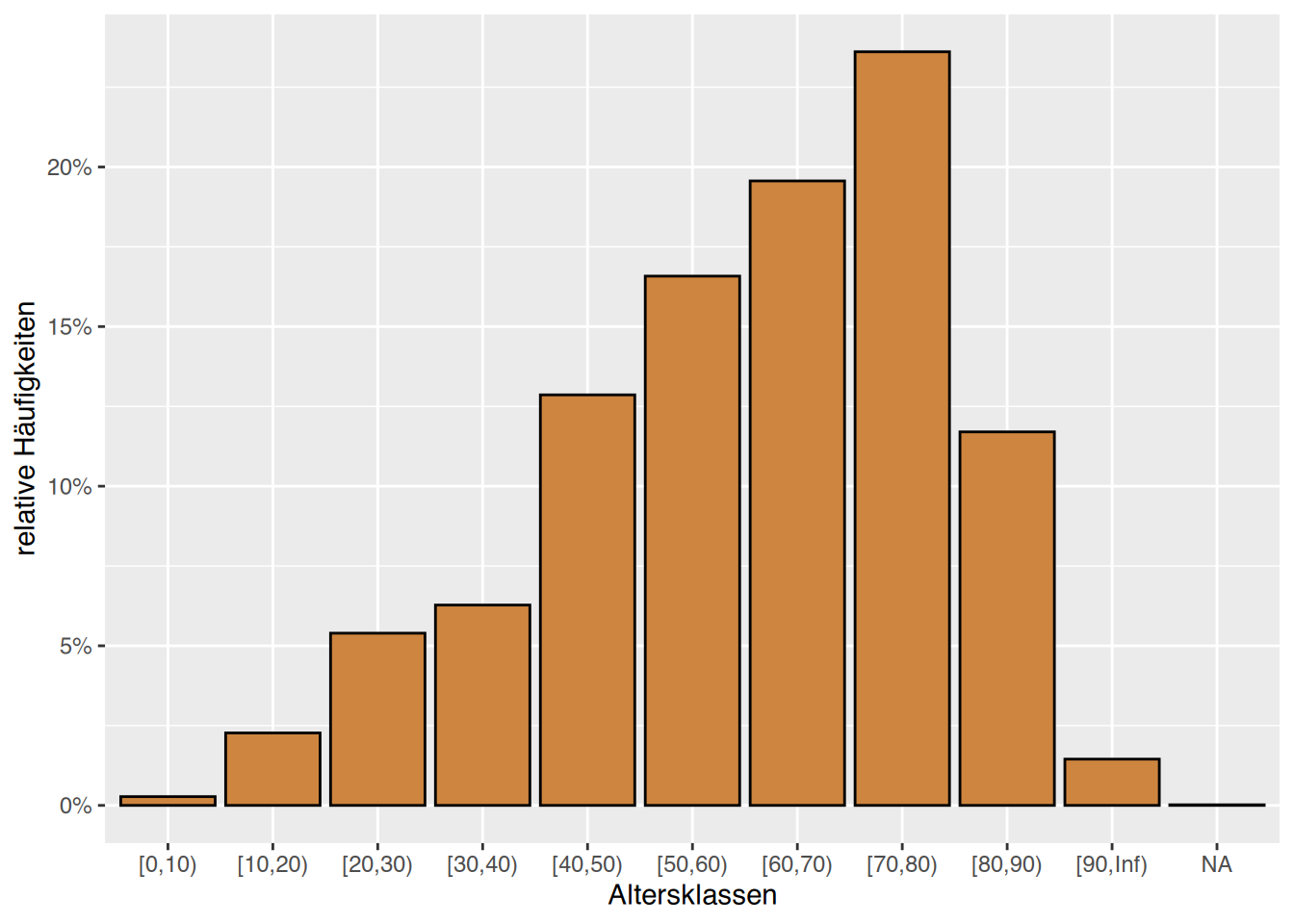
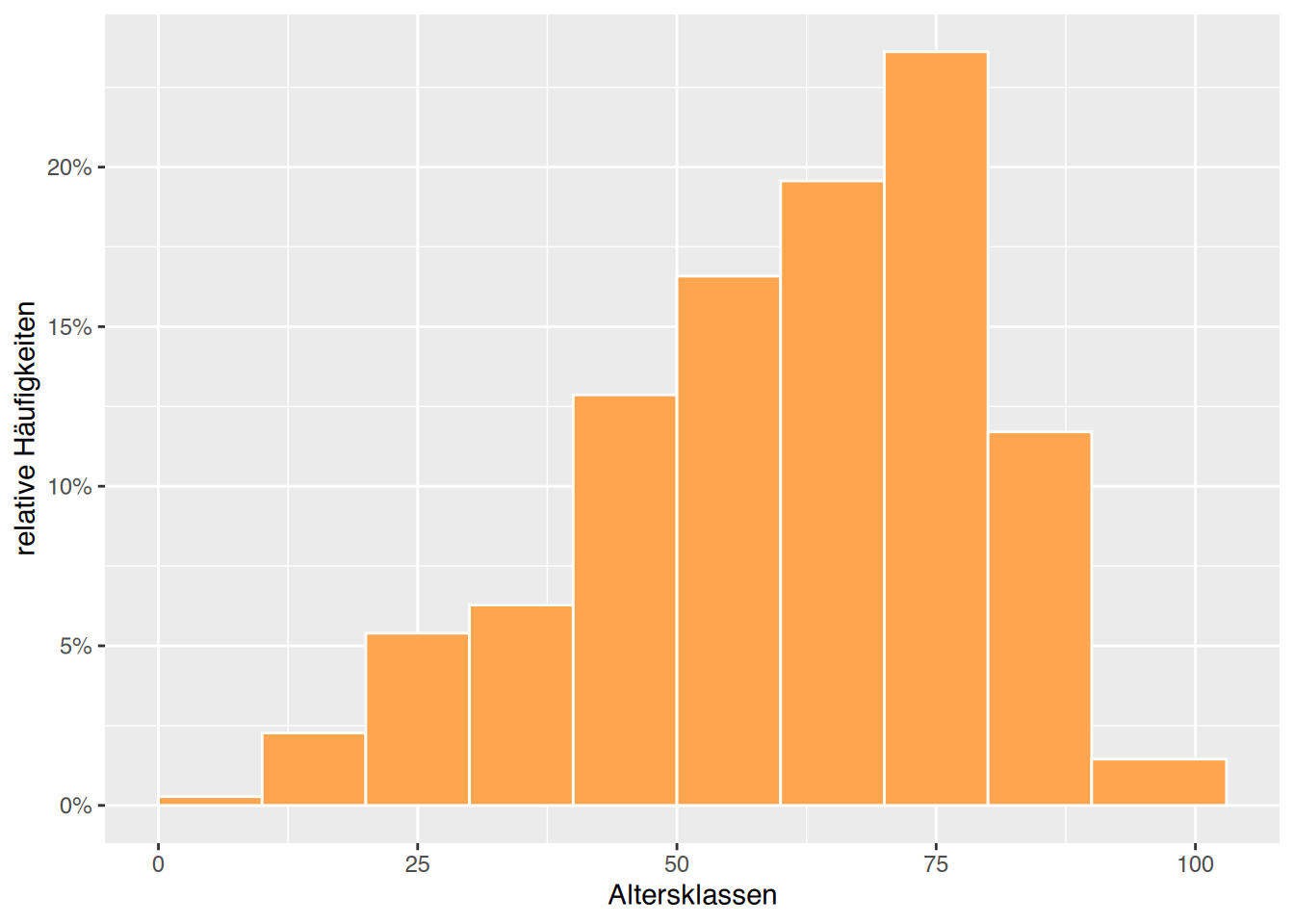
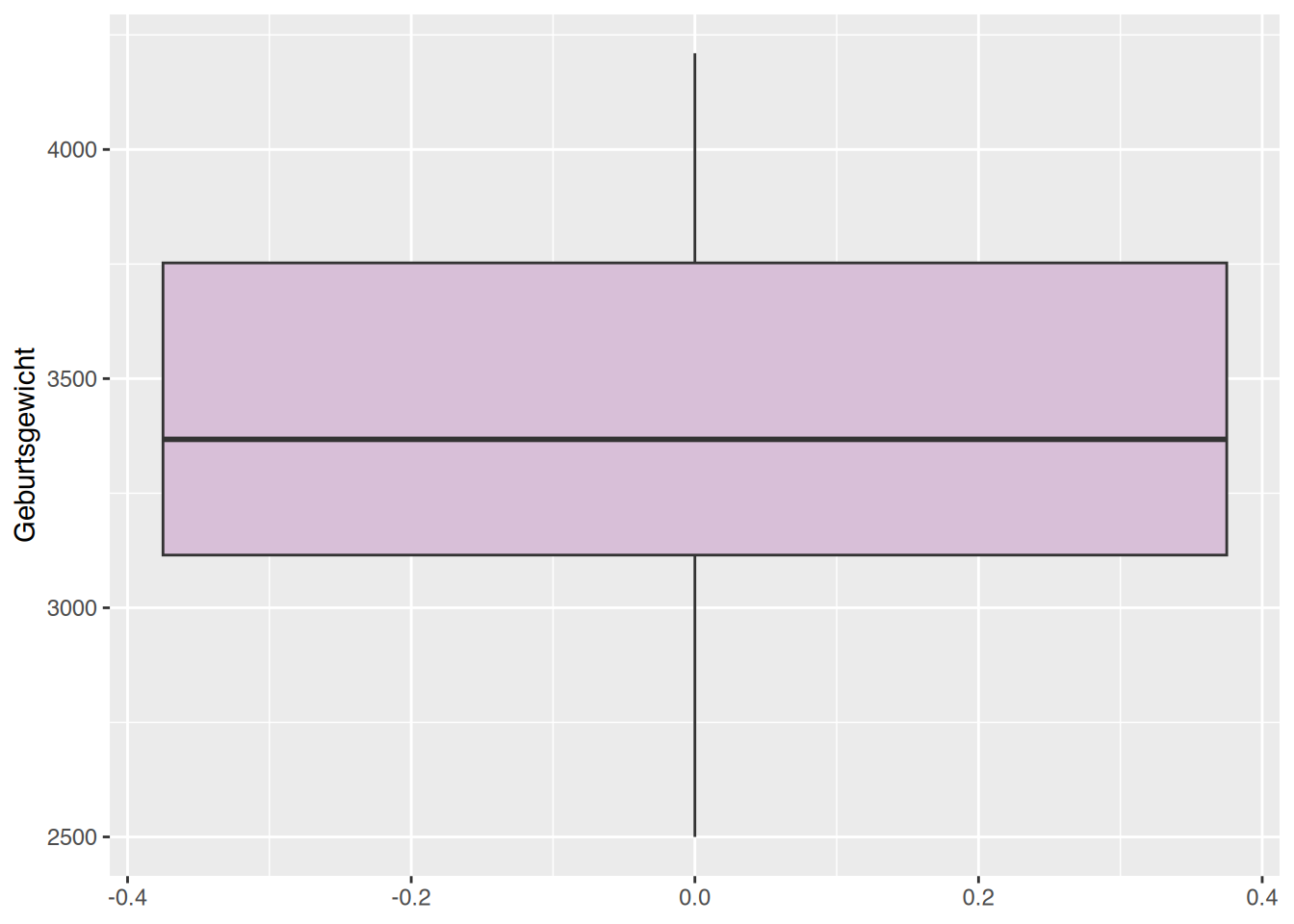
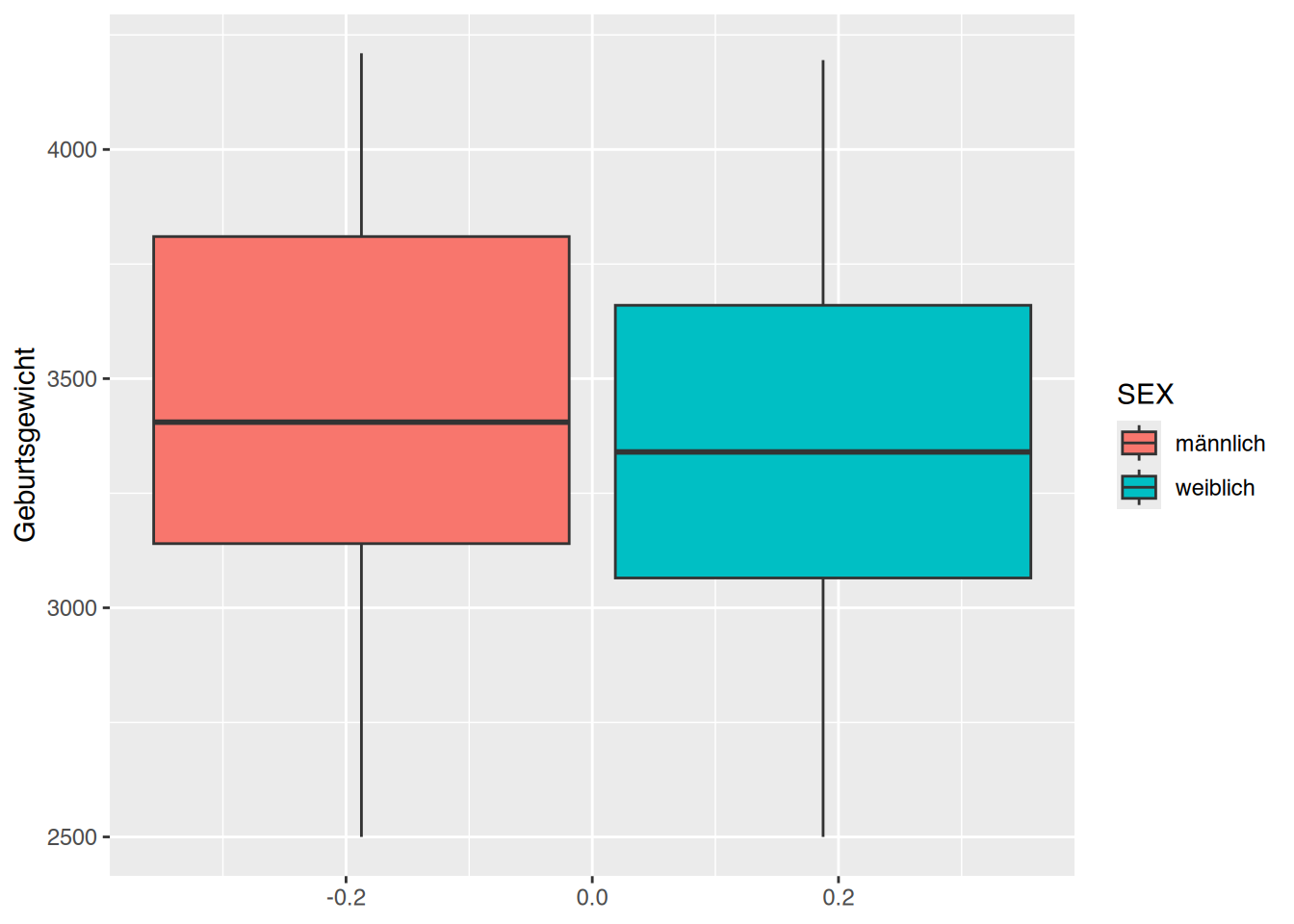
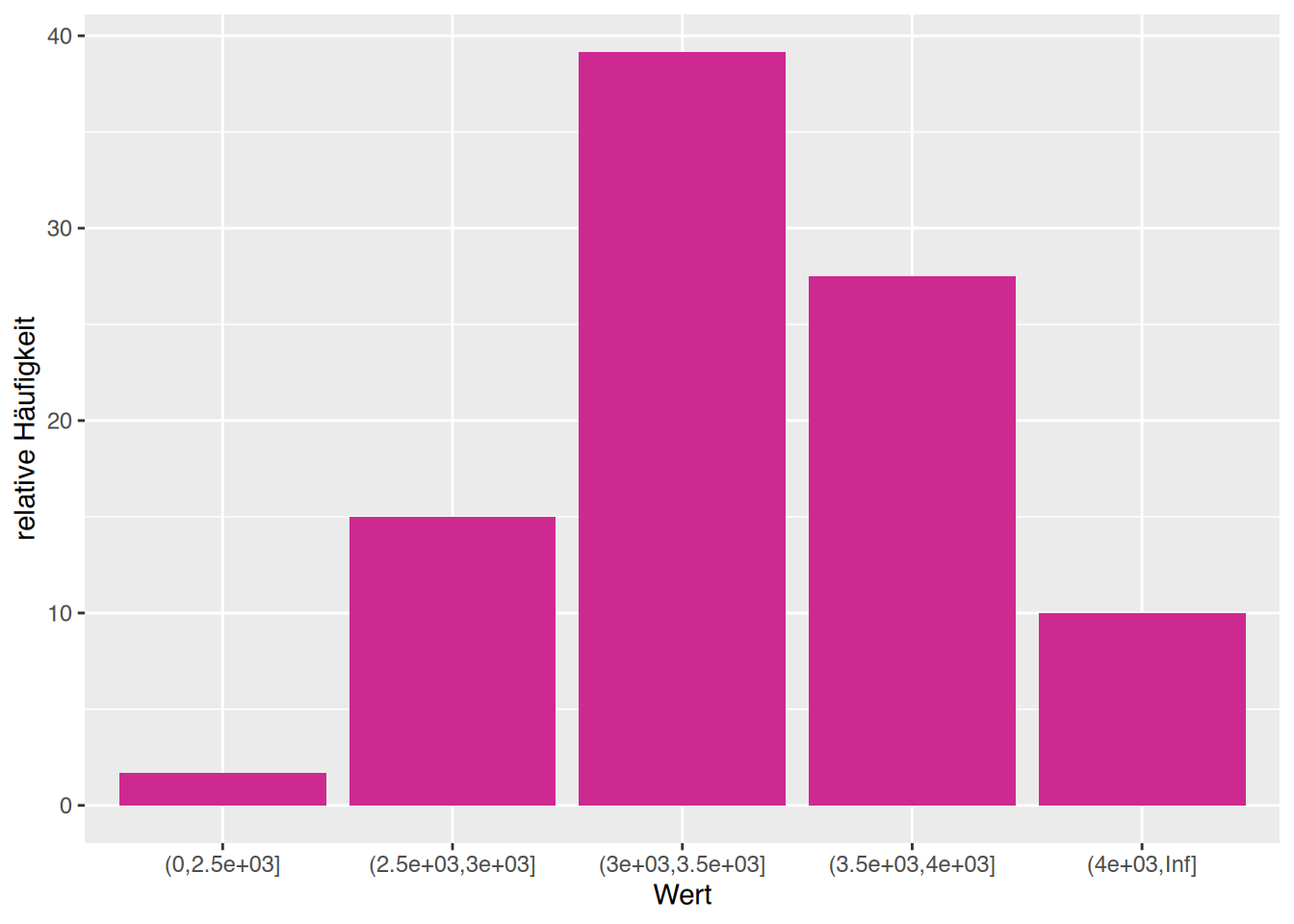
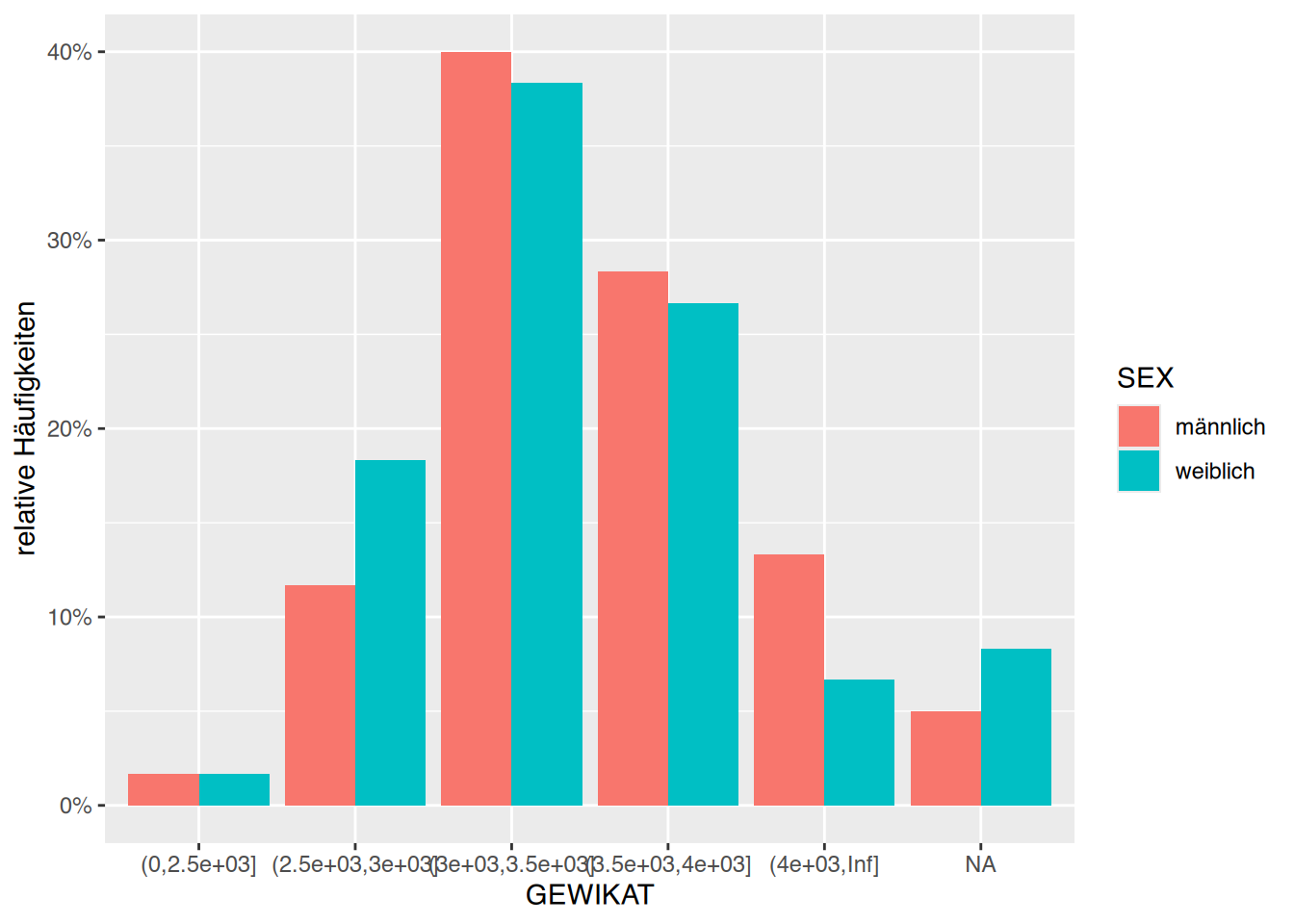
Name,Alter,Geschlecht,Gewicht,Größe,Cholesterol,BMI,Adipositas
Anna Tomie,18,W,85,179,182,26.528510346119,Übergewicht
Bud Zillus,32,M,115,173,232,38.4242707741655,Adipositas
Dieter Mietenplage,24,M,79,181,191,24.1140380330271,Normalgewicht
Hella Scheinwerfer,35,W,60,170,200,20.7612456747405,Normalgewicht
Inge Danken,46,W,57,158,148,22.8328793462586,Normalgewicht
Jason Zufall,68,M,96,174,249,31.7082837891399,Adipositas
Mitch Mackes,44,M,92,178,220,29.0367377856331,Übergewicht
Name,Alter,Geschlecht,Gewicht,Größe,Cholesterol,BMI,Adipositas
Bud Zillus,32,M,115,173,232,38.4242707741655,Adipositas
Dieter Mietenplage,24,M,79,181,191,24.1140380330271,Normalgewicht
Jason Zufall,68,M,96,174,249,31.7082837891399,Adipositas
Mitch Mackes,44,M,92,178,220,29.0367377856331,Übergewicht